벤지미다졸계 살균제 베노밀과 카벤다짐을 분해하는 Rhodococcus sp. 균주들의 분리 및 특성 분석

초록
본 연구는 잔류 시 유해한 영향을 줄 수 있는 벤지미다졸계 살균제 베노밀과 카벤다짐을 분해하는 세균을 농경지 토양으로부터 증균 배양을 통해 순수 분리하였다. rep-PCR 밴드 패턴 비교를 통해 3-2 균주와 6-2 균주를 선발하였으며 16S rRNA 염기 서열 분석 결과 두 균주는 모두 Rhodococcus 속으로 동정 되었다. 두 균주는 최소배지에서 베노밀 75 mg/L, 카벤다짐 235 mg/L를 60시간 내 미지의 대사산물을 거쳐 96% 이상 분해하였다. 3-2 균주를 이용한 토양 적용 실험 결과 3-2 균주는 30 mg/kg의 베노밀과 카벤다짐을 10일 이내 78-82% 분해하였으며, 균주를 접종하지 않은 경우 25~32%만이 제거되었다. 또한 두 균주에 의한 베노밀 및 카벤다짐 분해산물은 Penicillium roqueforti의 포자 발아를 저해하지 않아 두 균주가 베노밀과 카벤다짐을 완전히 무독화함을 확인하였다 이상의 결과로부터 본 연구에서 분리된 두 균주는 베노밀과 카벤다짐으로 오염된 토양의 정화에 적용할 수 있을 것으로 판단된다.
Abstract
To reduce the harmful effects of two benzimidazole fungicides, benomyl and carbendazim, we isolated two bacterial strains degrading the fungicides via enrichment culturing procedure. The rep-PCR band patterns and 16S rRNA gene sequence analysis identified the strains as Rhodococcus sp. 3-2 and Rhodococcus sp. 6-2. The two bacterial strains degraded 75 mg/L of benomyl and 235 mg/L of carbendazim in the minimum salt medium by more than 96% within 60 hr through an unknown metabolite. The soil application experiment demonstrated that strain 3-2 decreased 30 mg of benomyl and carbendazim/kg by 78-82% within 10 days compared with 25-32% in the uninoculated control soils. The fungal spore germination test was also conducted using Penicillium roqueforti to evaluate the toxicity of the degradation products of benomyl and carbendazim by the strains, which showed that the two bacterial strains completely detoxify the fungicides. These results shows that strains 3-2 and 6-2 can be utilized for the detoxification of soils contaminated with residual benomyl and carbendazim.
Keywords:
Bacteria, Benomyl, Biodegradation, Carbendazim키워드:
세균, 베노밀, 생분해, 카벤다짐서 론
농약의 개발로 농업 생산량은 크게 증가했으나 동시에 농약은 인체 및 생태계에 독성을 나타낼 수 있어 취급과 사용에 각별한 주의가 필요하다. 2019년 정부는 농약 허용물질목록 관리제도(Positive list system, PLS)의 시행을 통해 농약 사용량 감소와 농산물 안전성 확보에 큰 노력을 기울이고 있다. 하지만 농산물 뿐 아니라 토양 내 잔류농약에 대한 고려 역시 필요하다. 토양에 잔류하는 농약은 토양 내부의 유기물 등과 결합한 형태로 잔류하다가 특정 조건에서 식물과 동물에게 전이될 수 있다(Gevao et al., 2000). 2017년 국내 DDT 살충제 파동이 그 예로 DDT 잔류 토양을 닭이 섭취하여 계란으로 전이된 것으로 추정되고 있다.
베노밀(Benomyl)은 Methyl [1-[ (butylamino)carbonyl]-1H-benzimidazol-2-yl]carbamate (CAS No. 17804-35-2)의 형태를 가지는 벤지미다졸계(benzimidazole) 살균제로서 곰팡이에 의하여 발생하는 병 방제에 높은 방제 효과가 있어 과수, 채소, 원예 등 다양한 분야에 사용되고 있다(Yoon and Ko, 2019). 반면 베노밀은 미국 환경보호국(Environmental Protection Agency, US EPA)과 세계자연보전기금(World Wildlife Fund, WWF)에 의해 발암 가능성, 내분비계 장애(피부 자극, 두통, 설사, 성기능 장애)를 유발할 가능성이 있는 물질로 분류되어 있다(Park, 2009). 또한 청개구리(Hyla japonica)의 배아 발달, 지렁이(Lumbricus terrestis), 이리 응애(Amblyseius fallacis) 등 생태계를 구성하는 생물에게 전반적으로 미치는 유해한 영향은 오래 전부터 연구되어 왔다(Nakashima and Croft, 1974; Stringer and Wright, 1976; Ko, 2013). 베노밀은 환경에서 쉽게 가수분해되어 카벤다짐(Carbendazim)으로 변하며 이 물질이 농약의 유효물질로 알려져 있다(Yoon and Ko, 2019). 따라서 베노밀은 농산물이나 환경에서 검출되는 경우가 거의 없으나 카벤다짐의 경우 농산물과 하천에서 검출 빈도가 높은 농약으로 보고되고 있다(Kim et al., 2015; Kim et al., 2017; Hwang et al., 2019).
토양 내 농약을 제거하기 위해 다양한 방법들이 시도되었다. 물리화학적 방법은 다루기 쉽고 빠르다는 장점이 있으나 에너지 소모가 많고 비용이 많이 들며, 정화 후 환경에 대한 영향이 존재할 수 있다. 반면 미생물을 이용한 생물학적 정화 기술은 비교적 저비용으로 적용이 가능하고 환경과 인간에게 무해한 물질로 완전히 분해 가능하다는 이점이 있으나 분해 미생물에 대한 탐색과 특성을 파악하는 것이 중요하다(Morillo and Villaverde, 2017).
따라서 본 연구에서는 베노밀 및 베노밀의 분해산물로 발생하는 카벤다짐을 분해하는 미생물을 분리하여 특성을 구명하였다. 또한 베노밀 분해산물의 독성 확인을 위해 Penicillium roqueforti를 이용한 포자 발아 시험을 수행하였다.
재료 및 방법
시약 및 분석 방법
본 연구에 사용된 베노밀은 ㈜팜한농에서 원제(순도 96.8%)를 제공받았으며 카벤다짐은 시그마-알드리치 제품(Sigma Aldrich, USA)을 구매해 사용하였다. 시험에 사용된 acetone과 acetonitrile (ACN)은 HPLC급 시약을 사용하였다. 살균제의 농도는 Shimadzu HPLC (SCL-10Avp, Shimadzu, Japan)에 YMC-Triart C18 컬럼(150×4.6 mm, YMC, Japan)을 장착하여 PDA (photodiode array detector) 검출기를 이용해 분석하였다. 이동상은 ACN:water = 7:3, 유량은 0.7 mL/min으로 하여 베노밀은 286 nm,, 카벤다짐은 281 nm에서 흡광도를 측정하였다.
토양 시료는 10 g을 칭량하여 ACN 15mL을 첨가 후 30분 동안 교반하였으며 액체 시료는 ACN으로 2배 희석 후 각각 0.2 μm 실린지 필터를 이용해 여과한 후 HPLC 분석에 사용하였다.
증균 배양을 통한 베노밀 분해 미생물의 분리
베노밀 분해 미생물의 분리를 위해 전국 49개 농지의 토양을 채취해 동일한 양으로 섞은 다음 베노밀 원제의 농도가 50mg/kg이 되도록 혼합하였다. 토양 미생물의 활성 증진을 위해 클로버(Trifolium repens), 수크령(Pennisetum glaucum), 구절초(Siberian chrysanthemum)를 식재하여 온실에서 증균 배양(Soil enrichment) 하였다. 월 주기로 베노밀의 농도를 모니터링하여 농도가 검출 한계 미만으로 관찰되었을 때 토양 일부를 채취해 50 mg/L의 베노밀이 첨가 된 최소 배지(mm; minimum salt medium)에 옮겨 배양 후 농도 감소를 관찰하였다. 최소배지의 조성은 초순수에 Na2HPO4·12H2O 3.58 g, KH2PO4, 1.361 g, (NH4)2SO4 0.3 g, MgSO4·7H2O 0.05 g, CaCl2·H2O 0.0058 g, yeast extract 0.01 g과 trace minerals 10 mL, vitamin solution 1 mL을 첨가해 최종 부피가 1 L가 되도록 하였다(Ahn et al., 2016). 액체 배지에서 총 3회의 계대 배양 동안 농약 분해가 확인되면 R2A agar (Difco, USA) 배지에 도말, 28oC에서 배양해 콜로니 형태 별로 단일 콜로니를 순수 분리하였다.
베노밀 분해균의 유전 및 이화학적 특성 분석
증균 배양으로부터 분리된 균주의 유전학적 동일성을 확인하기 위해 rep-PCR 및 전기 영동을 수행하여 DNA 밴드 패턴을 비교하였다. PCR 반응은 GoTaq® DNA Polymerase (Promega, USA)를 사용하였고 프라이머로서(GTG)5를 사용하였다(Jarocki et al., 2016). 여기서 서로 다른 균주로 판단되면 16S rRNA 유전자 분석을 통해 동정 및 선발하여 실험에 사용하였다. 16S rRNA 유전자 PCR 방법 및 계통도 분석 방법은 이전 연구 방법(Ro et al., 2016)을 따랐다.
분리 균주의 최적 배양조건을 구명하기 위해 96-well plate에 R2A 배지를 첨가하고 염분(0-4.0% NaCl), pH (3.0-12.0), 온도 변화(8-30oC)에 따른 성장을 흡광도 분석(600 nm)을 통해 조사하였다(SpectraMax 340, Molecular Devices, USA). 가용 탄소원 검사를 위해 API 50 CH kit (bioMérieux, France)을 이용해 다양한 탄소원에서 성장 유무를 확인하였다.
배지와 토양에서 베노밀 및 카벤다짐 분해 시험
분리 균주의 살균제 분해 활성 시험을 위해 멸균한 15 mL glass tube에 최소 배지 3mL를 첨가해 베노밀, 카벤다짐의 농도를 각각 75 mg/L, 235 mg/L가 되도록 표준 원액을 첨가, 조제해 실험에 사용하였다. 분리된 균주를 0.85% NaCl 2 mL에 탁도 9.5 McFarland unit로 현탁한 후 100 μL을 취해 조제한 배지에 첨가한 다음(최종 108 cfu/mL) 28oC, 150 rpm에서 배양하여 시간에 따른 살균제 농도 감소를 분석하였다.
토양 내 분해 능력을 검증하기 위해 국립농업과학원 시험포장 내 토양 500 g을 취해 베노밀과 카벤다짐을 각각 50 mg/kg이 되도록 첨가한 후 분리 균주를 0.85% NaCl에 탁도 0.2 McFarland unit으로 현탁한 액 50mL를 혼합하여(최종 6.0×106 cfu/g) 28oC에서 보관하면서 시간에 따른 농도 감소를 관찰하였다. 토양의 물리화학성은 농촌진흥청 종합검정실 분석매뉴얼(RDA, 2017)을 적용하였으며, 시험 토양은 모래 42.6%, 실트 38.1%, 점토 19.1%의 양토로서 pH는 6.5, 수분함량은 12.34%, 유기물 함량은 19.39 g/kg, 총질소함량은 0.17%, 유효인산은 446.63 mg/kg로 나타났다.
베노밀 분해산물 독성 평가를 위한 곰팡이 포자 발아 시험
베노밀 분해 후 잔류 분해산물이 곰팡이 성장에 미치는 영향을 평가하기 위해 포자 발아 시험을 실시하였다. 먼저 1) 최소 배지 3mL에 PDB (potato dextrose broth) 0.072 g를 혼합해 대조구(mm+PDB)로 하고 2) mm+PDB에 베노밀 50 mg/L를 첨가한 배지(mm+PDB+benomyl)와 3) 베노밀 50 mg/L가 포함된 최소 배지에서 베노밀 분해균주 접종 후 9일 배양한 액에 PDB를 동량으로 첨가한 배지(균주+mm+benomyl+PDB)를 준비하였다. Penicillum roqueforti KACC 47196를 PDA (Potato dextrose agar) 배지에서 28oC에서 10일 이상 배양한 후 0.05%의 Tween 80이 포함된 멸균수에 혈구계를 이용하여 104 spores/mL이 되도록 포자를 현탁하였다. 위에서 조제한 배지 190 μL에 포자 현탁액 10 μL를 혼합한 후 96 well plate에 가하고 28oC에서 정치배양하면서 시간에 따른 포자의 발아 정도를 580 nm에서 흡광도를 측정하여 평가하였다. 또한 균주 처리 전후의 개선 효과 유의성을 통계적으로 검증하기 위해 엑셀의 데이터 분석기능을 활용하여 배양 동안의 흡광도 값(OD580)을 대상으로 대응표본 t 검정(paired t-test)을 수행하였다.
결과 및 고찰
베노밀 분해균의 선발 및 동정
베노밀 증균 배양으로부터 순수 분리된 균주 중 베노밀을 분해하는 5균주를 선발하였으며 균주의 동일성을 파악하기 위한 rep-PCR을 수행하였다(Fig. 1). 그 결과, 3-1-1, 3-2 균주가 동일하고 14-2, 15-1, 6-2 균주가 동일한 균주로 분석되어 3-2와 6-2 균주를 대상으로 후속 실험을 진행하였다. 두 균주의 16S rRNA 유전자 염기서열을 분석한 결과 Rhodococcus 속 표준 균주들과 유사성을 나타내었으며 특히 Rhodococcus degradans CCM 4446 및 Rhodococcus qingshengii JCM 15447과 99.9%의 유사도를 나타냈다(Fig. 2). Rhodococcus 속은 유류 오염 토양에서 분해 효소를 이용해 ring-dioxygenation, ring-monooxygenation, methyl monooxygenation 등에 의해 탄화수소계 오염물질을 분해할 수 있는 것으로 잘 알려져 있다(Kim et al., 2018). 선발된균주 중 3-2 균주는 국립농업과학원 미생물은행에 기탁번호 KACC 92274P로 기탁하였으며 3-2 균주와 6-2 균주의 16S rRNA 유전자 염기서열은 미국 국립생물정보센터(NCBI)에 각각 MN493844와 MN493845의 번호로 등록하였다.
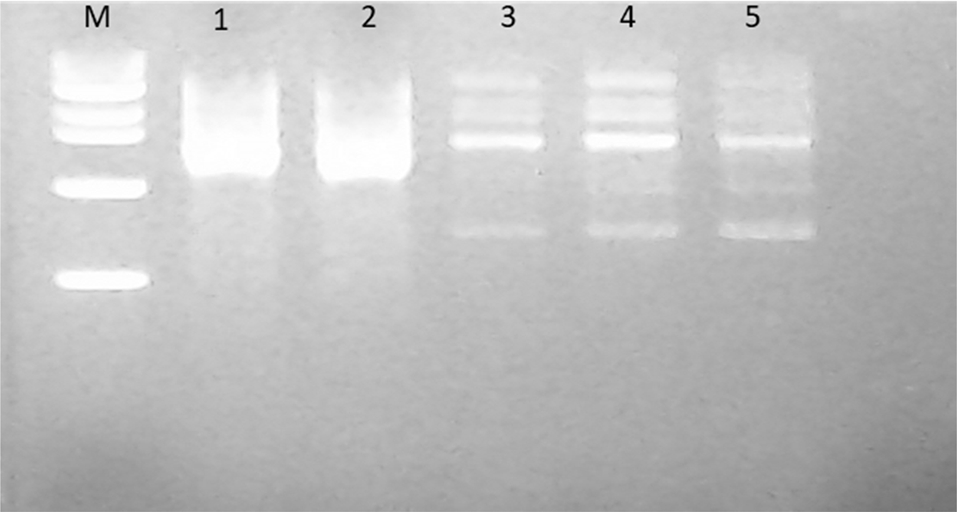
rep-PCR band patterns of isolated benomyl-degrading bacteria. * M, 100bp ladder; 1, strain 3-1-1; 2, strain 3-2; 3, strain 14-2; 4, strain 15-1; 5, strain 6-2.

Neighbor-joining tree based on the 16S rRNA gene sequences of the benomyl-degrading bacterial strains and related type strains.
3-2 균주와 6-2 균주의 최적 배양 조건을 구명하기 위해생리생화학적 특성을 분석하였다. 두 균주 모두 R2A 배지에서 활발한 성장을 보였으며, 3-2 균주는 0-7% NaCl, 8-40oC, 4.0-12.0의 pH에서 성장이 가능하며 0% NaCl, 28oC, pH 6.0에서 최적 성장을 나타내었다. 한편 6-2 균주는 0-6% NaCl, 8-40oC, 4.0-12의 pH에서 성장이 가능함을 확인하였으며 0.5% NaCl, 35oC, pH 8에서 최적 성장을 나타내었다. API 50 CH 분석 kit를 이용해 탄소원 49종에 대해 두 균주의 이용 가능성을 분석한 결과, 3-2, 6-2 균주 모두 D-trehalose를 제외하고 가용 탄소원의 성분이 동일함을 확인하였다(Table 1).
배지에서 베노밀 및 카벤다짐 분해
베노밀은 가수분해 및 미생물의 대사활동에 의해 카벤다짐으로 분해되는데 이 또한 잔류성과 독성이 있어 적절한 처리가 필요하다(Yarden et al., 1985; Lim and Miller, 1997). 따라서, 본 연구에서는 분리된 3-2, 6-2 균주를 75 mg/L의 베노밀과 235 mg/L의 카벤다짐을 포함한 최소배지에 접종하여 60시간에 걸쳐 배양하였다. 베노밀의 경우 60시간 후 대조구는 약 36.3% 정도 감소한 반면 3-2와 6-2 균주는 각각 97.7%, 99.8% 이상의 베노밀 감소를 보였으며, 카벤다짐의 경우 대조구, 3-2, 6-2는 각각 17.9%, 99.5%, 96.2%의 감소를 나타내었다(Fig. 3). 대부분의 감소는 12시간 내 이루어 졌는데 3-2 균주가 베노밀, 카벤다짐을 12시간 후 각각 97.5%, 73.2% 감소시켰으며 6-2의 경우 각각 94.6%, 81.1%의 감소가 확인되었다. 이는 Rhodococcus jialingiae djl-6-2를 이용해 60시간 배양을 통한 100mg/L의 카벤다짐을 94% 저감(Wang et al., 2010), Rhodococcus sp. D-1 을 이용해 200 mg/L의 카벤다짐을 2일 째 56.7% 저감(Bai et al., 2017) 등의 결과에 비해 우수한 분해 활성이다. 그 외 Rhodococcus erythropolis djl-11을 이용해 1,000mg/L의 카벤다짐의 제거를 평가한 사례도 있어 본 연구에서 분리된 균주가 더 높은 농도에서도 적용 가능성이 있을 것으로 판단된다(Zhang et al., 2013).
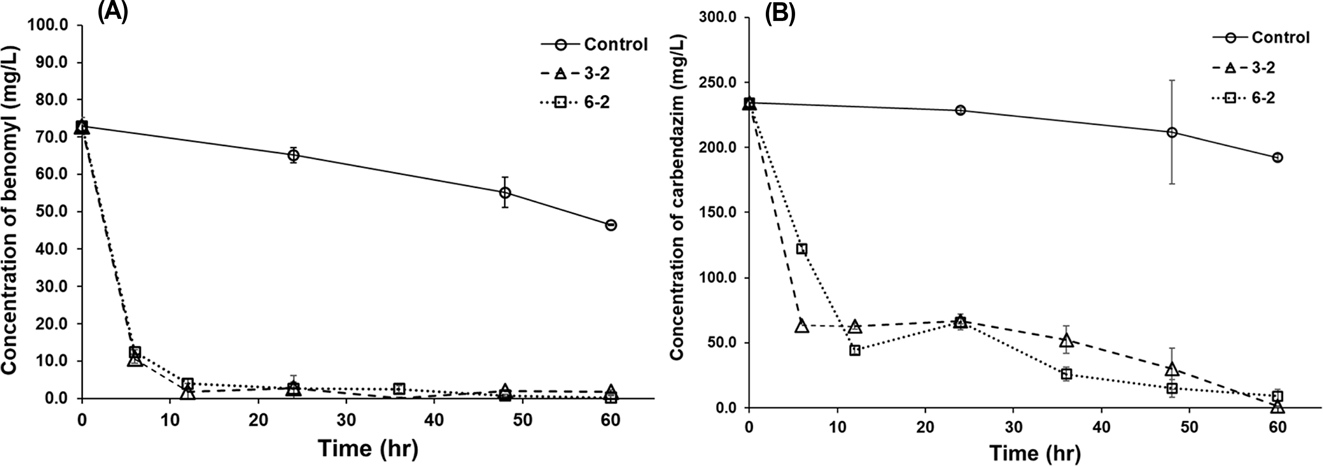
The concentrations of (A) benomyl and (B) carbendazim in minimal salt media during the growth of strains 3-2 and 6-2. “Control” indicates the same medium where the bacterial strains were not inoculated. Error bars indicate standard deviations of the mean (n=3).
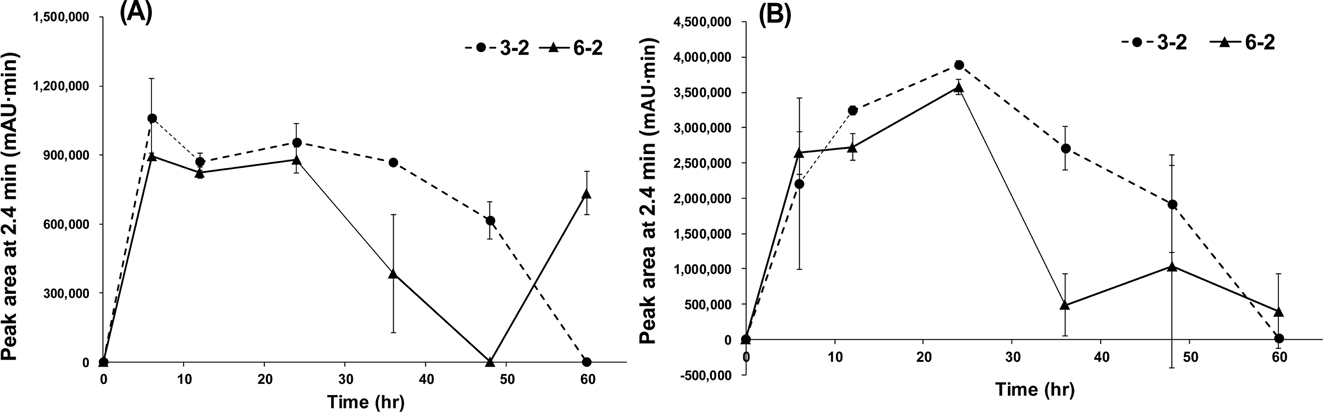
3-2와 6-2 균주에 의한 베노밀과 카벤다짐의 분해산물을 조사하기 위해 0시간, 24시간, 60시간에서의 HPLC 크로 마토그램을 분석한 결과, 24시간 배양 시 머무름 시간 2.4분에 중간 분해산물로 추정되는 물질이 검출되었다(Fig. 4). 미생물에 의한 베노밀 및 카벤다짐의 중간 분해산물은 대표적으로 2-aminobenzimidazole (2-AB), benzimidazole, 2-hydroxybenzimidazole (2-HB) 등이 보고되었다(Wang et al., 2010; Bai et al., 2017). 베노밀 및 카벤다짐의 분해 과정에서 발생한 분해산물의 거동을 분석하기 위해 머무름 시간 2.4분에 검출된 피크의 면적을 배양 시간에 따라 분석하였다(Fig. 5). 분석 결과 2.4분 피크는 베노밀과 카벤다짐 모두 처리 후 약 24시간까지 증가하다 이후 급속히 감소하였다. 이러한 경향은 이전 보고된 Rhodococcus sp. djl-6 균주에 의한 카벤다짐의 분해 과정에서 2-AB의 발생 경향과 유사함을 보이나(Zhang et al., 2013), 정확한 분석을 위해서는 향후 질량분석기를 통한 분자량 분석이 수행되어야 할 것으로 사료된다.
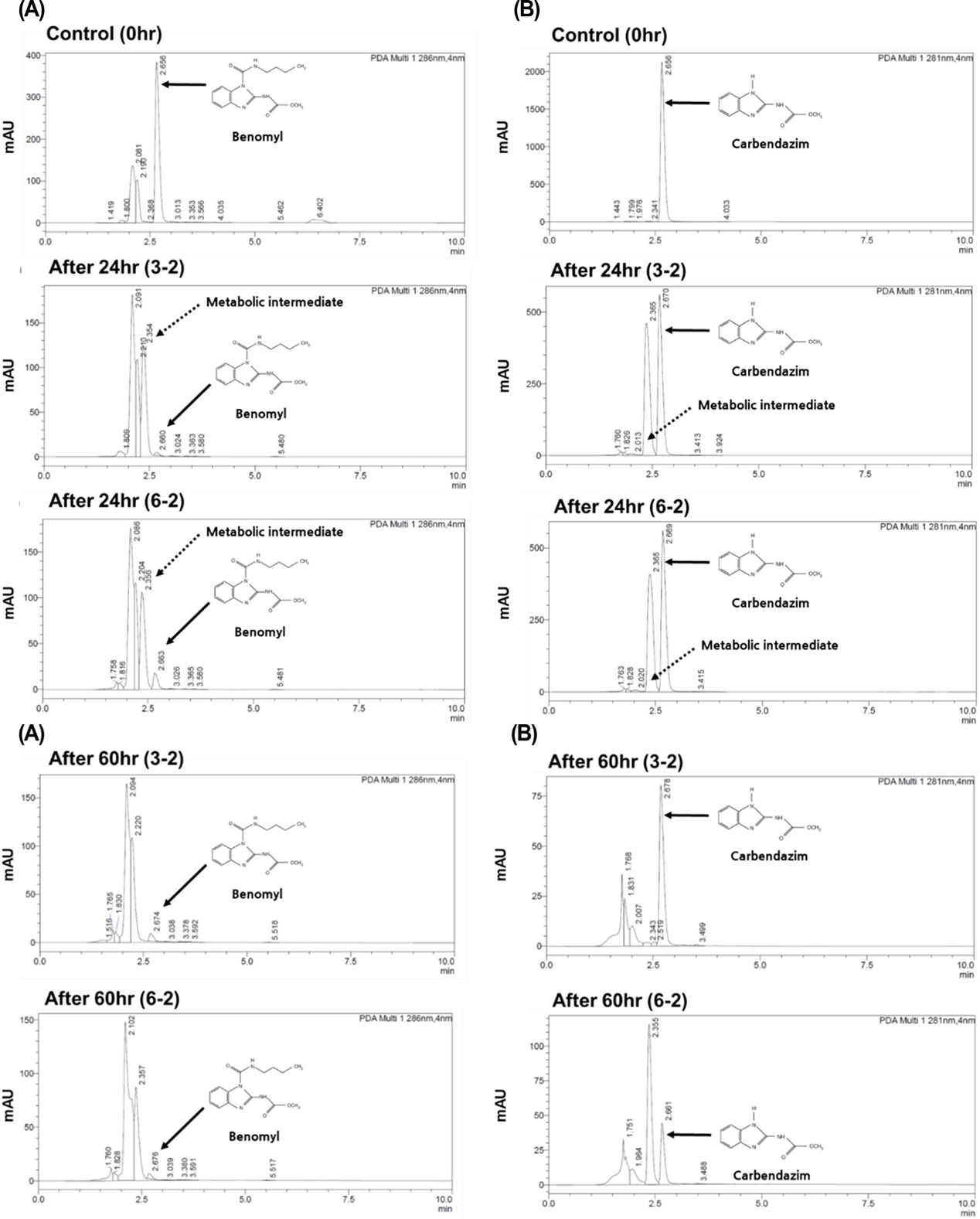
HPLC chromatograms of (A) benomyl and (B) carbendazim with their metabolic intermediate in minimal salt media inoculated with strains 3-2 and 6-2 initially containing 75 mg/L of benomyl and 253 mg/L of carbendazim, respectively, at 0-, 24-, and 60-hr incubations.
토양 잔류 베노밀 및 카벤다짐 분해
토양에서의 제거 효과를 분석하기 위해 약 30 mg/kg의 베노밀 및 카벤다짐이 포함된 토양에 3-2 균주를 접종 후 28oC에서 배양하였다. 3-2 균주는 바탕 시험으로부터 분석된 자연 감소에 비해 빠른 속도로 베노밀과 카벤다짐을 분해하였으며 10일 배양 후 베노밀과 카벤다짐의 농도는 대조구에서는 25-32% 감소한 반면, 3-2 균주를 접종한 경우 78-82% 감소하였다(Fig. 6). 하지만 10일 이후 베노밀 농도는 3-6 mg/kg로 유지되면서 뚜렷한 감소 경향을 나타내지 않았는데 이는 베노밀 및 카벤다짐이 이온결합, 수소결합, 리간드 결합, 공유결합 등에 의해 토양 입자에 흡착되어 생물 이용성이 감소되었기 때문으로 추정된다(Gevao et al., 2000).
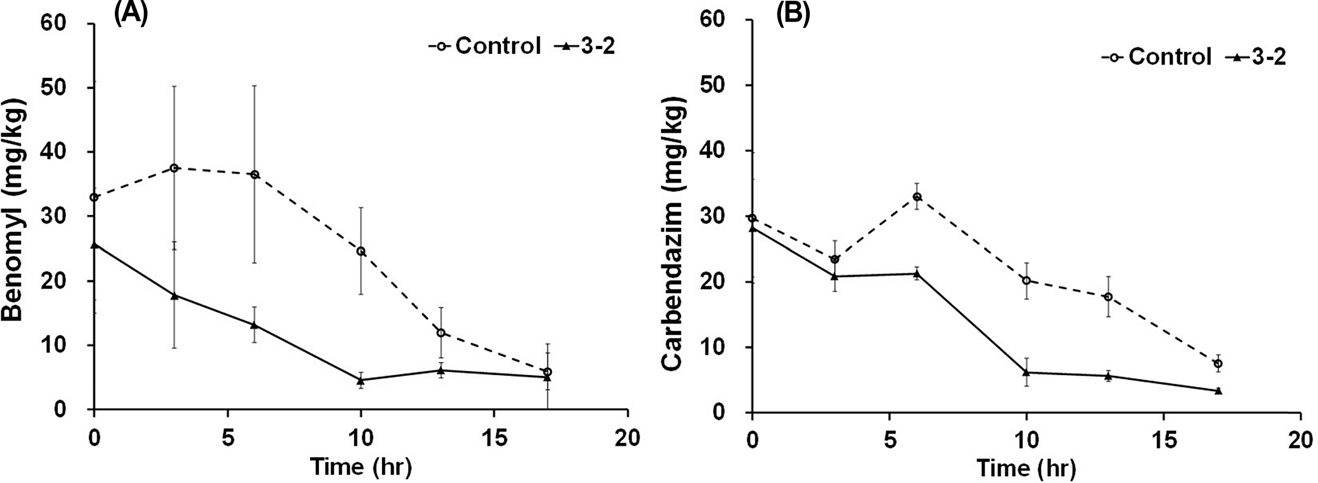
The behaviors of (A) benomyl and (B) carbendazim in soils inoculated with strain 3-2. “Control” indicates the benomly or carbendazim-containing soil where strain 3-2 was not inoculated. Error bars indicate standard deviations of the mean (n=3).
이전 연구에서 토양으로부터 분리된 Bacillus amyloliquefaciens의 카벤다짐 분해를 분석한 결과 토양 내 20, 50 mg/kg의 카벤다짐을 약 12일에 걸쳐 모두 제거하였고 이에 관여하는 효소 hy-1를 분리해 적용 하였을 때 더 빠르게, 높은 농도에서도 적용 가능함을 보고하였다(Li et al., 2019). 이는 본 연구에서 분리한 3-2, 6-2 균주 내 베노밀과 카벤다짐 분해 효소를 분리할 경우 더욱 향상된 효율을 가질 수 있음을 나타낸다. 또한, 두 종 이상의 조합(consortia) 또는 곰팡이 균주와의 공배양을 통한 생물증대(co-bioaugmentation)을 이용할 경우 베노밀과 카벤다짐의 분해 효율을 더욱 향상시킬 수 있을 것으로 판단된다(El-Hussein et al., 2014; Castro-Gutiérrez et al., 2019).
곰팡이를 이용한 배양액 잔류 독성 평가
3-2와 6-2 균주의 베노밀과 카벤다짐 분해산물의 독성을 평가하기 위해 곰팡이 포자 발아 시험을 수행하였다. 실험 결과 베노밀이 포함된 PDB배지에서는 Penicillium roqueforti의 성장이 완전히 억제된 반면 3-2와 6-2 균주를 이용해 9일에 걸쳐 베노밀을 분해한 배양액을 이용해 Penicillium roqueforti를 배양했을 때 성장은 베노밀이 포함되지 않은 대조구와 차이가 없는 것으로 나타났다(Fig. 7). 대응표본 t 검정(paired t-test) 결과 p-value가 두 균주 모두 0.05 이하로 분석됨에 따라 선발 균주에 의한 베노밀 분해산물은 Penicillium roqueforti에 독성이 없음을 확인하였다.
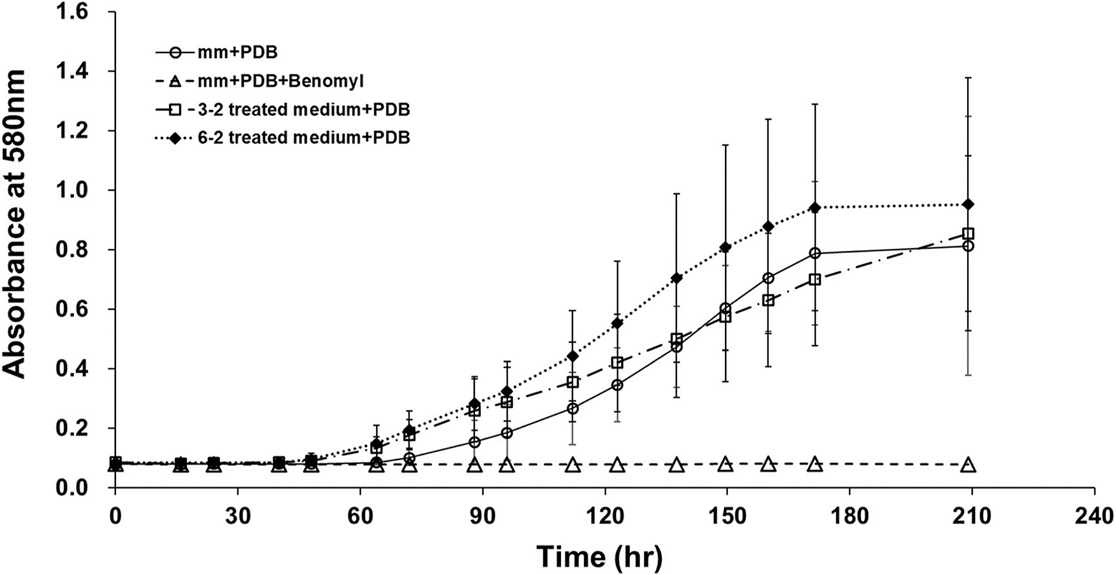
Effects of benomyl on the conidial germination of the Penicillium roqueforti according to treatment of the benomyl-degrading bacteria. mm, minimal salt medium; PDB, potato dextrose broth. Error bars indicate standard deviations of the mean (n=3).
이상의 실험결과를 바탕으로 증균 배양으로 분리된 Rhodococcus sp. 3-2와 6-2 균주는 베노밀을 완전히 무독화함을 확인하였고 1차 대사산물인 카벤다짐의 우수한 분해활성 또한 검증하였다. 따라서 향후 이 균주들은 베노밀 및 카벤다짐 오염 토양 및 농산물 정화에 활용할 수 있을 것으로 판단되며, 향후 대량 배양 조건 및 분해산물 구명 연구가 수행되어야 할 것으로 판단된다.
Acknowledgments
본 논문은 농촌진흥청 국립농업과학원 농업과학기반기술연구사업(과제번호: PJ01359601)에 의해 수행되었습니다.
이해상충관계
저자는 이해상충관계가 없음을 선언합니다.
Literate Cites
-
Ahn JH, Ro YM, Lee GH, Park IC, Kim WG, et al., 2016. Isolation and characterization of lactic acid bacteria for use as silage additives. Korean J. Microbiol. 52(4):432-442.
[https://doi.org/10.7845/kjm.2016.6060]

-
Bai N, Wang S, Abuduaini R, Zhang M, et al., 2017. Rhamnolipid-aided biodegradation of carbendazim by rhodococcus sp. D-1: Characteristics, products, and phytotoxicity. Sci. Total Environ. 590-591:343-351.
[https://doi.org/10.1016/j.scitotenv.2017.03.025]

-
Castro-Gutiérrez V, Masís-Mora M, Carazo-Rojas E, Mora-López M, Rodríguez-Rodríguez CE, 2019. Fungal and bacterial co-bioaugmentation of a pesticide-degrading biomixture: Pesticide removal and community structure variations during different treatments. Water Air Soil Pollut. 230(10):247.
[https://doi.org/10.1007/s11270-019-4282-y]

-
El-Hussein AA, Elsalahi RH, Osman AG, Sherif AM, El Siddig MA, 2014. Isolation and 16s rrna-based identification of benomyl-degrading bacteria. Biotechnol. J. Int.:670-683.
[https://doi.org/10.9734/BBJ/2014/10633]

-
Gevao B, Semple KT, Jones KC, 2000. Bound pesticide residues in soils: A review. Environ. Pollut. 108(1):3-14.
[https://doi.org/10.1016/S0269-7491(99)00197-9]

-
Hwang IS, Oh YJ, Kwon HY, Ro JH, Kim DB, 2019. Monitoring of pesticide residues concerned in stream water. Korean J. Environ. Agric. 38(3):173-184.
[https://doi.org/10.5338/KJEA.2019.38.3.21]

-
Jarocki P, Podleśny M, Komoń-Janczara E, Kucharska J, Glibowska A, 2016. Comparison of various molecular methods for rapid differentiation of intestinal bifidobacteria at the species, subspecies and strain level. BMC Microbiol. 16(1):159.
[https://doi.org/10.1186/s12866-016-0779-3]

-
Kim CS, Lee HD, Ihm YB, Son KA, 2017. Temporal patterns of pesticide residues in the keum, mangyung and dongjin rivers in 2002. Korean J. Environ. Agric. 36(4):230-240.
[https://doi.org/10.5338/KJEA.2017.36.4.38]

-
Kim D, Choi KY, Yoo M, Zylstra GJ, Kim EB, 2018. Biotechnological potential of rhodococcus biodegradative pathways. J. Microbiol. Biotechnol. 28(7):1037-1051.
[https://doi.org/10.4014/jmb.1712.12017]

-
Kim JY, Jung Y, Oh H, King ST, 2015. Monitoring and risk assessment of pesticide residues in commercial environment-friendly agricultural products distributed using lc-ms/ms in seoul metropolitan area. Korean J. Food Sci. Technol. 47(3):306-320.
[https://doi.org/10.9721/KJFST.2015.47.3.306]

- Ko SK, 2013. Effect of benomyl on the development of tree frog embryos. J. Ind. Technol 19(2):33-40.
-
Li Y, Chi M, Ge X, 2019. Identification of a novel hydrolase encoded by hy-1 from bacillus amyloliquefaciens for bioremediation of carbendazim contaminated soil and food. Int. J. Agric. Biol. Eng. 12(2):218-224.
[https://doi.org/10.25165/j.ijabe.20191202.4190]

-
Lim J, Miller MG, 1997. The role of the benomyl metabolite carbendazim in benomyl-induced testicular toxicity. Toxicol. Appl. Pharmacol. 142(2):401-410.
[https://doi.org/10.1006/taap.1996.8042]

-
Morillo E, Villaverde J, 2017. Advanced technologies for the remediation of pesticide-contaminated soils. Sci. Total Environ. 586:576-597.
[https://doi.org/10.1016/j.scitotenv.2017.02.020]

-
Nakashima MJ, Croft BA, 1974. Toxicity of benomyl to the life stages of amblyseius fallacis. J. Econ. Entomol. 67(5):675-677.
[https://doi.org/10.1093/jee/67.5.675]

-
Park JH, 2009. Photochemical degradation and toxicity reduction of methyl 1-[ (butylamino)carbonyl]-1h-benzimidazol-2-ylcarbamate in agricultural wastewater: Comparative study of photocatalysis and sonophotocatalysis. Desalination 249(2):480-485.
[https://doi.org/10.1016/j.desal.2009.01.018]

- RDA, 2017. Analysis manual for comprehensive assay. Rural Development Administration. Jeonju, Republic of Korea. 15-53.
-
Ro YM, Lee GH, Park IC, Kim WG, You JH, et al., 2016. Isolation and characterization of soil bacteria degrading a fungicide defenoconazole. Korean J. Pestic. Sci. 20(4):349-354.
[https://doi.org/10.7585/kjps.2016.20.4.349]

-
Stringer A, Wright MA, 1976. The toxicity of benomyl and some related 2-substituted benzimidazoles to the earthworm lumbricus terrestris. Pestic. Sci. 7(5):459-464.
[https://doi.org/10.1002/ps.2780070507]

-
Wang Z, Wang Y, Gong F, Zhang J, Hong Q, et al., 2010. Biodegradation of carbendazim by a novel actinobacterium rhodococcus jialingiae djl-6-2. Chemosphere 81(5):639-644.
[https://doi.org/10.1016/j.chemosphere.2010.08.040]

-
Yarden O, Katan J, Aharonson N, Ben-Yephet Y, 1985. Delayed and enhanced degradation of benomyl and carbendazim in disinfested and fungicide-treated soils. Phytopathol. 75(7):763-767.
[https://doi.org/10.1094/Phyto-75-763]

-
Yoon PS, Ko SK, 2019. Studies on toxicological evaluation of pesticides (fungicide, insecticide, herbicide) using tree frog embryos, hyla japonica. Korean J. of Environ. Ecol. 33(2):178-186.
[https://doi.org/10.13047/KJEE.2019.33.2.178]

-
Zhang X, Huang Y, Harvey PR, Li H, Ren Y, et al., 2013. Isolation and characterization of carbendazim-degrading rhodococcus erythropolis djl-11. PLoS One 8(10):e74810.
[https://doi.org/10.1371/journal.pone.0074810]