사과 내생 세균 자원에서 화상병원균에 대한 길항미생물 선발 및 분류
초록
Erwinia amylovora에 의해 발생하는 화상병은 다양한 장미과 식물에서 경제적 손실을 야기하고 있다. 국내에서는 2015년 처음으로 화상병이 발생해 전국 사과와 배 과수원으로 급속도로 확산되고 있다. 화상병 확산에 따라 발병 초기에는 병든 과수원 반경 내 숙주식물을 모두 제거하고, 화학물질 방제를 실시했지만 완전한 퇴치는 불가능하다. 따라서 본 연구에서는 생물적방제 후보 미생물을 선발하기 위하여, 사과 내성 세균 미생물 자원을 확보하고, E. amylovora에 대한 항세균력 균주 선발을 수행하였다. 그 결과 총 11개 화상병원균의 생육을 억제하는 사과 내생 미생물을 선발하였고, 분자생물학적 특성에 따라 Streptomyces sp. 또는 Paenibacillus sp.로 분류되었다. 선발된 내생미생물은 사과 잎에 접종하여 화상병에 대한 억제 능력을 검정하였으며 이는 사과 재배에서 화상병의 생물학적 방제제로 개발될 가능성이 높은 것으로 사료된다.
Abstract
Economic losses by Erwinia amylovora, a causal agent of fire blight disease, effected in various Rosaceae plants. In Korea, the fire blight disease was the first reported in 2015 and rapidly spread to apples and pears orchard nationwide. According to the spread of the fire blight disease, in the early stage of the outbreak, the whole plants within the radius of the diseased orchard were removed or chemical control was performed, but complete eradication of the disease is not possible. Therefore, in this study we conducted isolation and construction of apple endophytic bacterial library and screened the anti-E. amylovora strains. As result, we selected total 11 bacteria, which were classified as Streptomyces sp. and Paenibacillus sp. based on molecular characteristics. The selected bacteria have great potential to be developed as biological control agents in apple cultivation.
Keywords:
Antibacterial, Biological control, Erwinia amylovora, Paenibacillus, Streptomyces키워드:
항세균, 생물적방제, 화상병원균, 페니바실러스, 스트렙토마이세스서 론
과수 화상병(Fire blight disease)은 세균성 병원균인 Erwinia amylovora에 의해 발생하는 병해로 사과 뿐 아니라 배, 라즈베리 및 모과와 같은 장미과 식물의 조직에 검게 불에 탄듯한 병징을 나타낸다(Bubán and Orosz-Kovács, 2003; Born et al., 2017). 막대한 경제적 손실을 초래하는 것으로 알려진 화상병은 1780년 대 미국에서 처음 발견된 이후 50국 이상으로 퍼졌고 최근 한국, 중국 등 다양한 지역에서 발병 보고가 이루어지고 있다(Zhao et al., 2019). 국내 화상병은 2015년에 최초로 발생하였으며 이후 매년 지속적으로 발병하여 2019년 현재까지 원주, 충주, 음성, 이천, 파주, 용인, 연천 등으로 빠르게 확산되고 있다(Myung et al., 2016; Park et al., 2016).
E. amylovora은 바람, 비, 화분매개 곤충에 의해 단기간 빠르게 퍼지며 상당한 피해를 주는 매우 위험한 병원균으로 농림축산검역본부는 화상병원균을 식물방역상 금지병으로 지정하여 관리하며 화상병이 발생하고 있는 국가의 사과, 배 등 묘목 및 생과실 수입을 금지하고 있다(Miller and Schroth, 1972; Ordax et al., 2015; Lee et al., 2018; Kim et al., 2019). 또한, 발병 시점 초기에 즉시 발생 과수원 및 주변 반경 100 m 이내의 모든 기주 식물을 제거하여 매몰하는 공적 방제를 시행하고 있다(Lee et al., 2018).
화상병은 최초 발병 이후 발병 과수를 모두 제거하였으나 지속적으로 재 발생하여 병해 방제 및 병원균에 대한 근본적인 연구의 수행이 요구되고 있다. 이에 따라 동제(copper, 0.16 mM, 0.32 mM; Sholberg Yao et al., 2001)를 기반으로한 살균제 및 streptomycin (1.68 kg ha−1; Slack et al., 2020)과 oxytetracycline (1.68 kg ha−1; Slack e t al., 2020)의 항생제를 이용하여 화상병을 관리하고자 하였으나, 약제 저항성 병원균의 발생 빈도가 높았으며 화학적 방제 및 항생제 사용에 따른 독성 위험이 보고 되었다(Sholberg et al., 2001; McManus et al., 2002; Aćimović et al., 2015). 최근 유용미생물 Bacillus subtilis BD170, Lactobacillus plantarum PM411, TC54 and TC92, Pseudomonas fluorescens EPS62와 Pantoea agglomerans E325 등을 이용한 생물학적 방제가 대안으로 떠오르고 있다(Broggini et al., 2005; Cabrefiga et al., 2007; Pusey et al., 2011; Roselló et al., 2017).
이에 본 연구에서는 국내 사과 재배지에서 과수의 꽃과 잎에서 분리한 미생물을 이용하여 화상병에 대해 생물학적 방제 균주를 선발하고자 하였으며, 선발된 균주들의 기능적 분석을 통해 과수 화상병 맞춤형 방제 균주를 확보하고자 수행되었다.
재료 및 방법
사과 과수 선정 및 시료 채집
충청북도 음성군 사과농가 포장(36°56'46.3"N 127°43'13.0"E)에서 과수 간 수평 간격이 1.5m, 재식 간격이 3m인 사과 나무(cv. Fuji) 총 10 그루를 선정하여 각 시기에 해당하는 시료를 채집하였다. 시료 채집 시기로는 사과의 발달 생육 시기를 고려하여 2020년 3월 9일(1차), 2020년 4월 8일(2차), 2020년 4월 21일(3차), 2020년 5월 14일(4차), 2020년 6월 1일(5차), 2020년 7월 22일(6차) 총 6회 시료를 채집하였다. 1차에서는 잎 봉오리(n = 20), 2차에서는 꽃 봉오리(n = 20), 3차에서 꽃(n = 5 - 7), 4차에서 잎(n = 10 - 15)을 확보하였으며 5, 6차에서 잎과 과실(n = 3 - 5)을 각각 채집 후 시료를 밀봉하고 미생물 상 변화 방지를 위해 저온(4ºC)을 유지하여 실험실로 이동하였다.
사과 과수 조직 별 내생 미생물 분리
채집된 시료의 내생미생물을 분리하기 위해, 표면 살균을 진행하였다. 표면 살균을 진행하기 전, 10 g의 과실은 껍질을 벗겨 내생 미생물 분리 시료로 사용하였다. 그 외 조직인 잎 봉오리 2 g, 꽃 봉오리 2 g, 잎 5 g을 50-mL tube에 담은후, 1X Phosphate-buffered saline (10X PBS; 80 g NaCl, 2 g KCl, 14.4 g Na2HPO4, 2.4 g KH2PO4, pH 7.4 per L)로 50 mL까지 채워 초음파세척(35 kHz) 45초, vortexing 30초 총 3반복으로 시료 표면에 존재하는 미생물을 제거하였다(Yao et al., 2019). 표면 살균을 위해, 초음파 세척 후 각각의 시료를 70% ethanol 30초, 1% Sodium hypochlorite (NaOCl) 30초 침지하고, 멸균수로 2회 세척하여 무균대 내에서 약 1시간 상온 건조하였다. 건조된 시료는 5mL PBS buffer를 첨가하여 막자사발로 이후 갈아진 시료는 50% glycerol과 1:1로 혼합하여, -20oC에 보관하였다.
사과 내생 미생물 자원에서 E. amylovora 억제 미생물 선발
화상병원균에 대한 항세균성 미생물 선발은 총 3차에 걸쳐 진행되었다(Halder et al., 2017). 내생미생물 분리를 위해 TSA (Tryptic soya broth 30 g, agar 20 g per L) 배지 20 mL을 9 cm petri dish에서 건조 후 표면에 시료 100 μL 분주하여 도말 하였고, 28oC에서 2일간 정체 배양하였다. 대치 배양에 사용할 화상병원균 E. amylovora는 TSB (Tryptic soya broth 30 g per L) 배지에 2일 동안 배양 후, 멸균수를 이용해 OD600에서 0.2로 조절하여 0.2% carrageenan (Sigma-Aldrich, USA)와 1:1로 섞어 주었다. 그 후, 배양이 완료된 내생 미생물 plate에 E. amylovora (with 0.1 % carrageenan) 혼합액 2 mL 분주하여 무균대에서 1시간 동안 상온 건조하였다. 건조된 plate는 28oC 정체배양기에서 2일 동안 배양하였다. 항세균 능력을 나타내는 미생물 후보 군은 단일 균총을 확보하여 96-well plate (SPL, Republic of Korea)에 각 well 당 TSB 150 μL에 분주 후 접종하여 28oC에서 5일 간 진탕 배양하였다. 배양한 미생물들은 50% glycerol과 1:1로 섞어 보관 균주로 제작 후 -20oC에 보관하였다.
2차 대치배양은 9 cm petri dish에서 진행하였다. 96-well plate에 제작된 보관균주는 replicator를 이용하여 TSA 배지가 분주된 tray plate (127.9 × 85.5 × 16.2 mm; SPL, Republic of Korea)에 접종 후, 28oC 배양기에서 2일 동안 배양하였다. 배양 후 형성된 colony는 9 cm petri dish TSA를 4분할하여 균주를 배양하였다. 2일 후, 1차 대치배양과 동일한 방식으로 E. amylovora의 OD600를 0.2로 조정하였고 carrageenan과 1:1로 섞어 overlay 및 건조 후, 28oC 배양기에서 2일 배양하였다. 2차 대치배양 결과로 항균능력을 나타낸 균주들은 1차와 마찬가지로 96-well plate에 TSB 150 μL 배지에 보관균주를 제작하였다.
3차 대치배양은 2차 대치배양을 통해 확보된 균주들로 진행되었다. 3차 대치배양은 filter disk에 균주를 동일한 농도로 접종하여 실험을 수행하였다. 선발된 미생물은 멸균수에 섞어 동일한 OD600를 0.3으로 조절해 9 cm petri dish MGY (D-mannitol 10 g, L-glutamic acid 2 g, KH2PO4 0.5 g, NaCl 0.2 g, MgSO4∙7H2O 0.2 g, Yeast extract 1 g, agar 15 g per L, pH 7.0) 배지의 filter disk에 10 μL 접종하였다. 기중균사를 형성하는 Streptomyces sp.로 추정되는 균들은 MS (Mannitol Soya; Mannitol 20 g, Soya 20 g, Agar 20 g per 1 L) 배지에 배양해 생성된 포자를 멸균한 솜을 이용해 포자만을 모아 멸균수를 이용해 OD600 값을 조정하였다(Kim and Kwak, 2021). 접종일로부터 1~7일 경과 후, E. amylovora OD600 0.3과 0.2% carrageenan을 1:1로 혼합하여 overlay한후 28oC 인큐베이터에서 2일 배양하였다. 2일 후 항균능력을 나타내는 미생물들은 보관균주를 제작하였다. 모든 화상 병원균 대치배양은 국립농업과학원에서 수행하였다.
선발 항세균 미생물의 분자생물학적 동정 및 계통학적 분석
항세균성 능력을 나타내는 것으로 확인된 11개 선발 미생물들은 분자생물학적 방법으로 동정을 진행하였다. TSA 배지에 배양된 선발 미생물들은 DNA을 추출하기 위해 CTAB (Cetyltrimethylammonium bromide; 2.0% (w/v) CTAB (cetyltrimethylammonium bromide; 100 mM Tris-HCl, pH 8.0, 20 mM EDTA pH 8.0, 1.4 M NaCl) 방법을 사용하였다(Graham et al., 2003). 추출한 DNA는 NanoDropTM 2000c (Thermo Fisher, Waltham, MA, USA)를 이용해 정량하였다. 그 후 27F (5'- AGAGTTTGATCMTGGCTCAG-3') 1492R (5'-TACGGY TACCTTGTTACGACTT-3') 프라이머를 이용하여 16S rRNA 영역을 증폭하였다. PCR은 Genomic DNA 1 μL (100 ng/μL), Primer (10 pmol) 1 μL, dNTP (2 mM) 4 μL, 2 × PCR buffer 20 μL, KOD FX Neo 0.3 μL (0.1 U/μL, Toyobo, Japan)로 최종 40 μL으로 진행하였다. PCR 과정은 98oC 5분간 pre-denaturation을 수행한 후, 98oC denaturation 30초, 56oC annealing 30초, 72oC elongation 1분 30초 과정은 30회 반복하였다. 그 후, 72oC에서 final-elongation 10분간 진행하였다. PCR 산물들은 0.8% agarose gel에 전기영동 후, gel elution을 진행하였다. 전기 영동을 통해 얻어진 약 1.5 kb band는 gel elution을 위해 소독한 메스로 잘라주었고 ExpinTM Gel SV kit (GeneAll, Republic of Korea)를 이용하여 정제하였다. 절단된 agarose 절편은 microcentrifuge tube에 넣어주었고 300 μL GB buffer를 첨가해 50oC에서 10분간 반응하였다. 열을 가한 후, 식혀 column tube에 옮겨 원심분리를 해주었다. 원심분리한 column은 80% ethanol을 이용해 정제하였으며 건조시켜 EB buffer로 DNA를 column에서 분리하였다.
정제된 PCR 산물은 Cosmogenetech (Seoul, Korea)에서 Sanger sequencing method를 이용하여 염기서열 분석을 하였다. 분리된 미생물의 PCR 산물은 T-vector 클로닝을 위해 A-tailing과 정제를 진행하였다. A-tailing은 dATP (BioLabs, USA) 3 μL, 10X Reaction buffer (Bioneer, Republic of Korea) 3 μL, Taq DNA p olymerase ( 500U; Bioneer, Korea) 1 μL를 섞어 70oC에서 1시간 동안 반응시켜주었고 ExpinTM Gel SV kit을 사용하여 재정제하였다. A-tailing 산물은 프로토콜을 참고하여 동일하게 정제하였고 pGEM®-T Easy Vector Systems (Promega, USA)을 사용하여 클로닝을 진행하였다. 클로닝에서 pGEM®-T Easy Vector (50 ng) 1 μL, T4 DNA Ligase 1 μL, T4 DNA Ligase 7.5 μL 정제된 DNA 10 μL를 넣어 16oC에서 20시간 반응시켜주었다. 반응시킨 산물은 열충격을 통해 30분 동안 얼음에 넣은 E. coli strain DH5α와 형질전환을 하였다. 형질전환은 42oC에 1분 30초, 얼음에 2분동안 넣은 후, LB (Luria-Bertani 30 g per L) broth 900 μL를 넣어 1시간 동안 37oC 진탕배양기에서 배양시켜 진행하였다. 그 후, 원심분리기(Beckman Coulter, USA) 14,000 rpm에서 1분간 원심분리기를 작동시켰다. 결과로 분리된 850 μL 상층액은 제거하고 남은 상층액과 섞어 20 μL, 90 μL LB + Ampicillin (final concentration: 100 μg/mL) + X-gal (final concentration: 40 μg/mL; tech & innovation, Korea)을 넣은 배지에 도말하였다. 고체 배지에서 형성된 흰색 단일 균총을 5mL LB broth에 접종하여 16시간 동안 37oC에서 배양하였고 DokDo-Prep Plasmid Mini-Prep Kit (ELPIS, Korea)를 통해 플라스미드를 추출하였다. 배양한 세균은 원심분리기를 이용해 회수하고, 250 μL Sol I을 섞어준 후, 250 μL Sol II를 3분간 처리해 주었다. 3분 뒤 즉시 Sol III 350 μL를 첨가하여 13,000 rpm에서 원심분리를 작동하여 정제하였다. 추출한 플라스미드는 제한 효소 처리를 통해 band를 확인하였다. 제한 효소 ApaI (NEB, USA) 1 μL, NdeI (NEB, USA) 1 μL, 1X CutSmartTM Buffer (NEB, USA) 2 μL, DNA (1000 ng) 2~3 μL, ddH2O 13~14 μL를 섞어 37oC에서 4시간 동안 반응시켜, 0.8% agarose gel에서 3 kb (vector), 1.5 kb (16S rRNA)두 개의 band의 여부를 확인한 후, Cosmogenetech에 시료를 보내 염기서열 분석을 진행하였다. 염기서열이 진행된 각 샘플은 NCBI Nucleotide BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)에서 동정하였고 MEGA 10 program의 maximum likelihood 방법을 사용하여 분자계통도를 그렸다.
선발 미생물 효소 활성 검증
선발된 미생물은 cellulase, chitinase, proteinase, siderophore 활성을 확인하기 위해 다음과 같은 과정으로 실험을 진행하였다. Cellulase활성 검증은 배지(yeast extract 1 g, CMC 1 g, KH2PO4 4 g, MgSO4 1 g, MnSO4 0.05 g, FeSO4 0.05 g, CaCl2 2 g, NH4Cl 2 g, Agar 18 g per L; pH 7.0 ~ 7.4)에 선발된 미생물 OD600 0.3 10 μL를 8 mm filter disk (Advantec, Taiwan)에 접종하여 28oC에서 7일 동안 배양하였다(Carder, 1986). 배양한 플레이트는 0.1% Congo Red를 1 mL 분주하여 20분 건조 후 1 M NaCl로 3~5회 세척하고 투명한 영역 길이를 측정하였다. Chitin 분해 실험은 Chitinase 배지(Chitin 5 mL, Yeast 5 g, KH2PO4 0.7 g, K2HPO4 0.05 g, MgSO4 0.3 g, FeSO4 0.1 g, NaCl 0.1 g, Agar 20 g per L; pH 6.5 ~ 7.0)에 cellulase 실험과 동일하게 진행한 후, 0.2% Congo Red를 분주하여 플레이트상 투명한 영역 길이를 측정하였다(Hwang et al., 2016). Proteinase 활성 실험에서는 proteinase 배지(skim milk 10 g, Agar 20 g, per L)에 동일한 농도로 접종하여 5일 후, 플레이트상 투명한 영역 길이를 쟀다(Berg et al., 2005). Siderophore 활성 실험은siderophore 배지(MM9 salt solution 100 mL, ddH2O 750 mL, PIPES 32.24 g, Agar 15 g; pH 6.5 멸균 후, 50oC에서 casamino acid solution 30 mL, 20% glucose solution 10 mL, blue dye solution 100 mL)에 균주 OD600 0.3 10 μL 접종하여 siderophore 합성 실험을 진행하였다(Louden et al., 2011).
선발 미생물의 화상병 발병 억제력 검증 및 재분리
선발된 미생물 억제 능력을 검증하기위해 사과이용연구소(35°43'10.5"N 127°54'6.9"E)에서 사과 어린잎(cv. 홍로)을 채집하였다. 채집한 잎은 70% ethanol 10초, 0.5% NaOCl 10초 동안 침지 하였고 멸균수로 2회 세척하여 무균대에서 30분 동안 건조시켰다. 건조 후, 잎은 조절된 TSB 배지에서 배양한 미생물을 동일한 농도로 조절하여 침지 시켰다. 분리한 미생물의 염기서열을 비교하였을 때, 99% 이상 일치율을 나타낸 미생물은 제외하였으며 6개 미생물들은 OD600 0.3으로 조절하였다. 동일한 농도로 조절된 미생물들은 1% carboxy methyl cellulose (CMC)를 9:1 비율로 섞은 후, 선발 미생물에 잎을 10분간 침지하였고 무균대에서 건조시켰다. E. amylovora 처리구는 filter paper에 잎을 놓고 병원균의 농도를 OD600 0.1로 조절하여 스프레이 형식으로 분주하였고 30분간 건조시켰다. 건조된 잎을 올리기 위해 15 cm petri dish (SPL, Republic of Korea)에 filter paper를 깔고 멸균수를 넣어 수분을 유지시켰으며, 잎이 물에 직접적으로 접촉하지 아니하게 나무젓가락 위에 잎을 올려주었다. Petri dish는 랩으로 밀봉하여 28도 배양기에서 8일 동안 보관하였다. 실험 진행 후, 발생한 세균 유출물(ooze)은 멸균수 100 μL와 섞어 도말을 진행하였으며, 생장한 균체는 DNA 추출 및 PCR, 정제 과정을 거쳐 염기서열 분석을 진행하였다.
결과 및 고찰
화상병원균 억제 사과 내생 미생물 선발
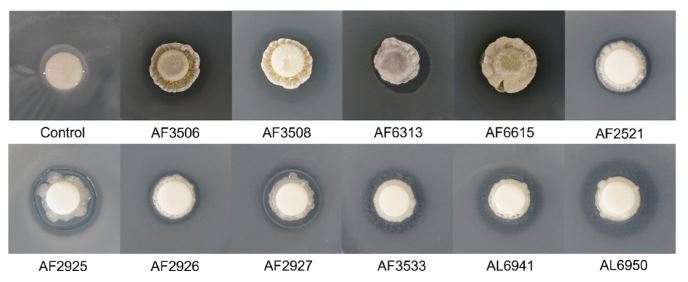
충청북도 음성군 농가에서 2020년 3월 9일부터 2020년 7월 22일까지 총 6회에 걸쳐서 사과의 조직별 시료를 채집하였다. 표면살균후 glycerol과 함께 -20oC에서 냉동 보관된 시료는 원액을 도말 후, E. amylovora와 1차 대치배양을 진행하였다. 그 결과로 총 181개의 균주가 분리되었고 2차, 3차 선발을 통해 과실에서 2개, 꽃 봉오리에서 3개, 꽃에서 4개 그리고 잎에서 2개를 확보하였고 최종적으로 11개의 후보 미생물을 선발하였다(Table 1; Fig. 1). 사과 과수 조직별로 확보된 11개의 균주는 E. amylovora에 대하여 0.5~5 mm까지 항세균능력을 나타내었다. 그 중 잎에서 분리된 2개의 균주 AL6941, AL6950는 3~5mm의 clear zone을 나타냈으며 가장 뛰어난 활성 효과를 확인하였다(Fig. 1).
화상병원균 억제 내생 미생물의 효소 활성
조직 별 사과 시료에서 선발된 총 11개의 균주는 cellulase, chitinase, proteinase, siderophore 효소 활성 실험을 진행하였다(Fig. 2). 대부분 균주들은 cellulase와 siderophore 활성과 생성은 낮았으나, chitinase와 proteinase의 활성은 높았다. AF2925, AF3533, AL6941, AL6950 균주는 cellulose 분해 배지에서 clear zone 길이 4~6 mm로 가장 좋은 활성을 나타냈다(Fig. 2a). Chitinase 활성 실험에서는 AF3506, AF3508, AF6313, AF6615는 낮은 활성을 보였다. 반대로 AF2925, AF2927, AF3533, AL6941, AL6950는 6~10 mm의 clear zone을 나타냈다(Fig. 2b). Protein 분해 활성은 AF2925를 제외한 나머지 균주 모두 clear zone을 8 mm 넘는 것으로 확인되었다(Fig. 2c). Siderophore의 생성 실험에서 AF6615균주가 3~4mm로 가장 큰 siderophore 생성 능력을 나타냈으며 나머지 균주들은 활성을 나타내지 않았다(Fig. 2d). AF2927, AF3533, AL6941, AL6950는 공통적으로 cellulose, chitin 분해 활성이 다른 균주에 비해 뛰어났고 AF3506, AF3508, AF6313, AF6615는 높은 protein 분해 활성을 나타내었다.
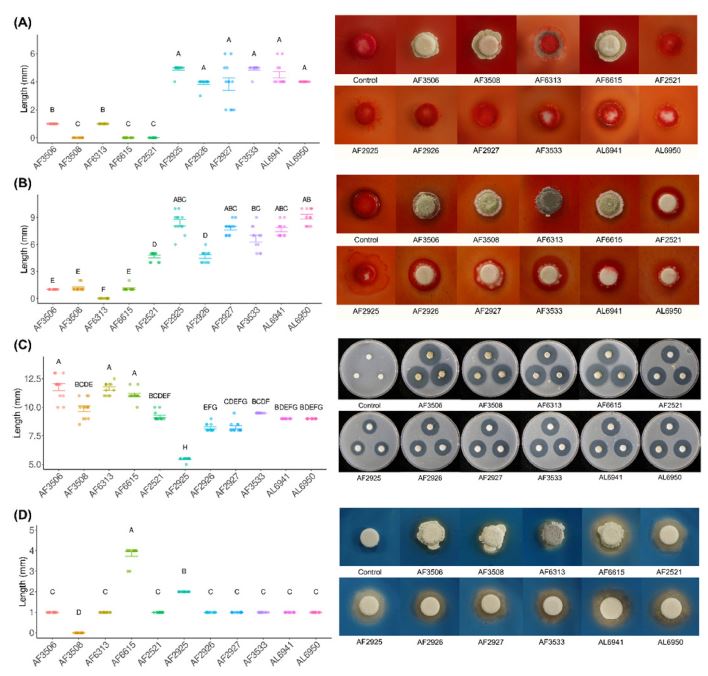
Enzyme assay of the selected bacteria. The bacteria are inoculated to OD600 0.3. After 5~7 days in 28oC, Clear zone is measured in enzyme test (n = 12). Statistical analysis is progressed using Kruskal-Wallis test and post-hoc is processed using Tukey HSD method. (A): Cellulose digestion ability in cellulose digest media. After 7 days of culturing bacteria, 0.1% Congo Red is overlaid in plates. (B): Chitin digestion ability in chitin digest media. After 7 days of culturing bacteria, 0.2% Congo Red is overlaid in plates. (C): Protein digestion ability in protein digest media. (D): Siderophore production in CAS Blue Agar media.
화상병원균 억제 사과 내생미생물의 계통학적 유연관계
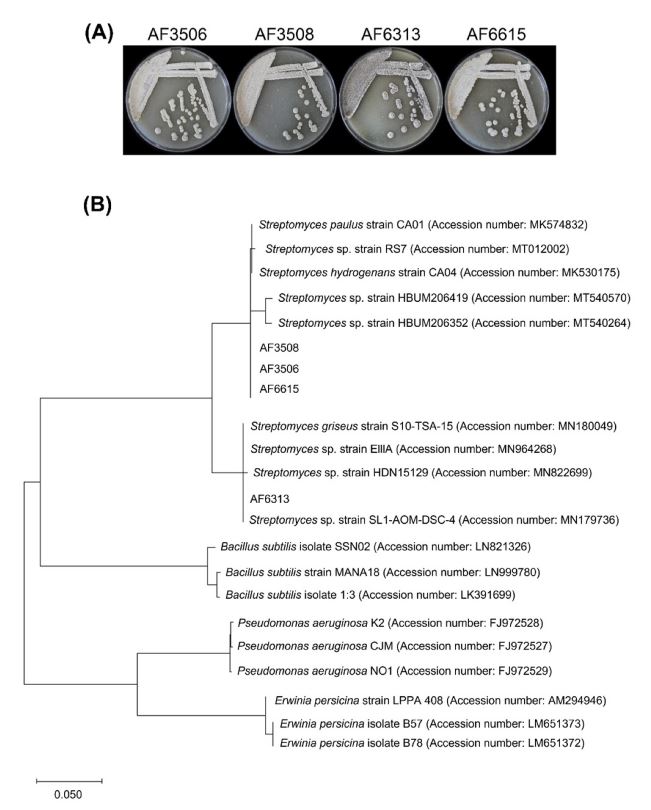
E. amylovora에 대해 높은 항균능력을 나타낸 11개의 사과 내생미생물은 16S rRNA 영역을 이용하여 동정을 진행하였다. 그 결과 98% 이상 일치율로 AF2521, AF2925, AF2926, AF2927, AF3533, AL6941, AL6950는 Paenibacillus sp., AF3506, AF3508, AF6313, AF6615는 Streptomyces sp.로 분류 동정되었다(Fig. 3; Fig. 4). E. amylovora에 대한 항균능력을 가지는 다양한 균들이 보고되어 있다. 그 중 Streptomyces의 다양한 대사산물은 E. amylovora를 억제한다고 밝혀진 바가 있다(Doolotkeldieva and Bobusheva, 2016). 또한, Paenibacillus sp.는 sucrose를 사용하여 EPS (Exopolysaccharide)를 생산할 수 있는 것으로 보고되어 있다(Choi et al., 2011; Wang et al., 2011). 특히 Paenibacillus sp.는 sucrose의 양분이용 경쟁을 통해 E. amylovora의 성장을 억제할 수 있을 것으로 판단되고, 사과의 생육시기중 꽃과 잎 조직에 존재하며, 각 조직에서 우점이 가능한 미생물로 판단된다. 향후 E. amylovora를 억제하는 것으로 알려진 분리된 11개 미생물(Streptomyces sp., Paenibacillus sp.)의 화상병의 생물학적 방제 능력 및 병원균 억제 기작을 생화학적 유전학적으로 구명하여 과수 화상병의 친환경적이고 효율적인 관리에 기여할 수 있을 것으로 사료된다.

Identification of the selected bacteria. (A): Colony type of the selected bacteria. The bacteria are streaked in MS media and incubated in 28oC for 5 days. (B): Phylogenetic tree of Streptomyces 16S rRNA region (1.5 kb) through the maximum-likelihood of MEGA 10 program.
화상병원균 선발 억제 미생물 능력 검증 및 병원균 재분리
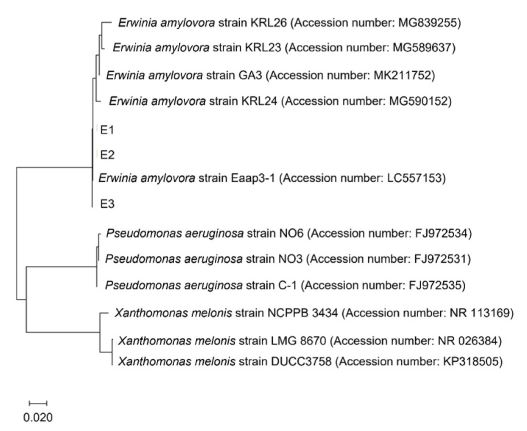
화상병원균에 대해 항균능력을 나타낸 11개의 내생 미생물 중 염기 서열 비교 후, 최종 6개의 미생물은 사과의 어린잎에 E. amylovora 억제 능력 검증 실험에 사용하였다. 6개 균주는 모두 육안상으로 억제 능력이 보였으며, 특히 Paenibacillus sp. AF2927 균주는 다른 균주에 비해 뛰어난 억제 능력을 보였다(Fig. 5). Welch's t-test를 통해 통계적 처리를 진행하였을 때, Paenibacillus sp. AF2521와 AF2927 균주 만이 통계적 차이가 유의미함을 나타내었다(Fig. 6). E. amylovora을 처리하지 않은 처리구에도 갈변되는 잎이 존재하였는데, 이는 기존에 다른 부생균의 우점 현상 또는 잎 자체가 시든 현상으로 사료된다. E. amylovora 처리구에서 나타난 세균성 점액질은 DNA 추출 및 염기서열 분석을 통해 E. amylovora와 100% 일치함을 확인하였다(Fig. 7).
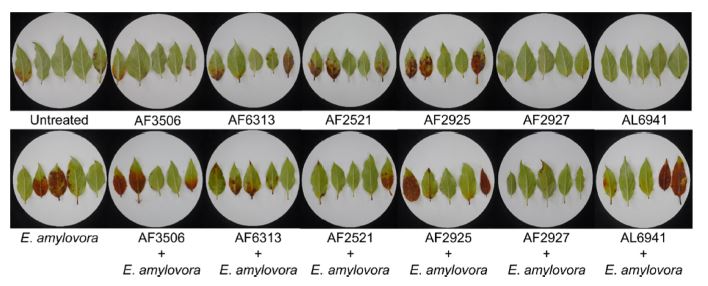
Disease prevent assay with the selected apple endophytic bacteria. Images were taken at 5 days after inoculation.
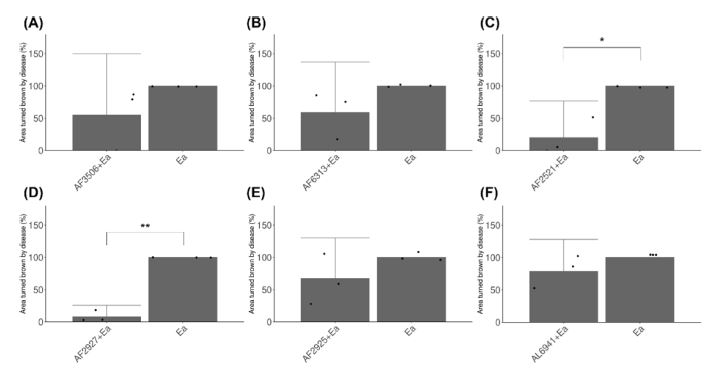
Biocontrol assay in apple leaves. Comparing the nucleotide sequence of the isolated bacteria, the experiment is conducted with 6 bacteria except to bacteria showed 99% or more identification rate. Bacteria and E. amylovora are treated by adjusting OD600 0.3, 0.1 in apple leaves and area turned brown by disease is measured (n = 3). Welch's t-test is used to statistical analysis between the group, treated bacteria and E. amylovora, and treatment of E. amylovora. (A): Comparison of AF3506+E. amylovora and E. amylovora; (B): Comparison of AF6313+E. amylovora and E. amylovora; (C): Comparison of AF2521+E. amylovora and E. amylovora; (D): Comparison of AF2927+E. amylovora and E. amylovora; (E): Comparison of AF2925+E. amylovora and E. amylovora; (F): Comparison of AL6941+E. amylovora and E. amylovora.
Acknowledgments
본 연구는 농촌진흥청의 연구과제(PJ014934)의 지원으로 수행되었습니다.
이해상충관계
저자는 이해상충관계가 없음을 선언합니다.
References
-
Aćimović SG, Zeng Q, McGhee GC, Sundin GW, Wise JC, 2015. Control of fire blight (Erwinia amylovora) on apple trees with trunk-injected plant resistance inducers and antibiotics and assessment of induction of pathogenesis-related protein genes. Front. Plant Sci. 6:16.
[https://doi.org/10.3389/fpls.2015.00016]

-
Berg G, Krechel A, Ditz M, Sikora RA, Ulrich A, et al., 2005. Endophytic and ectophytic potato associated bacterial communities differ in structure and antagonistic function against plant pathogenic fungi. FEMS Microbiol. Ecol. 51(2):215-229.
[https://doi.org/10.1016/j.femsec.2004.08.006]

-
Broggini GA, Duffy B, Holliger E, Schärer HJ, Gessler C, et al., 2005. Detection of the fire blight biocontrol agent Bacillus subtilis BD170 (Biopro®) in a Swiss apple orchard. Eur. J. Plant Pathol. 111(2):93-100.
[https://doi.org/10.1007/s10658-004-1423-x]

-
Born Y, Fieseler L, Thöny V, Leimer N, Duffy B, et al., 2017. Engineering of bacteriophages Y2:: dpoL1-C and Y2:: luxAB for efficient control and rapid detection of the fire blight pathogen, Erwinia amylovora. Appl. Environ. Microbiol. 83(12):e00341-17.
[https://doi.org/10.1128/AEM.00341-17]

-
Bubán T, Orosz-Kovács Zs, 2003. The nectary as the primary site of infection by Erwinia amylovora (Burr.) Winslow et al.: a mini review. Plant Syst. Evol. 238(1):183-194.
[https://doi.org/10.1007/s00606-002-0266-1]

- Cabrefiga J, Bonaterra A, Montesinos E, 2007. Mechanisms of antagonism of Pseudomonas fluorescens EPS62e against Erwinia amylovora, the causal agent of fire blight. Int. Microbiol. 10(2):123-132.
-
Carder JH, 1986. Detection and quantitation of cellulase by Congo red staining of substrates in a cup-plate diffusion assay. Anal. Biochem. 153(1):75-79.
[https://doi.org/10.1016/0003-2697(86)90063-1]

-
Choi HJ, Kim YE, Bang JH, Kim DW, Ahn CS, et al., 2011. Characterization of an indigenous antimicrobial substance-producing Paenibacillus sp. BCNU 5011. KSBB Journal. 26(2):100-106.
[https://doi.org/10.7841/ksbbj.2011.26.2.100]

-
Doolotkeldieva T, Bobusheva S, 2016. Fire blight disease caused by Erwinia amylovora on Rosaceae plants in Kyrgyzstan and biological agents to control this disease. Adv. Appl. Microbiol. 6(11):831.
[https://doi.org/10.4236/aim.2016.611080]

-
Graham J, Marshall B, Squire GR, 2003. Genetic differentiation over a spatial environmental gradient in wild Rubus ideaus populations. New Phytol. 157(3):667-675.
[https://doi.org/10.1046/j.1469-8137.2003.00693.x]

-
Halder D, Mandal M, Chatterjee SS, Pal NK, Mandal S, 2017. Indigenous probiotic Lactobacillus isolates presenting antibiotic like activity against human pathogenic bacteria. Biomedicines. 5(2):31.
[https://doi.org/10.3390/biomedicines5020031]

-
Hwang DH, Hong SW, Hwang HS, Chung KS, 2016. Purification and characterization of a chitinolytic enzyme produced by Bacillus licheniformis GA9. Kor. J. Microbiol. Biotechnol. 44(4):470-478.
[https://doi.org/10.4014/mbl.1607.07006]

-
Kim DR, Kwak YS, 2021. A genome-wide analysis of antibiotic producing genes in Streptomyces globisporus SP6C4. Plant Pathol. J. 37(4):389-395.
[https://doi.org/10.5423/PPJ.NT.03.2021.0047]

-
Kim YE, Kim JY, Noh HJ, Lee DH, Kim SS, et al., 2019. Investigating survival of Erwinia amylovora from fire blight-diseased apple and pear trees buried in soil as control measure. Korean. J. Environ. Agric, 38(4):269-272.
[https://doi.org/10.5338/KJEA.2019.38.4.36]

-
Lee MS, Lee IG, Kim SK, Oh CS, Park DH, 2018. In vitro screening of antibacterial agents for suppression of fire blight disease in Korea. Plant Dis. 24(1):41-51.
[https://doi.org/10.5423/RPD.2018.24.1.41]

-
Louden BC, Haarmann D, Lynne AM, 2011. Use of blue agar CAS assay for siderophore detection. J. Microbiol. Biol. Educ. 12(1):51-53.
[https://doi.org/10.1128/jmbe.v12i1.249]

-
McManus PS, Stockwell VO, Sundin GW, Jones AL, 2002. Antibiotic use in plant agriculture. Annu. Rev. Phytopathol. 40(1):443-465.
[https://doi.org/10.1146/annurev.phyto.40.120301.093927]

-
Miller TD, Schroth MN, 1972. Monitoring the epiphytic population of Erwinia amylovora. Phytopathology. 62:1175-1182.
[https://doi.org/10.1094/Phyto-62-1175]

-
Myung IS, Yun MJ, Lee YH, Kim GD, Lee YK, 2016. First report of fire blight caused by Erwinia amylovora on Chinese quince in South Korea. Plant Dis. 100(12):2521-2521.
[https://doi.org/10.1094/PDIS-04-16-0562-PDN]

-
Ordax M, Piquer-Salcedo JE, Santander RD, Sabater-Munoz B, Biosca EG, et al., 2015. Medfly Ceratitis capitata as potential vector for fire blight pathogen Erwinia amylovora: survival and transmission. PLoS ONE. 10(5):e0127560.
[https://doi.org/10.1371/journal.pone.0127560]

-
Park DH, Yu JG, Oh EJ, Han KS, Yea MC, et al., 2016. First report of fire blight disease on Asian pear caused by Erwinia amylovora in Korea. Plant Dis. 100(9):1946-1946.
[https://doi.org/10.1094/PDIS-11-15-1364-PDN]

-
Pusey PL, Stockwell VO, Reardon CL, Smits THM, Duffy B, 2011. Antibiosis activity of Pantoea agglomerans biocontrol strain E325 against Erwinia amylovora on apple flower stigmas. Phytopathology. 101(10):1234-1241.
[https://doi.org/10.1094/PHYTO-09-10-0253]

- Roselló G, Francés J, Daranas N, Montesinos E, Bonaterra A, 2017. Control of fire blight of pear trees with mixed inocula of two Lactobacillus plantarum strains and lactic acid. J. Plant Pathol. 99:111-120.
-
Sholberg PL, Bedford KE, Haag P, Randall P, 2001. Survey of Erwinia amylovora isolates from British Columbia for resistance to bactericides and virulence on apple. Can. J. Plant Pathol. 23(1):60-67.
[https://doi.org/10.1080/07060660109506910]

-
Slack S, Walters KJ, Outwater C, Sundin GW, 2020. Effect of kasugamycin, oxytetracycline, and streptomycin on in-orchard population dynamics of Erwinia amylovora on apple flower stigmas. Plant Dis. PDIS-07.
[https://doi.org/10.1094/PDIS-07-20-1469-RE]

-
Wang CL, Huang TH, Liang TW, Fang CY, Wang SL, 2011. Production and characterization of exopolysaccharides and antioxidant from Paenibacillus sp. TKU023. N. Biotechnol. 28(6):559-565.
[https://doi.org/10.1016/j.nbt.2011.03.003]

-
Yao H, Sun X, He C, Maitra P, Li XC, et al., 2019. Phyllosphere epiphytic and endophytic fungal community and network structures differ in a tropical mangrove ecosystem. Microbiome. 7(57).
[https://doi.org/10.1186/s40168-019-0671-0]

-
Zhao Y, Tian Y, Wang L, Geng G, Zhao W, et al., 2019. Fire blight disease, a fast-approaching threat to apple and pear production in China. Journal of Integrative Agriculture. 18(4):815-820.
[https://doi.org/10.1016/S2095-3119(18)62033-7]

Su-Hyeon Kim, Division of Applied Life Science (BK21Plus), Gyeongsang National University, Graduate student
Su In Lee, Department of Plant Medicine, Gyeongsang National University, Undergraduate student
Hyun Gi Kong, Crop Protection Division, National Institute of Agricultural Science, Rural Development Administration, Researcher
Gyeongjun Cho, Research Institute of Life Science, Gyeongsang National University, Researcher
Youn-Sig Kwak, Research Institute of Life Science, Gyeongsang National University, Professor, http://orcid.org/0000-0003-2139-1808