고추 재배지 TSWV 감염 파총채벌레(Thrips tabaci) 발견 및 체내 바이러스 증식
 ; Eticha Abdisa1 ; Mojtaba Esmaeily1 ; Falguni Khan1 ; Dae-Hong Lee2 ; Yonggyun Kim1, *
; Eticha Abdisa1 ; Mojtaba Esmaeily1 ; Falguni Khan1 ; Dae-Hong Lee2 ; Yonggyun Kim1, *
초록
다양한 식물을 대상으로 식물병을 유발하는 토마토반점위조바이러스(Tomato spotted wilt virus: TSWV)가 고추에 막대한 경제적 피해를 주고 있다. 이 바이러스의 수평전파는 일부 총채벌레에 의존하는 데, 국내에서 재배되는 고추에서는 꽃노랑총채벌레(Frankliniella occidentalis)와 대만총채벌레(F. intonsa)가 주요 매개충으로 알려졌다. 본 연구는 이들 두 총채벌레 이외에 파총채벌레(Thrips tabaci)도 고추에 TSWV를 매개할 수 있다는 결과를 보고한다. 월동 초기 고추 육묘장에 파총채벌레가 황색점착트랩에 포획되었다. 다중 PCR 진단기술을 이용하여 전체 포획 총채벌레에 대해서 기주와 TSWV 감염 유무를 판정한 결과, 기존의 두 총채벌레에 비해 더 높은 비율로 파총채벌레가 TSWV를 보독하는 것을 확인하였다. 보독된 파총채벌레에서 TSWV 유전자 가운데 N 유전자 염기서열(777 nucleotides)을 분석한 결과 동일 지역에서 보독된 꽃노랑총채벌레 및 대만총채벌레에서 유래된 TSWV 유전자 서열과 단 1개의 nucleotide를 제외하고 모두 일치하였다. 실제로 이들 총채벌레를 채집한 육묘장의 고추에서는 TSWV 감염 병징을 나타냈다. 보독된 파총채벌레 체내에서 바이러스 입자를 확인하기 위해 면역형광기법으로 상이한 조직을 관찰한 결과 중장 상피세포와 침샘에서 특이적으로 관찰되었다. 또한, 이 바이러스가 파총채벌레 체내에서 증식하는지를 알아보기 위해 RT-qPCR로 시기별로 바이러스 농도를 분석한 결과 유충에서 성충으로 발육하는 동안 최소 2회의 바이러스 농도 증가 피크를 보였다. 이상의 결과는 국내 서식하는 파총채벌레가 고추를 대상으로 TSWV 매개 능력을 보유한다는 것으로 제시하고 있다.
Abstract
Tomato spotted wilt virus (TSWV) infects various plant hosts and gives a massive economic damage to the hot pepper, Capsicum annuum. It is horizontally transmitted by specific thrips. In Korea, two types of the flower thrips (F. occidentalis and F. intonsa) mainly transmit the virus in the hot peppers. This study reports another candidate vector, the onion thrips, Thrips tabaci, which transmits the virus to the hot peppers. In early spring, the overwintering T. tabaci adults were captured on yellow sticky traps in the plant nurseries cultivating hot peppers. Using a multiplex diagnostic PCR technique, host thrips and their TSWV infections were determined. This diagnosis indicated that the viruliferous rate was higher in T. tabaci than that in F. occidentalis or in F. intonsa. To confirm the TSWV in the onion thrips, N gene encoded in TSWV was sequenced from the infected T. tabaci and showed only one site difference among 777 nucleotides compared to those derived from viruliferous F. occidentalis or F. intonsa. In fact, some hot plants in the nurseries exhibited a typical disease symptom of the TSWV infection. To monitor the viral particles in the viruliferous T. tabaci, TSWV was assessed in different tissues of the thrips by an immunofluorescence assay and specifically detected in the midgut epithelium and the salivary gland. To analyze the viral replication in T. tabaci, the viral titers were assessed by RT-qPCR and showed at least two increase peaks during immature development. These results suggest that the onion thrips is able to transmit TSWV to the hot peppers in Korea.
Keywords:
Onion thrips, TSWV, molecular diagnosis, insect vector키워드:
파총채벌레, 토마토반점위조바이러스, 분자진단, 매개충서 론
다양한 기주 범위를 가지고 농작물에 큰 피해를 주고 있는 토마토반점위조바이러스(Tomato spotted wilt virus: TSWV)는 총채벌레에 의해 전파된다(Gilbertson et al., 2015). 국내 서식하는 식물에 대해서도 기주 범위가 조사되었는데, 고추를 포함한 가지과는 물론이고 다양한 잡초를 감염시키는 것으로 획인되었다(Kil et al., 2020). 이 바이러스는 3개의 조각으로 구성된 RNA 게놈을 가지고 있으며 총 5가지의 단백질을 암호하고 있다. 특히 중간 크기의 게놈 조각인 M segment에는 GN/GC 유전자가 외피단백질을 지령하는데 이 단백질이 매개충의 중장세포 침입에 중요한 역할을 담당하는 것으로 알려지고 있다(Badillo-Vargas et al., 2019). 한편 작은 크기의 게놈 조각인 S segment에는 NSs라는 유전자가 기주 세포가 RNA 게놈에 대해서 항바이러스 기작을 유발하는 RNA 간섭 기구를 억제하여 자신의 게놈을 기주로부터 보호하는 기능을 담당하는 데 이러한 공통 작용이 식물은 물론이고 곤충 기주에서도 동일하게 작용하고 있다(Kim and Kim, 2022). 따라서 TSWV는 식물은 물론이고 곤충과 같은 동물에도 감염시키는 바이러스로서 주목받아 왔다(Reitz et al., 2020).
TSWV를 식물체로 매개하는 총채벌레는 총 9종으로 국한된다(Rotenberg et al., 2015). 세계적으로 약 7,700종이 알려진 총채벌레의 다양성을 비교하여 볼 때 바이러스와 총채벌레 사이에 선택적 분자 관계를 이루고 있다고 해석된다. 이들 TSWV 매개충 가운데 국내에는 꽃노랑총채벌레(Frankliniella occidentalis), 대만총채벌레(F. intonsa), 엉겅퀴총채벌레(Thrips setosus) 그리고 파총채벌레(T. tabaci) 등 4종이 분포하고 있다(Lee et al., 2001; Kim et al., 2022a). 이러한 국내 여건을 기반으로 총채벌레와 TSWV를 동시에 판별할 수 있는 다중 PCR 진단법이 개발되었다(Yoon et al., 2020). 이 기술은 TSWV와 두 종의 총채벌레인 꽃노랑총채벌레와 대만총채벌레를 대상으로 판별하는 기술로서 국화재배지를 대상으로 이 기술을 적용한 결과 이들 두 종의 총채벌레에 대한 보독 유무를 판별하였다. 흥미로운 사실은 이 연구결과에서 미동정 총채벌레에서 보독률이 높았는데 국화 기주로 볼 때 파총채벌레가 보독되었을 것으로 추정하였다. 이를 위해서는 파총채벌레 특이적 프라이머를 추가하여 3종의 총채벌레와 더불어 TSWV를 진단하는 다중 PCR 진단법을 개발할 필요가 있었다.
파총채벌레는 파, 마늘, 양파 등 백합과 및 감자, 가지, 고추 등 가지과에 속하는 여러 작물을 대상으로 유충이 엽육조직을 가해하면서 기주에 직접적 피해를 주는 것으로 알려져 있다(Wolfenbarger and Hibbs, 1958). 또한 파총채벌레는 TSWV를 비롯하여 국내에서는 아직 보고되지 않았지만 오소토스포바이러스(orthotospovirus) 속에 속한 Irish yellow spot virus를 매개하는 간접피해도 주는 것으로 알려져 있다(Gent et al., 2007). 따라서 이 해충을 방제하기 위해 약 90% 이상의 높은 야외 방제효과를 주는 네오니코티노이드, 스피노사드, 유기인계 또는 곤충생장조절제 등의 화학농약이 선발되었다(Kim et al., 2009; Kim et al., 2010; Park et al., 2019). 그러나 외국의 사례에서 보듯 이러한 화학 약제를 지속적으로 살포함에도 불구하고 이들 파총채벌레의 발생이 누그러들지 않는 것이 현실이고(Hsu et al., 2010), 여기에 복합 살충제 저항성을 발달시켜(Adesanya et al., 2020) 이 해충을 효과적으로 방제하기 위한 대체 방제 기술 개발이 절실하다. 더욱이 이 해충이 국내 월동은 물론이고 비교적 활발한 분산행동으로 여러 지역으로 보독충의 전파가 우려된다(Kim et al., 2021a). 단, 외국의 사례에서 보면 파총채벌레가 다양한 기주 또는 생식 집단에 따라 TSWV의 매개능력이 다양하기에(Westmore et al., 2013) 국내 파총채벌레가 이 바이러스를 매개할 수 있는지는 조사된 바가 없다.
안동과 영양지역은 대표적 국내 고추 산지로 39종의 고추 가해 식물병 가운데 바이러스에 의한 발병률이 가장 높다(Seo et al., 2011). 특히 고추 잎과 열매에 동심원 모양의 병징을 보이면서 특히 열매 피해 부위가 수확 후 건조 이후에도 퇴색된 상태로 남아있어 상품성이 저하되는 일명 칼라병으로 TSWV에 의해 발병된다(Moon et al., 2006; Seo et al., 2018). 고추 정식 이후에 시설재배지와 노지에서 발생하는 총채벌레를 연중 조사한 결과 꽃노랑총채벌레와 대만총채벌레가 전체 총채벌레 발생의 96% 이상을 차지하고 이들에게서 TSWV가 검출되어 이들이 고추 칼라병을 일으키는 TSWV의 주요 매개충으로 판명되었다(Kim et al., 2022a,b). 그러나 이 경북 북부지역에서 정식 이전의 육묘장에서 발생하는 총채벌레에 대해서는 아직 조사가 미흡하였다.
본 연구는 영양지역을 중심으로 월동 직후 고추 육묘장에서 포획되는 총채벌레를 조사하였다. 이 가운데 파총채벌레를 확인하였고, 이 종에서 TSWV 보독 유무를 다중 PCR 진단으로 분석하였다. 또한 TSWV가 파총채벌레에서 증식할 수 있는지를 면역형광기법 및 바이러스 정량 분석을 통해 추적하였다. 이러한 결과들을 통해 본 연구는 국내 파총채벌레 집단이 고추에 TSWV를 매개하는 능력이 있다는 것을 보고한다.
재료 및 방법
시험장소 및 모니터링
2023년 2월 9일부터 3월 31일까지 경북 영양읍, 청기면, 수비면에 소재한 시설 고추 육묘장에서 월동충을 대상으로 모니터링이 실시되었다. 포획용 트랩으로 황색점착트랩(10 × 7.5 cm, Green Agrotech, Gyungsan, Korea)을 이용하였고, 각 육묘장에 3반복으로 2월 6일에 설치하였고, 조사 기간 매주 수거하였다. 수거된 트랩은 총채벌레류의 밀도 조사를 위해 실험실에서 Kim et al. (2021b)의 종 특이적 형태 형질을 바탕으로 총채벌레를 구분하였다.
파총채벌레 실내 사육
경북 안동지역에서 재배되는 대파에 발생한 파총채벌레를 채집하였다(Khan et al., 2022). 사육 환경조건은 온도 25±2oC, 상대습도 65± 5%, 14 시간 광주기 조건을 유지하였다. 원형 사육 용기(지름 100 mm, 높이 40 mm)에 알부터 성충까지 사육하였으며, 대파(Allium fistulosum)를 유충과 성충의 먹이로 제공하였다.
다중 PCR 진단기술
황색점착트랩에 포획된 총채벌레의 TSWV 보독 여부를 다중 PCR 검정기술을 통하여 진단하였다. 기본적으로 Kim et al. (2022a)의 방법을 이용하였으며, 여기에 파총채벌레의 특이적 프라이머(Table 1)를 추가하여 3종의 총채벌레를 TSWV 진단과 동시에 구분하게 하였다. 이 PCR 분석을 위해 총채벌레 성충 1마리를 점착 트랩에서 헥산을 이용하여 분리한 후 5분간 건조하였다. 건조된 샘플은 QuickExtract DNA Extraction Solution (Bio-search, Hoddesdon, UK)을 이용하여 핵산을 추출하였다. 추출된 핵산은 Supreme RT-PCR premix (Genetbio, Daejeon, Korea)를 이용하여 50oC에서 30분간 역전사시켰다. 얻어진 cDNA는 다시 95oC에서 2분간 초기 변성과정 이후 35회 PCR이 95oC-30초, 55oC-30초, 72oC-45초의 온도 주기로 진행되었다. PCR 조성은 추출 핵산 4 μL, 종특이적 프라이머(Table 1) 1 μL, RT-PCR premix 12 μL로 구성되었다. 이러한 일련의 반응 후 72oC에서 10분 동안 최종 사슬연장을 진행하였다. 이후 1% 아가로즈젤에 전기영동하여 총채벌레 종류와 바이러스 보독 유무를 판명하였다.
TSWV 유전자 서열 분석
TSWV 게놈에 존재하는 N 유전자의 open reading frame (ORF) 서열을 얻기 위해 Kim et al. (2021b)이 제작한 프라이머를 이용하였다. PCR 반응은 초기 열처리(95oC, 2분) 이후 DNA 변성조건(95oC, 30초), 프라이머결합조건(52oC, 30초), 사슬연장조건(72oC, 1분)의 온도순환을 40회 반복하였다. PCR 증폭물은 PCR2.1 클로닝벡터(Thermo Fisher Scientific Korea)에 삽입하여 Top10 대장균 균주를 통해 클로닝하였다. 배양된 형질전환 대장균에서 재조합 벡터를 추출하여 염기서열 분석에 이용되었다. 염기서열 분석은 M13F와 M13R의 universal 프라이머를 이용하여 양방향으로 진행되었다(Macrogen, Seoul, Korea). 분석된 DNA 서열들은 Seqman 프로그램(DNAStar, Madison, WI, USA)을 이용하여 N 유전자 서열을 얻었다. 서로 다른 총채벌레에서 유래된 바이러스 N 유전자 서열은 DNAStar의 SeqAlign 프로그램을 이용하여 ClustalW 모드에서 상호 비교되고 이들의 서열 차이를 분석하였다.
TSWV 충체 감염 및 바이러스 농도 분석
TSWV에 감염된 고추로부터 바이러스 RNA 게놈은 Viral Gene-spin Viral DNA/RNA Extraction Kit (Intron, Sungnam, Korea)를 이용하여 추출하였다. 간략하게 기술하면, 감염 고춧잎(지름 0.5 cm 원형 시료)을 1.5 mL 튜브 넣고 500 μL의 용해용 완충용액을 첨가한 후 멸균된 막대로 마쇄하였다. 이후 10분간 상온에서 반응한 후 250 μL 상등액을 얻었다. 여기에 결합용 완충용액을 500 μL를 첨가하고 핵산결합컬럼을 이용하여 추출된 RNA를 모았다. 이를 다시 50 μL의 RNase가 제거된 물을 이용하여 수거하였다. 이렇게 얻은 바이러스 추출물에 대파 잎을 10분간 침지시켰다. 이후 바이러스 감염은 Kim et al. (2023)의 방법에 따라 상온에서 30분 건조한 후 6시간 절식시킨 파총채벌레 1령 유충에 3시간 섭식시켰다. 이후 건전한 먹이로 상기의 사육방법으로 성충까지 발육시켰다.
총채벌레 체내 TSWV의 농도를 측정하기 위해 Kim et al. (2021b)의 방법에 따라 RT-qPCR을 진행하였다. 측정당 100마리의 개체를 하나의 시료로 Trizol RNA 추출키트(Thermo Fisher Scientific Korea, Seoul, Korea)의 제조사 방법을 따라 RNA를 추출하였다. 추출된 RNA는 iScript Select cDNA Synthesis Kit (BioRad, Hercules, CA, USA)에 포함된 random primer를 이용하여 프라이머 결합 조건(25oC, 5분), 역전사(42oC, 45분) 및 역전사 효소 불활성(85oC, 5분)을 거쳐서 cDNA를 형성하였다. 바이러스 농도 측정을 바이러스 농도와 qPCR의 Ct 사이에 표준방정식을 우선 결정한다. 이를 위해 TSWV의 N 유전자에 특이적 프라이머 세트(GAGATTCTCAGAATTCCCAGT, AGAGCAATCGTGTCAATTTTATTC)를 이용하여 증폭물을 얻었다. 이 증폭물을 Expin Gel SV (GeneAll, Seoul, Korea)를 이용하여 DNA 추출하고 Nanodrop spectrophotometer (Theromo Fisher Scientific Korea)을 이용하여 DNA 정량분석하였다. 상이한 DNA 시료로 희석한 후 Step One Plus Real time PCR system (Applied Biosystems, Waltham, MA, USA)를 이용하여 DNA 양에 따른 상이한 Ct값을 얻어 회귀방정식을 결정하였다. qPCR을 위해서 SYBR Green Realtime PCR Master Mix (Toyobo, Osaka, Japan)에 의해 qPCR이 진행되었다. 곤충 체내 TSWV 농도는 동일한 RTqPCR을 통해 Ct 값을 얻고 이를 통해 상기의 표준방정식을 통해 산출하였다. 바이러스 농도는 virions per thrips로 표시하였다.
조직내 TSWV의 면역형광분석
총채벌레의 표피를 부드럽게 만들기 위하여 5분간 인산완충용액(PBS: 137 mM NaCl, 2.7 mM KCl, 10 mM Na2HPO4, 1.8 mM KH2PO4)에 침지하였다. 침지된 총채벌레를 곤충핀(No. 1, Shiga, Higashiomi, Japan)을 이용하여 머리와 몸을 분리하여 침샘과 기타 내부장기를 분리하였다. 이후의 모든 과정은 습기를 유지하기 위해서 밀폐용기(Lock and Lock, Seoul, Korea)에서 진행되었다. 분리된 조직을 상온에서 4% 파라포름알데히드(Duksan, Seoul, Korea) 20 μL로 1시간 동안 고정하였다. 이후 PBS를 이용하여 3회 세척한 후 1% Triton X-100 이 포함된 20 μL의 인산완충용액 속에 고정된 충체 시료를 10시간 동안 4oC에서 처리하였다. 이후 5 % bovine serum albumin (BSA)이 포함된 20 μL의 PBS에서 1시간 동안 상온 처리하였다. TSWV 형광염색을 위해서 TSWV 특이적 항체(1,000배 희석, Agdia, Elkhart, IN, USA)를 5% BSA가 포함된 PBS에 3시간 동안 침지하였다. 세포질을 염색하기 위해서 Rhodamine Phalloidin (1,000배 희석, Invitrogen, Carlsbad, CA, USA)을 5% BSA가 포함된 PBS로 3시간 동안 실온에서 처리하였다. 핵을 염색하기 위하여 4′,6-diamidino-2-phenylindole (DAPI, 1,000배 희석, Invitrogen)로 실온에서 3분간 처리되었다. 현미경 사용 시 건조 방지를 위하여 50% 글리세롤(Duksan, Seoul, Korea) 처리 후 커버글래스(18 × 18 mm, Marienfeld, Königshofen, Germany)로 덮어주었다. 시료는 형광현미경(DM2500, Leica, Wetzlar, Germany)을 이용하여 200배에서 관찰 및 사진 촬영되었다.
통계처리
처리 효과는 SAS의 PROC GLM (SAS Institute, 1989)을 이용하여 one-way ANOVA 분석을 하였다. 처리 평균간 비교는 LSD 방법을 이용하여 제I형 오류 확률 0.05를 기준으로 판별하였다.
결 과
고추 육묘장 파총채벌레 발생 및 TSWV 보독
경북 영양에 소재한 4개 고추 육묘장에서 총채벌레가 발생하였다(Fig. 1). 조사기간(2023년 2월 9일부터 3월 31일) 약 7주간 전체 262마리 총채벌레가 포획되었다. 포획된 총채벌레를 형태적으로 동정하면 5마리 미동정 총채벌레를 제외하고 모두 꽃노랑총채벌레, 대만총채벌레 그리고 파총채벌레였다. 특히 2월중에는 거의 모두 꽃노랑총채벌레였으며, 대만총채벌레와 파총채벌레는 3월 이후 발생하였다(Fig. 1A). 포획 개체를 모두 다중 PCR 동정 기술로 총채벌레 종을 재확인과 동시에 TSWV 감염 유무를 분석한 결과 보독 총채벌레는 꽃노랑총채벌레, 대만총채벌레 그리고 파총채벌레에서만 나타났고, 미동정 총채벌레에서는 나타나지 않았다(Fig. 1B). 흥미롭게도 보독률을 비교하여 보면 포획 밀도는 낮지만 파총채벌레에서 가장 높게 나타났다(X2= 12.4; df = 3; P = 0.0060).
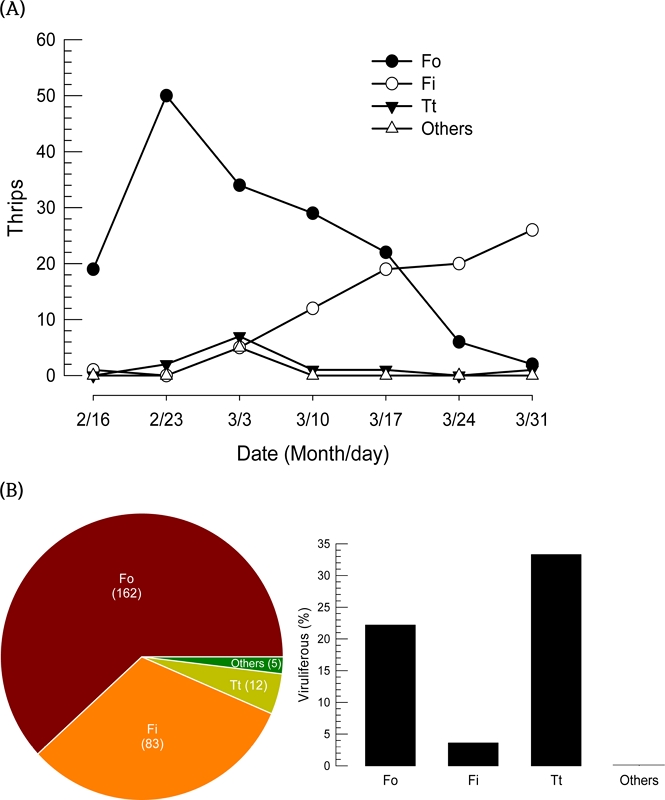
Thrips monitoring using yellow sticky traps in nursery farms at Yeonggyang in 2023. (A) Total thrips numbers in different thrips: F. occidentalis (‘Fo’), F. intonsa (‘Fi’), T. tabaci (‘Tt’), and others. (B) Comparison of TSWV-viruliferous rates among different thrips. Figures in the parentheses of the pie chart indicate the numbers of total thrips in each species.
파총채벌레에서 검출된 TSWV가 꽃노랑총채벌레 및 대만총채벌레에서 검출된 TSWV와 유전적 차이를 분석하기 위해 N 유전자를 대상으로 염기서열을 분석하였다(Fig. 2A). 전체 777개 염기서열 가운데 파총채벌레에서 분리된 TSWV는 단 1곳의 염기서열에서 꽃노랑총채벌레 또는 대만총채벌레에서 유래된 TSWV와 차이를 나타냈다. 이러한 염기서열 변화는 아미노산 서열에서도 차이를 주었다(Fig. 2B).
파총채벌레의 TSWV 매개 능력
TSWV에 보독된 파총채벌레가 체내 감염 및 증식 가능성을 분석하기 위해 실내 접종실험을 통해 분석하였다(Fig. 3). TSWV에 감염증세를 보이는 고추 식물체로부터 바이러스 추출물을 얻었다. 건전주에서 추출물에 대비하여 TSWV 항체에 특이적으로 반응하여 추출물이 TSWV를 함유한 것을 확인하였다(Fig. 3A). 이 바이러스 추출물을 파총채벌레유충에 섭식시킨 후 건전한 고추에서 발육시켜 성충에 도달하였을 때 다시 western blotting 분석을 하였다. 예상대로 건전주 추출물을 섭식한 개체에 비해 바이러스 감염 추출물을 섭식하여 발육된 성충에서 항체 특이적 반응을 보였다.
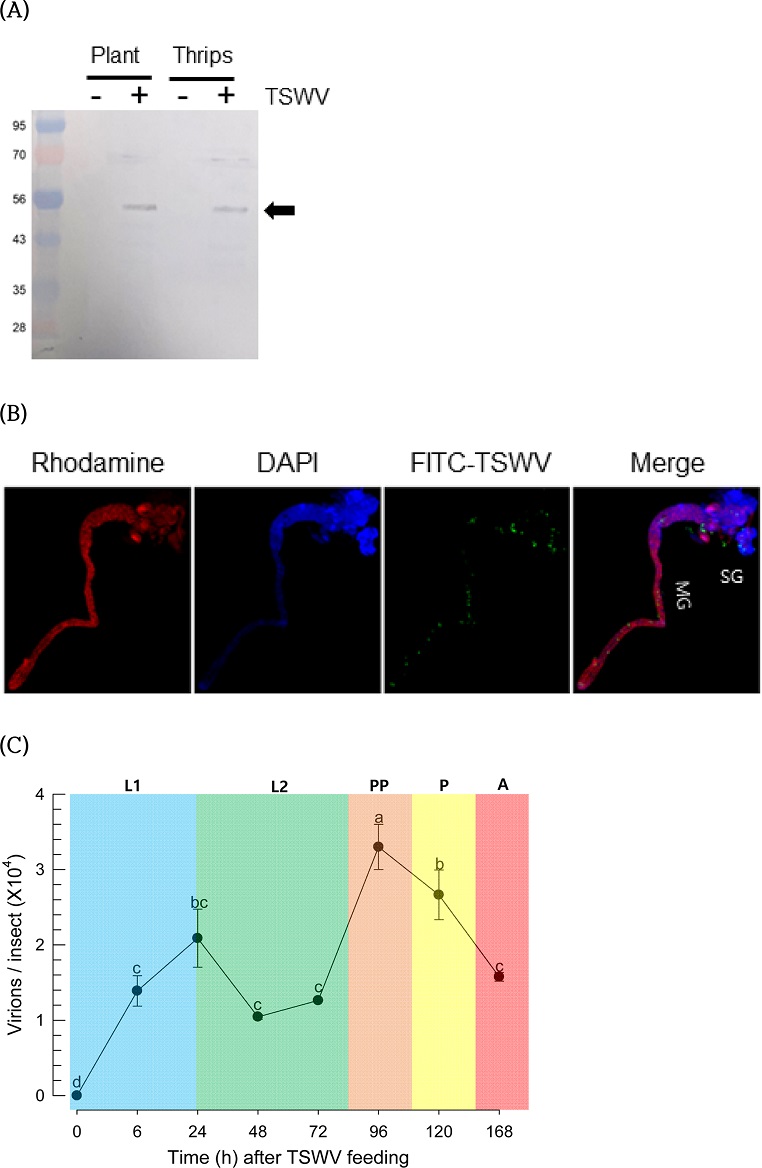
Infection of TSWV and multiplication in T. tabaci. (A) Infection of TSWV by feeding the plant extract. The extract was fed at the first instar and western analysis was performed at adult stage. Each lane contained 10 μg of proteins. ‘Plant’ indicates the extract of hot pepper exhibiting TSWV symptom, where ‘+’ and ‘-’ represent positive and negative symptom, respectively. ‘Thrips’ larvae were fed (‘+’) or not fed (‘-’) with the extract. (B) Detection of TSWV with specific antibody in the midgut (‘MG’) and salivary gland (‘SG’) of the adults treated with TSWV. (C) Quantification of TSWV titers at different developmental stages: the first and second instar larvae (‘L1’ and ‘L2’), prepupal (‘PP’), pupal (‘P’), and adult (‘A’).
TSWV 보독 성충에서 이 바이러스의 체내 분포를 면역형광기술로 분석하였다(Fig. 3B). 상이한 형광물질인 rhodamine과 DAPI로 세포질과 핵을 염색시켜 파총채벌레의 소화관과 침샘 부위를 확인하였다. 여기에 다시 TSWV에 특이적으로 반응한 항체에 FITC로 검출하였을 때 침샘을 중심으로 관찰되었으며 소화관의 여러 영역에서 이 바이러스가 검출되었다.
TSWV에 감염된 파총채벌레 체내에서 이 바이러스의 증식이 일어나는지 분석하기 위해 앞에서와 같이 1령충 시기에 바이러스를 감염시키고, 이후 건전주에서 발육시키면서 바이러스의 함량변화를 RT-qPCR로 추적하였다(Fig. 3C). 섭식 직후 6시간 경과 후 분석한 바이러스 함량은 총채벌레 개체당 약 13,500개 바이러스 밀도로 나타났다. 그러나 24시간이 경과하면서 바이러스 함량은 약 50% 증가하였다. 이후 2령충 시기에 바이러스 함량은 낮아지다가 전용 단계에서 급격하게 바이러스 함량이 증가하였다. 성충에 이르면서 이러한 바이러스 함량은 다시 줄어들어 1령 시기의 바이러스 밀도를 유지하게 되었다.
고 찰
고추에 일명 칼라병이라 불리는 바이러스 병이 수량 감소 및 상품성을 저하시켜 생산 농가에 심각한 우려를 주고 있다. 원인 병원균이 TSWV이고, 이를 매개하는 곤충이 총채벌레로 알려졌다(Rotenberg et al., 2015). 즉, 이 바이러스병의 발병을 억제하기 위해서는 매개충의 방제가 필수적이다. 이 연구가 진행되기 전에는 국내 고추에 TSWV를 매개하는 총채벌레는 꽃노랑총채벌레와 대만총채벌레로 이해되었다(Kim et al., 2022a,b). 그러나 또 다른 TSWV의 매개충으로서 국내에 자생하는 엉겅퀴총채벌레와 파총채벌레의 매개 가능성을 배제하지 않았다. 특별히 파총채벌레는 기주 범위가 매우 넓다. 국내에서 파, 마늘, 양파 등 백합과에서 많이 발생하지만(Kim et al., 2021a), 감자, 가지, 고추 등 가지과에도 빈번히 피해를 주고 있는 것으로 알려졌다(Woo et al., 1991). 여기에 시설지역에서 재배되는 아스파라거스(Asparagus officinalis L.), 난과(Orchidacea)에 속한 심비디움(Cymbidium spp.) 그리고 뽕나무과(Moraceae)에 속한 무화과(Ficus carica L.)에서도 파총채벌레의 발생하였다(Cho et al., 2013; Choi et al., 2014; Kim et al., 2014). 본 연구에서는 영양지역의 고추 재배지에서 파총채벌레가 발생하였다. 비록 꽃노랑총채벌레와 대만총채벌레 만큼의 높은 밀도는 아니지만, 이 해충 발생은 넓은 기주 범위를 갖는 이 종의 특성으로 이해될 수 있다. 특별히 발생된 파총채벌레는 3월경으로 월동충으로 여겨진다. Kim et al. (2021a)은 겨울 기간 시설재배지 대파 밭에서 파총채벌레의 대발생을 보고하였다. 아직 정확히 이들의 월동기작은 밝혀있지 않지만, 시설재배지를 중심으로 이 해충의 국내 월동이 가능하기에 본 연구에서 조사한 고추 육묘장의 발생이 이뤄진 것으로 사료된다.
월동세대에서 포획된 파총채벌레는 비교적 높은 비율로 TSWV 보독을 나타냈다. 비록 포획 밀도는 낮지만, TSWV의 주요 매개충인 꽃노랑총채벌레 보다 높은 보독률을 보였다. 이 보독률이 갖는 의미를 이해하기 위해 면역형광기술로 체내 바이러스의 분포를 조사한 결과 성충 시기에 침샘에서 바이러스 입자가 밀집되어 있는 것을 확인하였다. 꽃노랑총채벌레에서 보듯, TSWV는 감염 식물체에서 섭식을 통해 중장으로 들어오게 되고, 이후 중장 상피세포를 통해 충체로 감염된다(Hunter and Ullman, 1989; Badillo-Vargas et al., 2015). 이 세포에서 일부 증식된 TSWV는 다시 궁극적으로 침샘으로 이동하여 다음 식물체로 바이러스 전파를 꾀하게 된다(Kim et al., 2023). 즉, 본 연구에서 파총채벌레의 중장과 침샘에서 관찰된 TSWV는 이러한 바이러스의 매개충 조직 특이적 이동 현상으로 이해될 수 있다. 한편, 본 연구는 파총채벌레 유충 시기에 TSWV를 감염시킨 후 성충에 이르기까지 각 시기별로 TSWV 밀도 변화를 추적한 결과, 최소 2회 이상의 바이러스 농도 증가를 보였다. 이는 TSWV가 파총채벌레에서 바이러스 복제가 이뤄진 것으로 이해된다. 즉, 국내에 서식하는 파총채벌레는 TSWV 영속전파가 가능한 것으로 판명된다.
파총채벌레의 TSWV 매개에 관해서는 이 종의 상이한 집단에 따라 매개 능력의 차이를 보였다. 앞에서 기술한 TSWV의 전파에서 보듯 식물바이러스가 매개충의 체내 감염 및 증식을 통한 영속전파의 경우 궁극적으로 침샘에 도달한 바이러스의 밀도에 의해 매개충의 매개능력을 판단할 수 있다. 따라서 매개충 바이러스 매개 능력은 바이러스가 매개충의 체내 방어기작을 극복하는 능력을 반영하는 데 여기에 매개충의 기주 및 환경 요인과 같은 외부 요인들에 의해서도 영향을 받게 된다(Stumpf and Kennedy, 2005; Agarwal et al., 2017). 예를 들어, 꽃노랑총채벌레의 경우 TSWV 감염에 대해서 특이적 면역반응을 보였으며, 이 면역 기작을 억제할 경우 체내 바이러스 농도의 증가를 보였다(Kim et al., 2023). 파총채벌레의 TSWV 매개 능력은 집단에 따라 상이하여 특히 지역 차이보다는 매개충의 기주에 따른 유전변이에 민감한 것으로 보인다. 예를 들어, 호주의 파총채벌레 경우 지역 차이에 따른 집단 분화보다는 기주 식물에 따른 집단 분화에 민감하여 국화 또는 파 기주 집단과 달리 감자를 기주로 하는 집단에서만 TSWV를 전파하였다(Westmore et al., 2013). 여기에 생식양식에 따른 차이도 파총채벌레의 TSWV 매개 능력 변이에 영향을 주었다. 즉, 파총채벌레는 단성생식을 보이는 데 이때 담배를 기주로 하는 집단의 경우 수컷생산 단성생식을 보이지만, 파를 가해하는 총채벌레의 경우 암컷생산 단성생식을 진행한다. 이때 수컷생산 단성생식을 보이는 파총채벌레의 경우 비교적 높은 TSWV 매개능력을 보이지만 파를 기주로 하며 암컷생산 파총채벌레의 경우 TSWV 매개능력을 보이지 않았다(Chatzivassiliou et al., 2002). 한편, 본 연구에서 분석한 파총채벌레는 대파에서 채집된 집단으로서 암컷생산 단성생식을 보였다(Khan et al., 2022). 그러나 앞에서 기술한 바와 같이 TSWV의 감염 및 증식이 일어나는 것으로 미뤄 외국의 사례와 대조적 결과를 보였다. 이러한 차이는 아마도 파총채벌레의 집단 간 TSWV 매개 능력에 집단 변이의 중요성을 다시 입증하여 주는 것으로 사료된다. 향후 국내 다양한 지역 및 집단을 대상으로 파총채벌레의 TSWV 매개 능력을 비교 분석하여 볼 필요가 있다.
Acknowledgments
본 결과물은 농림축산식품부의 재원으로 농림식품기술기획평가원의 작물바이러스 및 병해충대응 산업화 기술개발사업의 지원(321100-3)을 받아 연구되었습니다.
이해상충관계
저자는 이해상충관계가 없음을 선언합니다.
References
-
Adesanya AW, Waters TD, Lavine MD, Waish DB, Lavine LC, et al., 2020. Multiple insecticide resistance in onion thrips population from Western USA. Pestic. Biochem. Physiol. 165:104553.
[https://doi.org/10.1016/j.pestbp.2020.104553]

-
Agarwal A, Parida M, Dash PK, 2017. Impact of transmission cycles and vector competence on global expansion and emergence of arboviruses. Rev. Med. Virol. 27:e1941.
[https://doi.org/10.1002/rmv.1941]

-
Badillo-Vargas IE, Chen Y, Martin KM, Rotenberg D, Whitfield AE, 2015. Discovery of novel thrips vector proteins that bind to the viral attachment protein of the plant bunyavirus Tomato Spotted Wilt Virus. J. Virol. 93:e00699-19.
[https://doi.org/10.1128/JVI.00699-19]

-
Chatzivassiliou EK, Peters D, Katis NI, 2002. The efficiency by which Thrips tabaci populations transmit Tomato spotted wilt virus depends on their host preference and reproductive strategy. Phytopathology 92:603-609.
[https://doi.org/10.1094/PHYTO.2002.92.6.603]

-
Cho MR, Jeon SW, Kang TJ, Kim HH, Ahn SJ, et al., 2013. Pests occurring on Cymbidium. Korean. J. Appl. Entomol. 52:403-408
[https://doi.org/10.5656/KSAE.2013.08.0.035]

-
Choi KS, Song JH, Yang JY, Choi H, Kim DS, 2014. Pest species, damages and seasonal occurrences on greenhouse cultivated asparagus in Jeju, Korea. Korean J. Appl. Entomol. 53:231-237.
[https://doi.org/10.5656/KSAE.2014.06.0.006]

-
Gent DH, Martin RR, Ocamb CM, 2007. First report of Iris yellow spot virus on onion and leek in Western Oregon. Plant Dis. 91:468.
[https://doi.org/10.1094/PDIS-91-4-0468A]

-
Gilbertson RL, Batuman O, Webster CG, Adkins S, 2015. Role of the insect supervectors Bemisia tabaci and Frankliniella occidentalis in the emergence and global spread of plant viruses. Annu. Rev. Virol. 2:67-93.
[https://doi.org/10.1146/annurev-virology-031413-085410]

-
Hunter WB, Ullman DE, 1989. Analysis of mouthpart movements during feeding of Frankliniella occidentalis (Pergande) and F. schultzei Trybom (Thysanoptera: Thripidae). Intl. J. Insect Morph. Embryol. 18:161-171.
[https://doi.org/10.1016/0020-7322(89)90024-X]

-
Khan F, Roy MC, Kim Y, 2022. Thelytokous reproduction of onion thrips, Thrips tabaci Lindeman 1889, infesting welsh onion and genetic variation among their subpopulations. Insects 13:78.
[https://doi.org/10.3390/insects13010078]

-
Kil EJ, Chung YJ, Choi HS, Lee S, Kim CS, 2020. Life cycle-based host range analysis for Tomato spotted wilt virus in Korea. Plant Pathol. J. 36:67-75.
[https://doi.org/10.5423/PPJ.FT.12.2019.0290]

-
Kim C, Kim Y, 2022. In vivo transient expression of a viral silencing suppressor, NSs, derived from tomato spotted wilt virus decreases insect RNAi efficiencies. Arch. Insect Biochem. Physiol. 24:e21982.
[https://doi.org/10.1002/arch.21982]

- Kim SG, Kim DI, Kang BY, 2009. Efficacy of insecticides against Thrips tabaci on spring onion. Research Report. Rural Development Administration, Jeonju, pp. 1381-1382.
- Kim SG, Kim DI, Ko SJ, Kang BY, 2010. Efficacy of insecticides against Thrips tabaci on spring onion. Research Report. Rural Development Administration, Jeonju, pp. 1189-1190.
-
Kim DH, Cho MR, Yang CY, Kang TJ, Kim HH, et al., 2014. Occurrence and damage by thrips on greenhouse cultivated fig. Korean J. Appl. Entomol. 53:485-490.
[https://doi.org/10.5656/KSAE.2014.11.0.048]

- Kim C, Choi D, Kim Y, 2021a. Report on an outbreak of the onion thrips, Thrips tabaci, infesting welsh onion during winter season. Korean J. Appl. Entomol. 60:247-254.
- Kim C, Choi D, Kang J, Ahmed S, Kil E, et al., 2021b. Thrips infesting hot pepper cultured in greenhouses and variation in gene sequences encoded in TSWV. Korean. J. Appl. Entomol., 60:387-401.
- Kim C, Choi D, Lee D, Khan F, Kwon G, et al., 2022a. Yearly occurrence of thrips infesting hot pepper in greenhouses and differential damages of dominant thrips. Korean J. Appl. Entomol. 61:319-330.
- Kim C, Choi D, Khan F, Hrithik TH, Hong J, et al., 2022b. Comparative analysis of cold tolerance and overwintering site of two flower thrips, Frankliniella occidentalis and F. intonsa. Korean J. Appl. Entomol. 61:409-422.
-
Kim C, Ahmed S, Stanley D, Kim Y, 2023. HMG-like DSP1 is a damage signal to mediate the western flower thrips, Frankliniella occidentalis, immune responses to tomato spotted wilt virus infection. Dev. Comp. Immunol. 144:104706.
[https://doi.org/10.1016/j.dci.2023.104706]

-
Lee GS, Lee JH, Kang SH, Woo KS, 2001. Thrips species (Thysanoptera: Thripidae) in winter season and their vernal activities on Jeju island, Korea. J. Asia Pac. Entomol. 4:115-122.
[https://doi.org/10.1016/S1226-8615(08)60112-0]

- Moon HC, Cho IK, Im JR, Goh BR, Kim DH, et al., 2006. Seasonal occurrence and damage by thrips on open red pepper in Jeonbuk Province. Korean J. Appl. Entomol. 45, 9-13.
- Park YU, Lee JS, Jeong JH, Min JH, Chang WB, et al., 2019. Occurrence and susceptibility to several insecticides of Thrips tabaci and Acrolepiopsis sapporensis on northern-type garlic fields in Chungbuk Province. Korean J. Appl. Entomol. 58:251-258.
-
Reitz SR, Gao Y, Kirk WDJ, Hoddle MS, Leiss KA, et al., 2020. Invasion biology, ecology, and management of western flower thrips. Annu. Rev. Entomol. 65:17-37.
[https://doi.org/10.1146/annurev-ento-011019-024947]

-
Rotenberg D, Jacobson AL, Schneweis DJ, Whitfield AE, 2015. Thrips transmission of tospoviruses. Curr. Opin. Virol. 15:80-89.
[https://doi.org/10.1016/j.coviro.2015.08.003]

- SAS Institute, Inc., 1989. SAS/STAT User’s Guide. SAS Institute, Inc., Cary, NC.
-
Seo J, Yi Y, Kim B, Hwang JM, Choi SW, 2011. Disease occurrence on red-pepper plants surveyed in Northern Kyungbuk province, 2007-2008. Res. Plant Dis. 17:205-210.
[https://doi.org/10.5423/RPD.2011.17.2.205]

-
Seo MH, Lee SC, Yang CY, Yoon JB, Park J, 2018. Monitoring occurrence status of thrips populations on field-cultivated pepper at major cultivated region in west coast, Korea. Korean J. Environ. Biol. 36:544-549.
[https://doi.org/10.11626/KJEB.2018.36.4.544]

-
Stumpf CF, Kennedy GG, 2005. Effects of tomato spotted wilt virus (TSWV) isolates, host plants, and temperature on survival, size, and development time of Frankliniella fusca. Entomol. Exp. Appl. 114:215-225.
[https://doi.org/10.1111/j.1570-7458.2005.00251.x]

-
Westmore GC, Poke FS, Allen GR, Wilson CR, 2013. Genetic and host-associated differentiation within Thrips tabaci Lindeman (Thysanoptera: Thripidae) and its links to Tomato spotted wilt virus-vector competence. Heredity 111:210-215.
[https://doi.org/10.1038/hdy.2013.39]

-
Wolfenbarger D, Hibbs ET, 1958. Onion thrips (Thrips tabaci Lind.) infesting cabbage. J. Econ. Entomol. 78:394-396.
[https://doi.org/10.1093/jee/51.3.394]

- Woo KS, Cho KS, Kweon OK, 1991. Studies on distribution, host plants and taxonomy of Korean thrips (Insecta: Thysanoptera). Seoul Nat’ Univ. J. Agric. Sci. 16:133-148.
-
Yoon JY, Yoon JB, Seo MH, Choi SK, Cho IS, et al., 2020. Application of multiplex RT-PCR for simultaneous identification of tomato spotted wilt virus and thrips species in an individual thrips on chrysanthemum. Res. Plant Dis. 26:264-271.
[https://doi.org/10.5423/RPD.2020.26.4.264]

Chulyoung Kim, Andong National University, Post Master Researcher, http://orcid.org/0009-0008-4221-9306
Eticha Abdisa, Andong National University, MS student
Falguni Khan, Andong National University, Post Master Researcher
Mojtaba Esmaeily, Andong National University, Post Doctoral researcher
Dea-Hong Lee, GyeongsangBuk-Do Agricultural Research & Extension Services Researcher
Yonggyun Kim, Andong National University, Professor, http://orcid.org/0000-0002-6840-2167
Research planning, Yonggyun Kim; Field monitoring, Chulyoung Kim, Dae-Hong Lee and Yonggyun Kim; TSWV analysis, Eticha Abdisa, Mojtaba Esmaeily and Falguni Khan; Draft writing, Chulyoung Kim and Yonggyun Kim; Review & editing, Chulyoung Kim, Eticha Abdisa, Mojtaba Esmaeily, Falguni Khan, Dae-Hong Lee and Yonggyun Kim

