우리나라 논에서 분리한 benomyl 저항성 벼키다리병균의 beta-tubulin 유전자 변이 분석
초록
국내에서 분리한 벼키다리병균(Fusarium fujikuroi)을 대상으로 benomyl에 대한 감수성, 중도저항성, 저항성을 나타내는 31개 균주를 확보하였다. 해당 균주들을 대상으로 benomyl의 작용점인 β-tubulin2 단백질을 암호화하는 유전자의 염기서열을 분석하고, 그 차이를 확인하였다. 감수성 균주와 비교하였을 때 benomyl 중도저항성 균주는 200번 아미노산이 TTC (Phenylalanine)에서 TAC (Tyrosine)로, 235번 아미노산이 GGC (Glysine)에서 GGT (Glysine)으로 치환되었음을 확인하였다. 저항성 균주의 경우, 167번과 235번의 아미노산 코돈이 각각 TTC (Phenylalanine)에서 TAC (Tyrosine), G G C (G lysine)에서 GGT (Glysine)으로 치환되었음을 확인하였다. 이러한 유전적 차이를 이용하여 benomyl 감수성, 중도저항성, 저항성 균주를 검출할 수 있는 특이 프라이머를 개발한 결과, 각각의 프라이머가 486 bp, 211 bp, 217 bp 크기의 밴드를 증폭하는 것을 확인하였다. 본 연구를 통해 우리나라에서 분리한 벼키다리병균의 benomyl 감수성, 중도저항성, 저항성 균주의 아미노산 코돈의 치환 부위를 처음으로 확인하였다. 또한 개발된 특이 프라이머를 활용한 PCR법에 의해 신속하고 정확한 benomyl 약제저항성균 스크리닝 방법을 구축하였다. 이를 통해 농업 현장에서 분리된 키다리병균의 약제저항성 발생에 대한 모니터링을 신속하게 수행할 수 있을 것으로 생각된다.
Abstract
In a previous study, 31 strains of sensitive, moderately resistant, and resistant to benomyl Fusarium fujikuroi were isolated from paddy fields in Korea. Analysis of the sequence of genes encoding β-tubulin protein, the mode of action point in response to benomyl, was performed on Korean isolates of F. fujikuroi to confirm differences in the sequences. The isolates that showed moderate resistance to benomyl had a mutation of the 200th amino acid codon from TTC (Phenylalanine) to TAC (Tyrosine), and the 235th amino acid codon from GGC (Glycine) to GGT (Glycine) compared to the benomyl sensitive isolates (F200Y, G235G). In the case of resistant isolates, replacement of amino acid codons 167 and 235 with TAC (Tyrosine) from TTC (Phenylalanine), and GGC (Glycine) to GGT (Glycine), respectively (F167Y, G235G), was confirmed. Based on these genetic differences, specific primers for detecting the benomyl sensitive, moderately resistant, and resistant isolates were designed, and the results confirmed that the products of the expected size (sensitive 486 bp, moderately resistant 211 bp, resistant 217 bp) were amplified only in target isolates. In this study, the substitution site of the amino acid codon was identified by conducting a comparison and analysis of the difference in the base sequence of benomyl sensitive, moderately resistant, and resistant isolates. In addition, by developing specific primers manufactured using the differences described above, a rapid and accurate method for screening benomyl resistant isolates of F. fujikuroi that will enable rapid monitoring for development of fungicide resistance in F. fujikuroi isolates from paddy fields in Korea was established.
Keywords:
benomyl resistance, Fusarium fujikuroi, beta-tubulin gene키워드:
베노밀 저항성, 벼키다리병균, 베타 튜불린 유전자서 론
벼 키다리병은 대표적인 종자전염성 곰팡이병으로 주로 Fusarium fujikuroi에 의해 발생하며 벼 못자리부터 본답 후기까지 발생한다. 유기수은제를 사용했던 1960년대에는 거의 문제되지 않았고, 유기수은제 사용이 금지된 이후에도 효과적인 종자소독제를 사용하여 국내에서 벼 키다리병은 문제되지 않았다(Park et al., 2003). 그러나 2000년대 초반부터 국내에서 발생이 증가하기 시작하였고, 2013년에는 본답 발병률이 약 31%로 크게 증가하는 등 벼의 문제 병해 중 하나로 손꼽히고 있다(Choi et al., 2015a).
1970년대까지 우리나라를 비롯한 일본, 중국에서는 벼 키다리병의 방제를 위해서 benomyl이나 thiophanate-methyl과 같은 benzimidazole계 살균제를 사용하였고, 이후 prochloraz 등 EBI계 살균제를 종자소독제로 사용하였다. 그러나 우리나라에서는 2010년대에 들어서면서 prochloraz, benomyl에 대한 약제저항성 벼키다리병균의 분포가 증가하고 있는 것으로 보고되었다(Choi et al., 2015a; Choi et al., 2015b). 일본의 경우, 벼 종자소독제로 benomyl를 반복적으로 사용한 결과, 벼 키다리병이 대발생하였고 그 원인이 benzimidazole계 살균제 저항성 병원균의 분포가 증가했기 때문임을 확인한 바 있다(Ogawa and Takeda 1990). Benzimidazole계 살균제는 침투이행성이 있는 살균제로 benomyl, carbendazim(MBC), thiabendazole 등이 이 계통에 속해있다. 특히, 식물병원 진균의 β-tubulin 단백질에 결합하여 미세소관형성을 저해함으로써 병원균의 세포분열을 억제하고, 그 생장을 억제시키는 기작으로 병을 방제하는 것으로 알려져 있다. Benzimidazole계 살균제에 대한 저항성 기작은 β-tubulin의 특정 아미노산이 다른 아미노산으로 치환되면서 살균제 성분이 β-tubulin 단백질에 결합하지 못하게 되어 나타나는 것으로 보고되었다(Yarden & Katan, 1993). Benzimidazole계 살균제는 다양한 식물 진균병의 방제에 활용되어 왔으며, 이에 따라 약제저항성에 대한 보고가 다수 있는데, 오이 흰가루병과 시클라멘 잿빛곰팡이병, 사과 검은별무늬병 등에서도 약제저항성 균주가 출현하여 약효가 감소되었다는 보고가 있다(Bollen and Scholten, 1971; Schroede and Provvidenti, 1969; Koenraadt et al., 1992). 한편, 맥류에 붉은곰팡이병을 일으키는 Fusarium graminearum과 여러 식물에 탄저병을 일으키는 Colletotrichum gloeosporioides 등을 대상으로 benzimidazole계 저항성 균주의 β-tubulin2(TUB2) 유전자 변이를 분석 한 결과, 6번, 50번, 167번, 198번, 200번 아미노산의 치환이 확인되어 보고되었는데, 이는 병원균이나 약제의 종류에 따라 다소 차이가 있다(Banno et al., 2008; Yarden and katan 1993; Kwak et al., 2017; Orbach et al., 1986; Mamika Minagawa 2021; Jung et al., 1990; Zhan and Huang, 2007; Romualdo Cabanas et al., 2009). 그러나 벼키다리병균의 경우, benzimidazole계 저항성 균주를 대상으로 한 유전자 분석은 전 세계적으로 거의 이루어진 바 없으며, 최근 중국의 장쑤성에서 분리한 벼키다리병균의 분석 결과, carbendazim 저항성 균주가 198번, 200번, 235번에서 변이가 있다고 보고되었다(Chen et al., 2014).
본 연구에서는 우리나라 논에서 분리한 벼키다리병균 중 Fusarium fujikuroi 균을 대상으로 benomyl에 대한 약제 반응을 조사하여 저항성, 중도저항성, 감수성으로 구분하고, 각 균주의 TUB2 유전자의 염기서열을 분석하여 유전자 변이 양상을 확인하였다. 또한, 점돌연변이가 일어난 부위를 특이적으로 검출할 수 있는 프라이머 세트를 개발하여 신속하고 정확하게 benomyl 저항성 벼키다리병균을 모니터링할 수 있는 방법을 확립하였다.
재료 및 방법
시험균주
2006~2007년, 2013~2014년에 우리나라 전국의 논 또는 못자리에서 키다리병이 발생한 시료를 채집하고, 병원균을 순수분리하여 국립농업과학원 작물보호과에 보존하고 있던 31개 균주를 선발하여 시험에 사용하였다(Table 1). 이전 연구에서 시험균주는 형태적, 배양적 특성과 TEF (translation elongation factor 1 alpha) 유전자 염기서열 분석에 의해 모두 Fusarium fujikuroi로 동정되었으며, 볍씨를 이용한 병원성 검정 방법을 통해 모두 벼에 키다리병을 일으키는 것으로 확인되었다(Choi et al., 2015a). 또한 한천희석법을 사용하여 benomyl에 대한 약제반응 조사를 실시하였고, EC50(effect concentration of 50%) 값과 최소억제농도(minimum inhibitory concentration, MIC) 값을 조사하여 저항성 기준을 설정하였다(Choi et al., 2015b). 즉, EC50 값이 2.4 μg/mL 이하인 경우는 감수성 균주로, EC50 값이 2.4 μg/mL 이하 이면서 MIC 값이 25 μg/mL 이상인 경우는 중도저항성, EC50 값이 2.4 μg/mL 이상이고, MIC 값이 25 μg/mL 이상인 경우를 저항성 균주로 구분하였다. 이 기준에 따라 본 연구에서 사용된 31개 균주는 각각 benomyl 감수성 13균주, 중도 저항성 2균주, 저항성 16균주로 확인되었다.
Genomic DNA 추출
시험균주의 TUB2 유전자를 분석하기 위하여 genomic DNA를 추출하였다(Ha et al., 2022). 각 균주를 감자한천액체배지(Potato dextrose broth, Difco)에 5일간 정치배양한 후, 멸균된 miracloth를 이용하여 균사체를 회수하고, 동결건조 후 마쇄하였다. Maxwell® RSC PureFood GMO and Authentication Kit (Promega, Madison, WI, USA)를 이용하여 마쇄된 균사체 파우더로부터 DNA를 분리하였다. 분리한 genomic DNA는 -20oC에 보관하여 실험에 사용하였다.
TUB2 유전자 증폭 및 염기서열 분석
시험균주의 TUB2 유전자를 증폭하기 위하여 beta2-F(TGC TGA TAC TCG CTG ACC), beta2-R(TTC TGC TTT GAG GGA CAT) 프라이머를 사용하여 PCR 증폭하였다(Zhang et al., 2015). PCR 반응액 조성은 10× buffer 5 μL, dNTP mixture 4 μL, 10 pmole의 forward primer와 reverse primer를 각각 2 μL, genomic DNA (33 ng/μL) 3 μL, Taq DNA polymerase (5 U/μL) 0.25 μL (TaKaRa Ex Taq)에 멸균수를 첨가하여 최종 50 μL의 PCR 반응액을 준비하였다. PCR 반응조건은 94oC에서 5분간 첫 변성단계를 거친 후, 94oC에서 1분, 54oC에서 30초, 72oC에서 90초를 35회 반복하였고, 마지막으로 72oC에서 10분간 반응시킨 후 종료하였다. PCR 증폭산물은 전기영동을 수행하여 유전자 증폭여부를 확인한 후, kit (Wizard® SV Gel and PCR clean-Up system)를 사용하여 정제하였고, Bionics (Seoul, Korea)에 의뢰하여 염기서열을 분석하였다. 분석된 TUB2 유전자 염기서열은 Edit sequence 프로그램을 이용하여 정리 및 alignment를 수행하였고, MEGA X (Kumar et al., 2018)에서 염기서열을 정렬한 후 비교 분석하였다.
PCR 분석에 의한 benomyl 저항성 벼키다리병균 스크리닝
TUB2 유전자 염기서열 분석 결과, 감수성, 중도저항성, 저항성 균주를 판단할 수 있는 점돌연변이 부위를 확인하였다. PCR을 통해 신속하고 정확하게 균주의 저항성 정도를 구분하기 위한 특이 프라이머를 만들고자 SNP (single nucleotide polymorphism) marker analysis 방법을 참고하여 Primer3 프로그램으로 후보 프라이머 세트의 염기서열을 선정하였다(Liu et al., 2012). 감수성 균주 3개, 중도저항성 균주 2개 및 저항성 균주 3개를 대상으로 후보 프라이머를 PCR 분석하여 저항성 여부를 구별할 수 있는 프라이머 세트를 최종 선발하였다(Table 3). 최종 선발된 프라이머 세트를 이용하여 시험 균주들의 저항성 정도를 스크리닝하였다. 31개 시험균주의 genomic DNA를 주형으로 PCR반응을 수행하였다. PCR 반응액 조성은 10× buffer 2 μL, dNTP mixture 2 μL, 5 pmole forward primer와 reverse primer를 각각 1 μL, genomic DNA (33 ng/μl) 3 μL, nTaq-Tenuto DNA polymerase (5 U/μL) 0.2 μL (nTaq-Tenuto, enzynomics)에 멸균수를 첨가하여 최종 20 μL의 PCR 반응액을 준비하였다. PCR 반응조건은 95oC에서 10분간 첫 변성단계를 거친 후, 95oC에서 30초, 58oC에서 30초, 72oC에서 25초를 30회 반복하였고, 마지막으로 72oC에서 4분간 반응시킨 후 종료하였다. PCR 증폭 산물은 1.5% 아가로스겔(Inclone Agarose LE)에 전기영동하여 증폭 여부 및 크기를 확인하였다.
결과 및 고찰
벼키다리병균의 TUB2 염기서열 및 변이 분석
이전 연구에서 31개 시험균주의 benomyl에 대한 약제반응을 조사한 결과, 13개 균주는 감수성, 2개 균주는 중도저항성, 16개 균주는 저항성인 것으로 나타났다(Table 1). 이들 균주를 대상으로 benomyl 살균제의 작용점인 β-tubulin2(TUB2) 유전자를 증폭하여 약 1,490 bp 크기의 염기서열을 확보하여 분석하였다. 그 결과, 167번, 200번, 235번 아미노산 코돈에서 일정한 점돌연변이를 확인할 수 있었다(Fig. 1). 한천희석법에 의해 저항성으로 구분한 균주들 중 2개 균주(CF322, CF379)를 제외한 14개 균주는 모두 167번과 235번의 아미노산 코돈이 각각 TTC (Phenylalanine)에서 TAC (Tyrosine), GGC (Glysine)에서 GGT (Glysine)로 치환된 것을 확인할 수 있었다(F167Y, G235G). 또한, 중도저항성으로 판단하였던 RF052, RF237 균주는 200번 아미노산의 염기서열이 TTC (Phenylalanine)에서 TAC (Tyrosine)로, 235번 코돈이 GGC (Glysine)에서 GGT (Glysine)로 치환되었다(F200Y, G235G). 그러나 한천희석법에 의해 저항성 균주로 분류되었던 CF322, CF379 균주는 중도저항성균주와 동일한 부위인 F200Y, G235G에서 변이가 일어난 것으로 나타났다. 감수성 균주들 중 특이한 것은 CF292와 CF350 균주의 235번째 아미노산 염기서열이 GGC에서 GGT로 변이가 있었다는 점이다. 이는 235번째 부위에서 비록 변이는 있었지만 같은 아미노산을 암호화하기 때문에 단백질 구조의 변화가 없어 약제에 대한 반응이 바뀌지 않은 것으로 생각되지만, 당진에서 분리한 CF292의 경우, MIC 값이 25 μg/mL 이상인 것으로 볼 때, 이들 균주는 중도저항성 또는 저항성으로 변이될 수 있을 것으로 추측되며, 이에 대한 추후 연구가 필요할 것으로 생각된다.
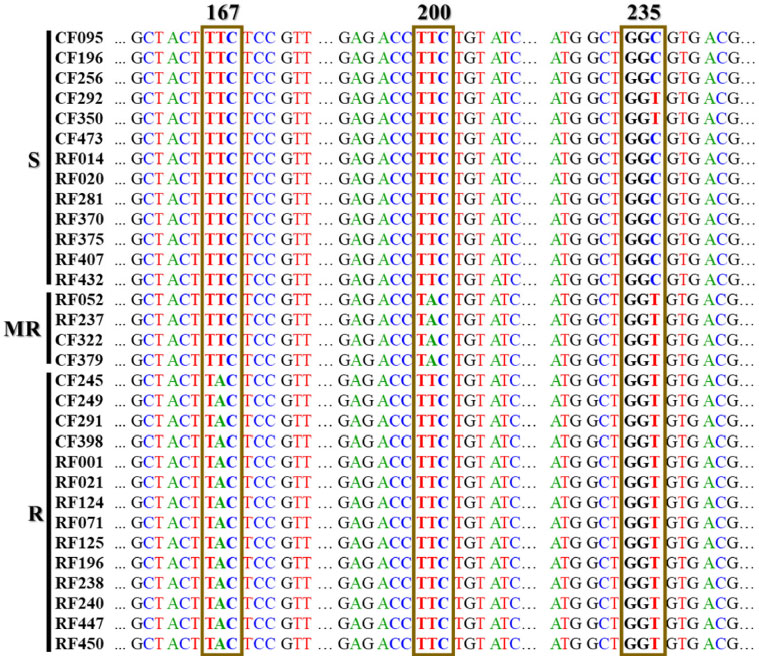
The mutation sites of the β-tubulin gene(TUB2) from benomyl sensitive, moderate resistant and resistant isolates of Fusarium fujikuroi collected from the paddy fields in Korea (S, benomyl sensitive; MR, benomyl moderate resistance; R, benomyl resistance).
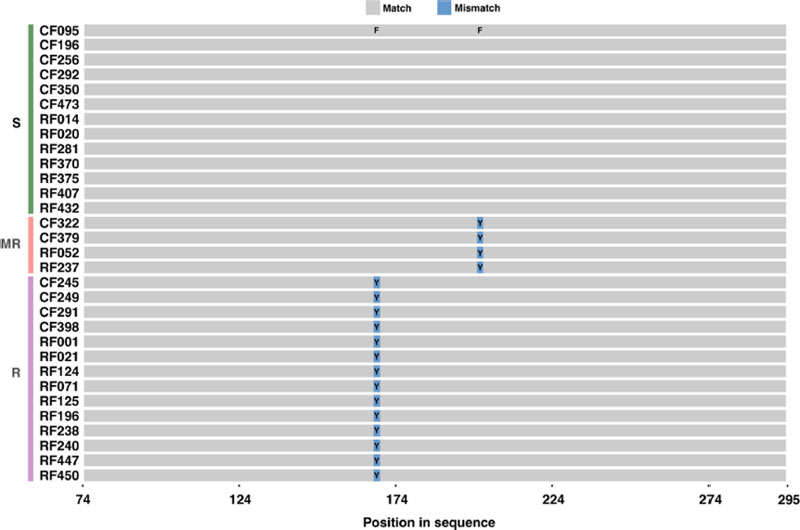
Comparison of amino acid sequence mutation sites among benomyl sensitive, mediate resistance, and resistance isolates (S, benomyl sensitive; MR, benomyl moderate resistance; R, benomyl resistance).
뿐만 아니라 한천희석법에 의해 감수성 분류된 CF292와 CF350균주는 235번 코돈에서 중도저항성, 저항성 균주와 동일한 변이인 G235G(GGC→GGT)가 확인되었다. 일반적으로 한천희석법에 의한 EC50 값과 MIC 값을 기준으로 한 감수성 기준은 연구자마다 다소 차이가 있다. 예를 들면, 우리나라에서는 F. fujikuroi 균주의 EC50 값이 2.4 μg/mL 이하이면서 MIC 값이 25 μg/mL 이상인 경우는 중도저항성, EC50 값이 2.4 μg/mL 이상이고, MIC 값이 25 μg/mL 이상인 경우를 저항성으로 정하였다(Choi et al., 2015b). 하지만, 중국에서 분리한 F. fujikuroi 균주는 EC50 값이 2.41 μg/mL이면서 MIC 값이 50~100 μg/mL는 중도저항성, EC50 값이 3.32 μg/mL이고, MIC 값이 100 μg/mL 이상인 경우를 저항성으로 구분하였다(Chen et al., 2014). 따라서 한천희석법에 의해 설정한 우리나라 벼키다리병균의 benomyl 저항성기준을 재설정할 필요가 있는 것으로 판단된다. 그 기준은 한천희석법에 의해 저항성 균주로 구분하였으나 TUB2 분석에 의해 중도저항성 균주로 분류되는 CF322, CF379균주의 EC50 값이 4.14 μg/mL이므로 EC50 값이 4.2 μg/mL 이상이고, MIC 값이 25 μg/mL 이상인 경우를 저항성으로 하는 것이 타당할 것으로 생각된다.
한편, 중국에서도 benzimidazole계 약제 중 하나인 carbendazim을 대상으로 논 포장에서 분리한 F. fujikuroi 균주의 TUB2 유전자를 분석하였는데, 생물검정을 통해 carbendazim 약제의 EC50과 MIC 값을 도출하여 감수성, 중도저항성, 저항성 균주를 분류하였고, 해당 균주의 TUB2 유전자염기서열의 변이를 확인하였다(Chen et al., 2014). 그 결과, 감수성 균주의 염기서열과 비교하였을 때 중도저항성 균주는 200번과 235번 코돈이 각각 TTC (Phenylalanine)에서 TAC (Tyrosine), GGC (Glysine)에서 GGT (Glysine)으로 치환되었고, 저항성 균주는 198번, 235번 코돈이 각각 GAG (Glutamic acid)에서 GTG (Valine), GGC (Glysine)에서 GGT (Glysine)으로 치환된 것으로 나타났다(Table 2). 이를 국내 균주의 변이와 비교하였을 때, 중도저항성 균의 경우 중국 균주와의 변이 부위와 염기서열의 차이가 없었다. 그러나 저항성 균주의 경우 공통적으로 235번 아미노산에 같은 염기서열로 변이가 발생하였지만, 국내 균주의 경우 167번 아미노산 염기서열이 변이가 발생한 반면, 중국 균주의 경우 198번 아미노산 염기서열에 변이가 발생하였다(Chen et al., 2014). 또한 일본에서 분리한 F. fujikuroi의 thiophanate-methyl 저항성 변이분석 결과, TUB2 유전자의 198번과 200번째 부위에서 각각 GAG (Glysine)가 AAG(Lysine), TTC (Phenylalanine)에서 TAC (Tyrosine)로 변이되었다(Li et al., 2022). 일반적으로 benzimidazole계 살균제 저항성의 경우, TUB2의 다양한 위치에서 유전자 치환이 보고되어 있으나, 198번째 아미노산이 가장 많다(Kwak et al., 2017). 본 연구에서 사용한 약제인 benomyl에 대한 저항성 부위 역시 Venturia inaequalis, Monilinia fructicola, Penicillium expansum 등 다양한 식물병원균의 TUB2 유전자의 198번째 아미노산인 것이 보고되었다(Koenraadt et al., 1992). 하지만 우리나라의 벼키다리병균은 198번이 아닌 167번 아미노산에서 변이가 나타났으며, 이는 같은 계열의 benzimidazole 약제이지만 benomyl과 carbendazim, thiophanate-methyl의 약제 성분이 다르기 때문에 변이부위에 차이가 나타났거나, 중국, 일본과 우리나라의 재배환경이나 균주 특성의 차이에 의한 것으로 생각된다. 한편, 또 다른 연구에서는 벼키다리병균과 동일한 Fusarium 속(genus)인 F.verticillioides의 경우 β-tubulin 1 유전자의 돌연변이로 인해 carbendazim 저항성이 나타난다고 보고되었는데(Yan and Dickman, 1996), 본 연구에서도 β-tubulin 1 유전자의 돌연변이를 확인하였으나 변이를 확인할 수 없었다(데이터 미제시).
우리나라 벼키다리병균의 benomyl 저항성균 스크리닝을 위한 특이 프라이머 개발
이미 우리나라에는 benomyl 저항성 F. fujikuroi가 전국포장에 다수 존재하는 것을 확인할 수 있었다. 특히 TUB2유전자의 특이적인 부위에 변이가 나타나므로 해서 저항성을 획득하는 것을 확인하였는데, 이는 한천희석법으로 분석한 EC50 값과 MIC 값의 감수성 기준 설정과 정확하게 일치하지 않는 것으로 나타났다. 따라서 신속하고 정확한 benomyl 저항성균 스크리닝을 위한 PCR방법에 활용할 특이 프라이머를 개발하였다. 저항성 균주의 경우, 167번과 235번의 아미노산 코돈이 각각 TTC에서 TAC로, GGC에서 GGT로 치환되었고(F167Y, G235G), 중도저항성 균주는 200번째 부위에서 TTC가 TAC로, 235번의 아미노산 코돈이 GGC에서 GGT로 치환되었다(F200Y, G235G). 이러한 점돌연변이를 특이적으로 증폭할 수 있는 특이 프라이머로서 감수성 균주를 검출할 수 있는 세트인 MBCSF1/R1, 중도저항성 균주를 검출할 수 있는 MBCMRF1/MBCRR1, 저항성 균주를 특이적으로 검출할 수 있는 MBCHRF1/MBCRR1를 선발하였다(Table 3). 선발된 프라이머 세트를 활용하여 PCR 증폭을 실시한 결과, 감수성 균주는 486 bp, 중도저항성 균주는 217 bp, 저항성 균주 211 bp 크기의 band를 확인할 수 있었고, 이 프라이머 세트를 이용하여 실험에 사용된 31개 균주를 PCR 분석한 결과, 감수성, 중도저항성, 저항성 균주를 구분할 수 있는 것으로 확인되었다(Fig. 3).
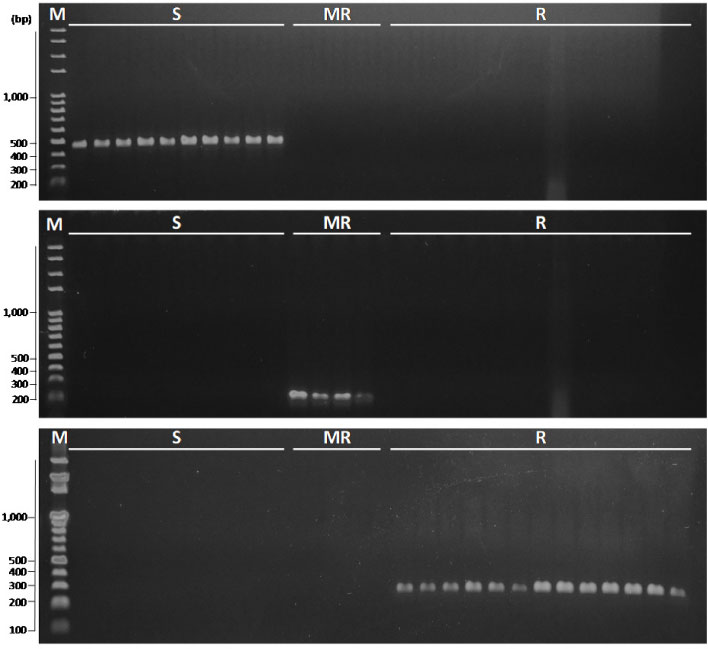
Gel electrophoresis result using specific primer sets (M, marker; S, benomyl sensitive; MR, benomyl moderate resistance; R, benomyl resistance).
본 연구를 통해 우리나라에 분포하는 benomyl 저항성 벼키다리병균의 TUB2 유전자 부위의 점돌연변이 발생 위치와 아미노산 치환에 대해 확인할 수 있었다. 특히 전 세계적으로 벼키다리병균인 F. fujikuroi의 bezimidazole계 살균제 저항성에 대한 보고는 중국과 일본을 제외하면 거의 없으며, 우리나라에서도 benomyl 저항성 균주의 TUB2 변이 분석에 대한 보고는 처음이다. 또한, 특이 프라이머를 활용하여 PCR 분석에 의해 저항성 여부를 스크리닝할 수 있는 방법을 구축함으로써 약제저항성 발생에 대한 모니터링을 신속하게 수행할 수 있도록 하였다.
Acknowledgments
본 연구는 국립농업과학원 기관고유 연구사업(과제번호: PJ015003)에 의해 수행되었습니다. 이에 감사드립니다.
이해상충관계
저자는 이해상충관계가 없음을 선언합니다
References
-
Bollen GJ, Scholten G, 1971. Acquired resistance to benomyl and some other systemic fungicides in a strain of Botrytis cinerea in cyclamen. Neth. J. Plant Pathol. 77:83-90.
[https://doi.org/10.1007/BF01981496]

-
Chen Z, Gao T, Liang S, Liu K, Zhou M, et al., 2014. Molecular mechanism of resistance of Fusarium fujikuroi to benzimidazole fungicides. FEMS Microbiol. Lett. 357(1):77-84.
[https://doi.org/10.1111/1574-6968.12504]

-
Choi H-W, Lee YH, Hong SK, Lee YK, Nam YJ, et al., 2015a. Monitoring for the resistance to prochloraz of Fusarium species causing bakanae disease in Korea. Kor. J. Mycol. 43(2):112-117. (In Korean)
[https://doi.org/10.4489/KJM.2015.43.4.260]

-
Choi H-W, Lee YH, Hong SK, Lee YK, Lee JG, et al., 2015b. Baseline sensitivity and monitoring for the resistance to benomyl of Fusarium species causing bakanae disease in Korea. Kor. J. Mycol. 43(4):260-266. (In Korean)
[https://doi.org/10.4489/KJM.2015.43.4.260]

- Ha MS, Ryu H, Hong SK, Ju HJ, Choi H-W. 2022. Occurrence of root rot caused by Fusarium fujikuroi on Adzuki bean in Korea. Kor. J. Mycol. 50(4):319-329.
-
Jung MK, Oakley BR, 1990. Identification of an amino acid substitution in the benA, β‐tubulin gene of Aspergillus nidulans that confers thiabendazole resistance and benomyl supersensitivity. Cell Motil Cytoskeleton. 17(2):87-94.
[https://doi.org/10.1002/cm.970170204]

-
Koenraadt H, Somerville SC, Jones AL, 1992. Characterization of mutations in the beta-tubulin gene of benomyl-resistant field strains of Venturia inaequalis and other plant pathogenic fungi. Phytopathology. 82(11):1348-1354.
[https://doi.org/10.1094/Phyto-82-1348]

-
Kumar S, Stecher G, Li M, Knyaz C, Tamura K. 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms. Mol Biol Evol. 35(6):1547-1549.
[https://doi.org/10.1093/molbev/msy096]

-
Kwak Y, Min J, Song J, Kim M, Lee H, et al., 2017. Relationship of resistance to benzimidazole fungicides with mutation of β-tubulin gene in Venturia nashicola. Res. Plant. Dis. 23(2):150-158. (In Korean)
[https://doi.org/10.5423/RPD.2017.23.2.150]

-
Li FJ, Komura R, Nakashima C, Shimizu M, Kageyama K, et al., 2022. Molecular diagnosis of thiophanate-methylresistant strains of Fusarium fujikuroi in Japan. Plant Dis. 106(2):634-640.
[https://doi.org/10.1094/PDIS-07-21-1501-RE]

-
Liu J, Huang S, Sun M, Liu S, Liu Y, et al., 2012. An improved allele-specific PCR primer design method for SNP marker analysis and its application. Plant Methods. 8:34.
[https://doi.org/10.1186/1746-4811-8-34]

-
Ogawa K, Takeda S, 1990. Population of benomyl resistant Rice “Bakanae” fungus in paddy fields. Jpn J Phytopathol. 56(2):247-249.
[https://doi.org/10.3186/jjphytopath.56.247]

- Park HG, Shin HR, Lee Y, Kim SW, Kwon OD, et al., 2003. Influence of water temperature, soaking period, and chemical dosage on Bakanae disease of rice (Gibberella fujikuroi) in seed disinfection. Korean J. Pestic. Sci. 7(3):216-222. (In Korean)
- Schroede WT, Provvidenti R, 1969. Resistance to benomyl in powdery mildew of cucurbits. Plant Dis Rep. 53(4):271-275.
-
Yan K, Dickman MB, 1996. Isolation of a β-tubulin gene from Fusarium moniliforme that confers cold-sensitive benomyl resistance. Appl. Environ. Microbiol. 62:3053-3056.
[https://doi.org/10.1128/aem.62.8.3053-3056.1996]

-
Zhang Z, Chen Z, Hou Y, Duan Y, Wang J, et al., 2015. PIRAPCR for detection of Fusarium fujikuroi genotypes with carbendazim-resistance alleles. Plant Dis. 99(9):1241-1246.
[https://doi.org/10.1094/PDIS-01-15-0009-RE]

Hyunjoo Ryu, Department of Agro-food Safety and Crop Protection, National Institute of Agricultural Sciences, RDA, Researcher, https://orcid.org/0000-0001-7998-6754
Jae Geum Lee, Department of Agro-food Safety and Crop Protection, National Institute of Agricultural Sciences, RDA, Researcher
Eun Hye Kang, Department of Agro-food Safety and Crop Protection, National Institute of Agricultural Sciences, RDA, Researcher
Sung Kee Hong, Department of Agro-food Safety and Crop Protection, National Institute of Agricultural Sciences, RDA, Doctor of Philosophy, https://orcid.org/0000-03-4204-8371
Hyo-Won Choi, Extension Service Bureau, Disaster Management Division, RDA, Doctor of Philosophy, https://orcid.org/0000-0002-3496-3855

