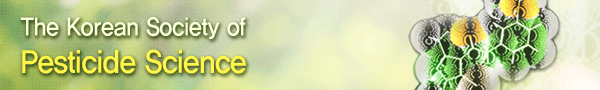
복합기능성 Bacillus sp. GH1-13 균주의 특징
초록
바실러스는 토양 및 반추동물의 소화기관과 같은 다양한 곳에서 분리되고 있으며, 작물의 생육 촉진과 병 방제를 위한 미생물 재제로서 널리 사용되고 있다. 바실러스 GH1-13 균주는 전남 완도의 간척지 논에서 분리되었으며, 16S rRNA 유전자와 gyrB 유전자를 이용하여 계통유전학적으로 분석한 결과 Bacillus velezensis인 것으로 동정되었다. GH1-13 균주의 특성을 분석한 결과 생육촉진에 관련된 IAA를 생성할 뿐 아니라 벼 뿌리의 생육을 촉진하는 것을 확인하였다. 또한 벼의 주요 병원균의 생육을 억제할 뿐 아니라 작물의 병원균인 다양한 곰팡이의 생육을 저해하였다. 더불어 식물병원 진균 및 세균의 생육 억제와 밀접하게 관련된 것으로 판단되는 bacillomycin, bacilycin, fengycin, iturin, surfactin을 생성하는 생합성유전자를 보유한 것으로 확인되었다. 본 연구는 GH1-13균주가 작물의 생육촉진과 병 방제를 동시에 해결할 수 있는 강력한 복합기능성 미생물제로의 가능성이 있음을 보여주었다.
Abstract
Several microorganisms in particular Bacillus subtilis group have been isolated from diverse places such as soils and the gastrointestinal tract of ruminants etc., and used as boicontrol agent against various plant pathogens and utilized as plant growth promoting agents. Among them, Bacillus is well known as one of the most useful bacteria for biocontrol and plant growth promotion. Bacterium GH1-13 was isolated from a reclaimed paddy field in Wando Island and identified as Bacillus velezensis using phylogenetic analysis on the basis of 16S rRNA and gyrB gene. It was confirmed that GH1-13 produced indole acetic acid (IAA) associated with promoted growth of rice root. GH1-13 showed characteristics of antagonization against the main pathogen of rice as well as diverse pathogenic fungi. GH1-13 had biosynthetic genes, bacillomycin, bacilycin, fengycin, iturin, and surfactin which are considered to be associated closely with inhibition of growth of pathogenic fungi and bacteria. This study showed that GH1-13 could be used as a multifunctional agent for biocontrol and growth promotion of crop.
Keywords:
Bacillus, biocontrol, growth promotion, multifunction, secondary metabolites키워드:
바실러스, 생육촉진, 방제, 복합기능, 이차대사물질서 론
농업에서 작물의 병을 방제하고 생산성을 증대시키기 위해 다량으로 사용되고 있는 화학농약과 비료를 대체할 수 있는 환경친화적인 소재로서 미생물에 대한 연구가 많이 이루어지고 있다(Avis et al., 2008; Santoyo et al., 2012). 최근 다양한 미생물이 시설하우스, 밭, 과수원 등의 농업 현장에서 활용되고 있으며, 시군 농업기술센터에서 다양한 미생물을 배양하여 농가에 공급하고 있다. 많은 시군 농업기술센터에서 배양하여 보급하는 미생물은 주로 바실러스(Bacillus), 유산균(Lactic acid bacteria), 효모(Saccharomyces), 광합성균(Photosynthetic bacteria) 등이며 몇몇 곰팡이(Aspergillus)와 방선균(Streptomyces), 슈도모나스(Pseudomonas) 등도 활용되고 있다. 이들 중 바실러스는 작물의 생육촉진, 병 저항성 증진, 병 방제뿐 아니라 해충 방제, 악취 저감, 사료제조 등의 목적으로 다양한 농업 현장에 사용되고 있다(Velivelli et al., 2014).
바실러스는 non-ribosomal peptide synthetase (NRPS)와 polyketide synthase (PKS)를 이용하여 관련된 여러 항생제뿐만 아니라 다양한 휘발성 물질 등을 생산하여 식물의 생장을 촉진하고 병 저항성을 강화시켜 병 방제에도 효과가 있는 것으로 알려져 있다(Ongena and Jacques, 2008; Insam and Seewald, 2010; Raaijmakers et al., 2010; Chowdhury et al., 2015). 오이(Cucumis sativus)균핵병을 방제할 수 있는 Bacillus sp. M27 균주와 벼(Oryza sativa)의 생육촉진 및 병을 억제할 수 있는 Bacillus vallismortis EXTN-1 등이 일반적으로 알려져 있다(Park et al., 2006; Lee et al., 2013). 인삼 밭 토양으로부터 미생물을 분리하여 인삼(Panax ginseng) 뿌리썩음병균에 길항력이 있는 바실러스 균주를 선발하여 동정하고, 항진균 활성에 대한 특성 조사 또한 이루어진 바 있다(Kim et al., 2012).
최근 친환경 농산물의 수요 증대와 저농약 농산물의 폐지 등으로 작물 병 방제 및 생육촉진 등의 활성을 가진 미생물제의 수요가 증대하고 있으며, 특히 내생포자를 생성하여 제제화 하기가 용이한 바실러스는 다양하게 미생물제재로서 개발되어 상용화되고 있다(Nicholson, 2002; Cawoy et al., 2011). 이와 같이 친환경 미생물제의 개발을 위해 작물의 근권 및 주변 토양 뿐 아니라 다양한 환경으로부터 유용한 미생물을 분리하여 특성을 밝히고 현장에 적용하는 연구가 수행되고는 있으나 여전히 생육촉진과 병 방제를 동시에 수행하는 미생물에 대한 연구는 미흡한 실정이다.
본 연구에서는 간척지 작물의 병 방제와 더불어 생육촉진 등에 활용할 수 있는 유용미생물을 분리하기 위해 대표 간척지 3지역(전남 고흥, 충남 당진, 전남 완도)의 논으로부터 시료를 채취하여 염에 내성을 가지며, 작물의 주요 병원균에 대한 길항 활성이 우수한 균주를 선발하고 특성을 분석하였으며, 16S rRNA와 gyrB 유전자를 이용하여 동정하였다.
재료 및 방법
균주 분리
내염성을 가지며, 작물 생육 촉진 및 병 방제에 활용할 수 있는 세균을 분리하기 위해 2015년 5월 간척지 논(전남 고흥, 충남 당진, 전남 완도 일대)에서 약 1 cm의 표토를 제거하고 오우거(auger)를 이용하여 15 cm까지의 토양을 채취하였다. 각 시료 1 g을 9 ml의 멸균수에 현탁하여 3% NaCl을 포함한 R2A 배지에 현탁액 0.1 ml를 도말한 후 28oC 항온기에서 3일간 배양하였다. R2A배지의 조성은 Yeast extract 0.5 g, Proteose peptone 0.5 g, Casamino acids 0.5 g, Glucose 0.5 g, Soluble starch 0.5 g, Na-pyruvate 0.3 g, K2HPO4 0.3 g, MgSO4·7H2O 0.05 g, Agar 15.0 g, distilled water 1,000 mL이며 pH 7.0으로 조절하였다. 다양한 세균을 분리하기 위해 무작위로 다른 형태나 색깔을 보이는 집락을 순수분리 하였다.
분리 균주의 항균활성 검정
분리균주의 항균활성 검정은 대치배양법을 이용하였다. 병원성 곰팡이인 경우 페트리 접시에서 병원균과 분리균을 대치배양하여 식물병원균의 생장 저해능을 검정하였으며, 세균인 경우 병원균을 배지에 도말한 후 분리균을 그 위에 접종하여 3-5일간 배양 한 후 분리균에 의한 식물병원균의 생장 저해능을 검정하였다. 사용된 균주는 벼에 해를 입히는 주요 병원균 및 다양한 작물에서 해를 입히는 주요 병원균을 사용하였으며, 이들 균주는 농촌진흥청 농업유전자원센터(Korean Agricultural Culture Collection (KACC), www.genebank.go.kr)에서 분양 받아 사용하였다(Table 1).
선발 균주의 생육촉진 활성 검정
선발 균주의 생육촉진 활성을 검정하기 위해 L-tryptophan을 첨가한 배지(0.1% wt/v)와 무첨가 R2A broth배지에 각각 균주 접종 후 30oC, 150 rpm에서 약 48시간 동안 균주를 배양하였다. 균주 밀도가 1 × 109 cfu/ml 수준으로 배양 완료된 배약액 1 ml를 4oC, 4000 rpm, 15분간 원심분리하여 균체를 침강시킨 후 상등액만 회수하였다. 배양상등액 0.5 ml과 Salkowski 시약 1 ml (35% HClO4 50 mL, 0.5 M FeCl3 1 mL)을 혼합하여 상온에서 30분간 반응시킨 후 분광광도계(Infinite 200 PRO multimode reader, Tecan, Austria)를 이용하여 530 nm에서 흡광도를 측정하여 균주의 IAA 생성능을 확인하였다. 이때 표준곡선은 Sigma사의 IAA (Indole-3-Acetic Acid)를 0, 10, 30, 50, 100 mg/L 농도로 제조하여 R2A broth 배지에 첨가 후 시료와 동일한 조건하에서 반응 후 측정하였다(Naqqash et al., 2016).
선발 균주의 작물 생육 촉진 활성을 검정하기 위해 tryptic soy broth (TSB) 배지에서 40시간 배양(약 1 × 109 cfu/ml)한 후, 균체를 13,000 rpm, 15분간 원심분리하고 멸균증류수로 두 번 세척하였다. TSB배지의 조성은 Tryptone 17.0 g, Soytone 3.0 g, Glucose 2.5 g, Sodium Chloride 5.0 g, Dipotassium hydrogen phosphate 2.5 g, distilled water 1,000 mL이며, pH 7.3으로 조절하였다. 수확한 선발 균주의 균체는 0.03 M MgSO4 용액에 1 × 106 cfu/ml의 농도로 현탁하였다. 활성 검정 대상 작물인 벼, 토마토(Solanum lycopersicum), 오이 종자는 1차 소독액(70% 에탄올, 0.1% Triton X-100)에서 1분간 침지한 후, 2차 소독액(10% 차아염소산(NaOCl), 0.1% Triton X-100)에 15분간 침지하였다. 침지된 종자를 멸균수로 5번 세척하여 준비하였다. 각 종자를 선발균주의 현탁액(A600≒0.5)에 3시간 침지한 후 풍건하고 생육검정 파우치(PhytoTC, USA)에 종자를 올려놓고 28oC 항온기(16시간 광조건)에서 10일간 배양한 후 줄기의 생육 길이와 무게, 뿌리의 발달 개수 및 뿌리털의 발달 개수를 육안으로 관측 후 계수하였다.
선발 균주의 이차대사물질 유전자의 검출
바실러스의 병원균에 대한 길항 활성을 나타내는NRPS와 PKS의 합성 관련 유전자를 검출하기 위해 특이 프라이머를 활용하여 PCR을 실시하였다(Athukorala et al. 2009) (Table 2). PCR은 PCR 반응 혼합액(2X TOPsimple DyeMIXTenuto; Enzymonics Co., Korea)을 이용하였으며, 1 uL의 genomic DNA (30 ng)와 10 pmol의 프라이머를 혼합하여 20 uL를 최종 부피로 하여 실행하였다. PCR은 초기 변성반응(initial denaturation; 95oC, 3분) 후, 증폭반응(변성 95oC 30초, 결합 55oC 45초, 신장 72oC 1분)을 30회 반복하였으며, 최종 신장반응은 72oC에서 5분간 실시하였다. 증폭한 PCR 산물은 전기영동 하여 존재여부 및 크기를 확인하였다.
선발 균주의 동정
GH1-13 균주의 동정을 위해 16S rRNA 유전자와 Bacillus spp.의 분류에 유용한 gyrB 유전자(DNA gyrase유전자)의 염기서열을 분석하였다(Wang et al., 2007). GH1-13 균주는 TSB에 접종하여 28oC에서 24시간 이상 배양한 후 원심분리하여 균체를 수확하였다. QIAamp DNA mini kit (Qiagen, USA)를 이용하여 genomic DNA를 추출하였다. 16S rRNA 유전자는 범용 프라이머인 27F와 1492R을 사용하였고, gyrB 유전자는 범용 프라이머 UP-1과 UP-2r을 사용하여 PCR한 후 각각 유전자의 증폭산물을 얻었다(Yamamoto and Harayama, 1995; Song et al., 2004). 16S rRNA 유전자의 염기서열 분석은3개의 프라이머(800R, 518F, 984F)를 이용하고 gyrB 유전자는 두 개의 염기서열 분석용 프라이머(UP-1S, UP-2Sr)를 이용하여 분석하도록 제노텍(대전, 한국)에 의뢰하였다(Table 1). 얻어진 염기서열은 Seqman (DNASTAR, USA)을 이용하여 직접 오류를 검정하고 연결하였다. 각 유전자의 비교분석을 위해서 16S rRNA 유전자의 염기서열은 EzTaxon-e 서버를 통해서 얻었으며, gyrB 유전자의 염기서열은 Genbank (www.ncbi.nlm.nih/genbank)를 통하여 얻었다. 계통도의 작성을 위해 MEGA 5.0 프로그램의 Clustal W 프로그램으로 염기서열을 정렬하였으며, Neighbor-joining 알고리즘을 사용하고 1,000회 반복 bootstraping을 통해 계통도의 신뢰성을 확인하였다(Tamura et al., 2011).
통계분석
통계분석은 SAS 프로그램(버전 9.2)을 이용하여 실시하였으며, t-검정을 통해 유의 수준 5%에서 균주 처리에 따른 생육증진 효과를 비교하였다.
결과 및 고찰
균주 분리 및 항균활성 검정
간척지로 알려진 전남 고흥군, 충남 당진군, 전남 완도군 일대의 논 토양으로부터 내염성이 있는 균주 총 137균주를 분리하였다. 분리한 균주의 45%는 Bacillus 속에 속한 균주였으며, 이들 중 대부분(80%)은 B. aryabhattai와 B. marisflavi, B. stratosphericus, B. megaterium 등인 것으로 분석되었다.
대치배양법을 이용하여 분리균주의 벼의 주요 병원균에 대한 항균활성 검정을 수행한 결과, 벼의 주요 병원균에 대한 생장 억제 활성을 나타내는 다양한 세균을 확인하였으며 이들 중 가장 강한 활성을 보이는 바실러스 균주(GH1-13)를 선발하였다(Fig. 1). GH1-13 균주는 벼의 주요 곰팡이병원균인 Fusarium fujikuroi (키다리병균), Pyricularia grisea(도열병균), Bipolaris oryzae (깨씨무늬병균), Rhizoctonia solani (잎집무늬마름병균)에 대한 강한 활성을 보였으며, 벼의 주요 세균 병원균인 Xanthmonas oryzae (흰잎마름병균), Acidovorax avenae (세균성줄무늬병균), Burkholderia glumae(알마름병균)에 대해서도 생육 억제능을 보였다(Fig. 1). 또한 GH1-13은 다양한 작물에 병을 일으키는 것으로 알려진 Cercospora capsici (갈색무늬병균), Phytophthora capcisi(역병균), Sclerotinia sclerotiorum (균핵병균), Colletotrichum acutatum (탄저병균), Alternaria alternata (검은곰팡이병균), Botrytis cinerea (잿빛곰팡이병균), Fusarium oxysporum (뿌리썩음병균), Streptomyces scabiei (더뎅이병균), Clavibacter michiganensis (궤양병균), Pectobacterium carotoborum (무름병균)에 대한 생육 억제 활성을 보였다(Table 1). 인삼 뿌리썩음병의 원인균인 Cylindrocarpon destructans (뿌리썩음병균), Sclerotinia nivalis (뿌리썩음병균)에 대한 강한 생육 억제활성을 보였다(Table 1). 이 결과를 통해 GH1-13균주가 벼를 포함한 다양한 작물의 병해 방제를 위한 미생물제로서의 잠재적 가치가 큰 것으로 평가된다. 향후 실제 농가에서 생물 검정을 바탕으로 GH1-13 균주의 현장 적용성 평가가 수행 되어야 할 것으로 판단되며, 이를 바탕으로 토착 세균을 이용한 우수한 미생물제로서의 활용 가능성 확인 및 다양한 병원균에 대한 생육 억제 활성 기작에 대한 연구가 진행되어야 할 것으로 여겨진다.
Growth inhibition of pathogenic fungi and bacteria of rice (Oryza sativa L.) by Bacillus sp. GH1-13. (A) Gibberella moniliformis KACC 44022 and Fusarium fujikuroi, (B) Bipolaris oryzae KACC 41025, (C) Pyricularia grisea KACC 40435, (D) Rhizoctonia solani KACC 40123, (E) Xanthomonas oryzae KACC 10331, (F) Acidovorax avenae KACC 10162, (G) Burkholderia glumae KACC 10138.
선발 균주의 생육촉진 활성 검정
GH1-13 균주의 IAA 생성능을 분석한 결과, tryptophan이 배지에 추가되었을 때의 IAA생성량(31.4 μg/mL)은 없을 때의 생성량(6.3 μg/mL)보다 약 5배 정도 증가되었으며 타 바실러스 균주와 비교하여 큰 차이를 나타내지는 않았다(Fig. 2). 이는 GH1-13균주가 tryptophan을 전구체로 사용하여 IAA를 만드는 tryptophan 의존형 경로를 가진 세균이라는 것을 보여준다(Idris et al., 2007).
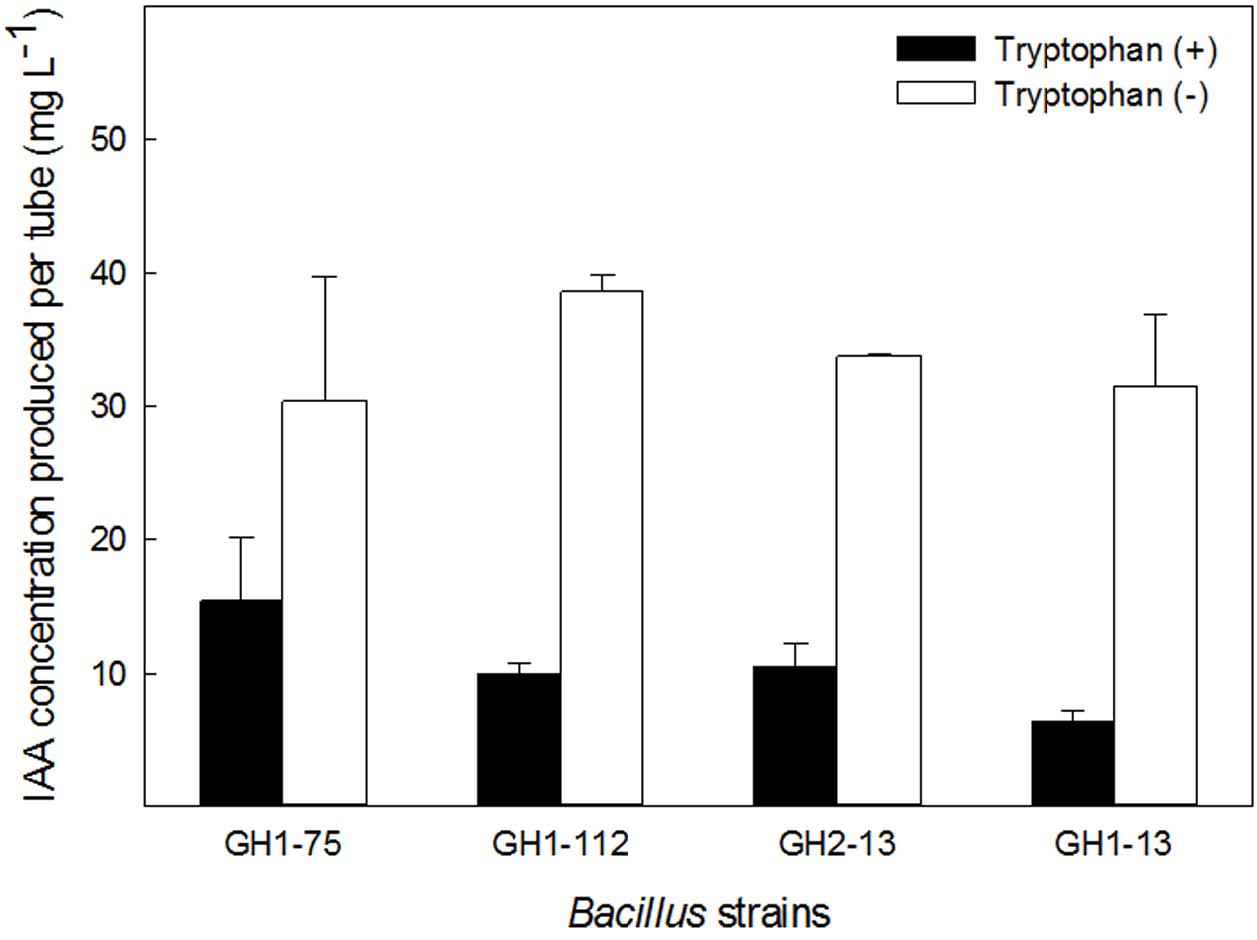
IAA production potential from several Bacillus stains in the presence or absence of tryptophan in R2A media. Bars indicate standard deviations (n=3).
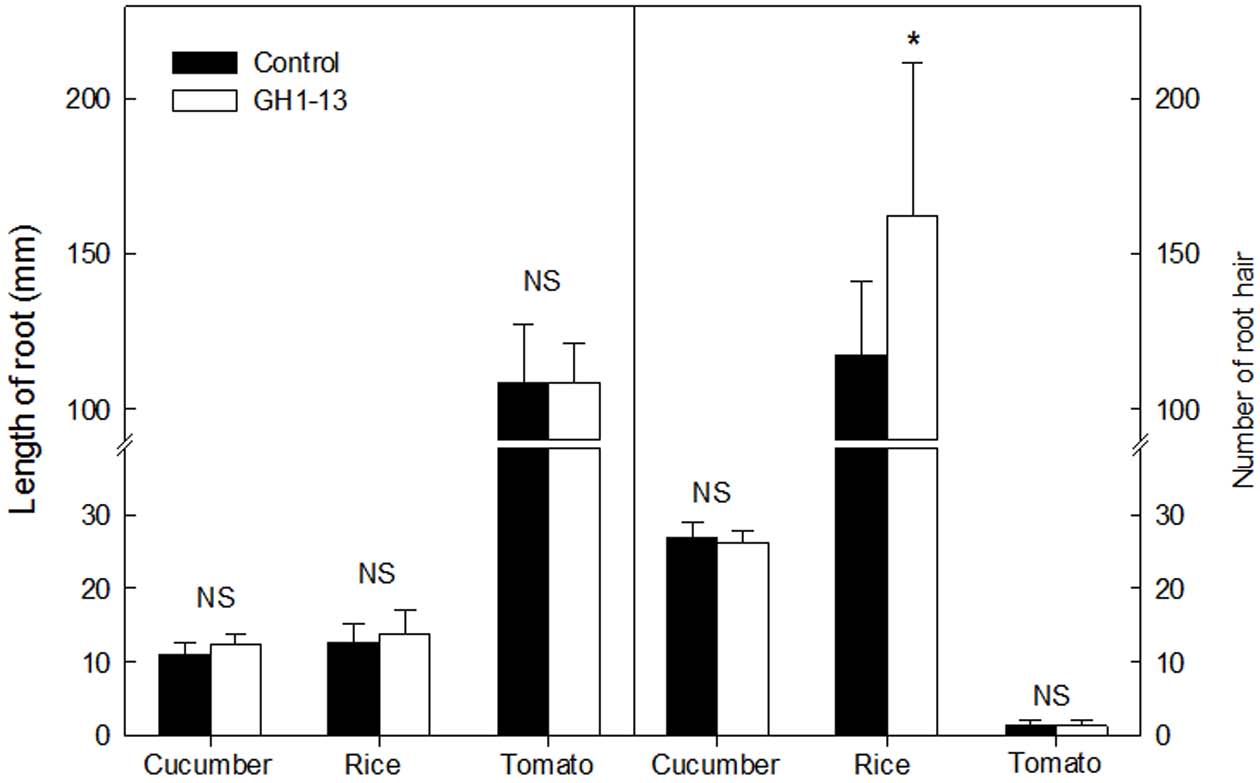
작물생육촉진 활성을 검정하기 위하여 GH1-13 균주로 침지 처리한 벼, 토마토, 오이의 종자를 생육검정 파우치를 이용하여 검정하였다(Fig. 3). 토마토와 오이에 대한 생육촉진 효과는 통계적으로 유의한 차이가 없는 것으로 나타났다. 벼의 경우 뿌리의 길이에 대한 생육촉진 효과는 통계적인 차이는 없었으나 뿌리털의 개수에는 통계적인 유의성을 보였다(p<0.05, n=9)(Fig. 3). 이러한 결과는 GH1-13 균주가 식물 생육촉진과 관련 있는IAA같은 물질 분비를 통해 벼뿌리털의 발달을 촉진시켜 벼의 생육을 촉진하는 역할을 하는 것으로 판단된다(Richardson et al., 2009). 이러한 균주의 특징을 이용하여 벼의 직파에 활용한다면 벼의 생장 초기에 뿌리의 발달에 도움을 줌으로써 입모율을 향상시켜 벼의 생육 및 생산성을 개선할 수 있는 미생물제의 개발이 가능할 것으로 여겨진다.
선발 균주의 길항활성 관련 유전자의 검출
GH1-13 균주는 벼의 주요 병원균뿐만 아니라 채소 등의 작물에서 발생하는 많은 병원균, 특히 진균에 의한 병원균에 강한 길항 활성을 보였다(Fig. 1). 이는 바실러스 균주들이 다양한 생합성 유전자를 이용하여 2차 대사산물을 생산하는 것처럼 GH1-13균주가 생합성 유전자를 가지고 관련된 항생 물질을 생산할 것으로 판단하여 기존에 알려진 생합성 유전자, bacD (bacilycin), bmyA (bacillomycin D), fenD(fengycin), ituA (iturin A), srfA (surfactin), and zwiA(zwittermicin A)를 검출할 수 있는 특이 프라이머를 이용하여 PCR을 수행하였다(Ramarathnam et al., 2007; Athukorala et al., 2009; Kim et al., 2012)(Fig. 4). GH1-13 균주는 bacillomycin D (875 bp), bacilycin (749 bp), fengycin (964 bp), iturin (647 bp), surfactin (441 bp)을 생성하는 유전자를 지니는 것으로 조사되었으나, zwittermicin 생성 관련 유전자는 검출되지 않았다. Kim et al. (2012)에 의해 보고된 Bacillus amyloliquifaciens GR4-5 균주와 비교할 때 fengycin 생합성 유전자가 추가적으로 존재함을 확인하였으며 더불어 세균에 길항력을 보이는 bacilycin 생합성 유전자도 확인하였다. 이러한 결과는 GH1-13균주가 다양한 작물의 진균 및 세균성 병원균의 생육을 억제할 수 있는 강력한 미생물제로 개발될 수 있음을 보여준다.
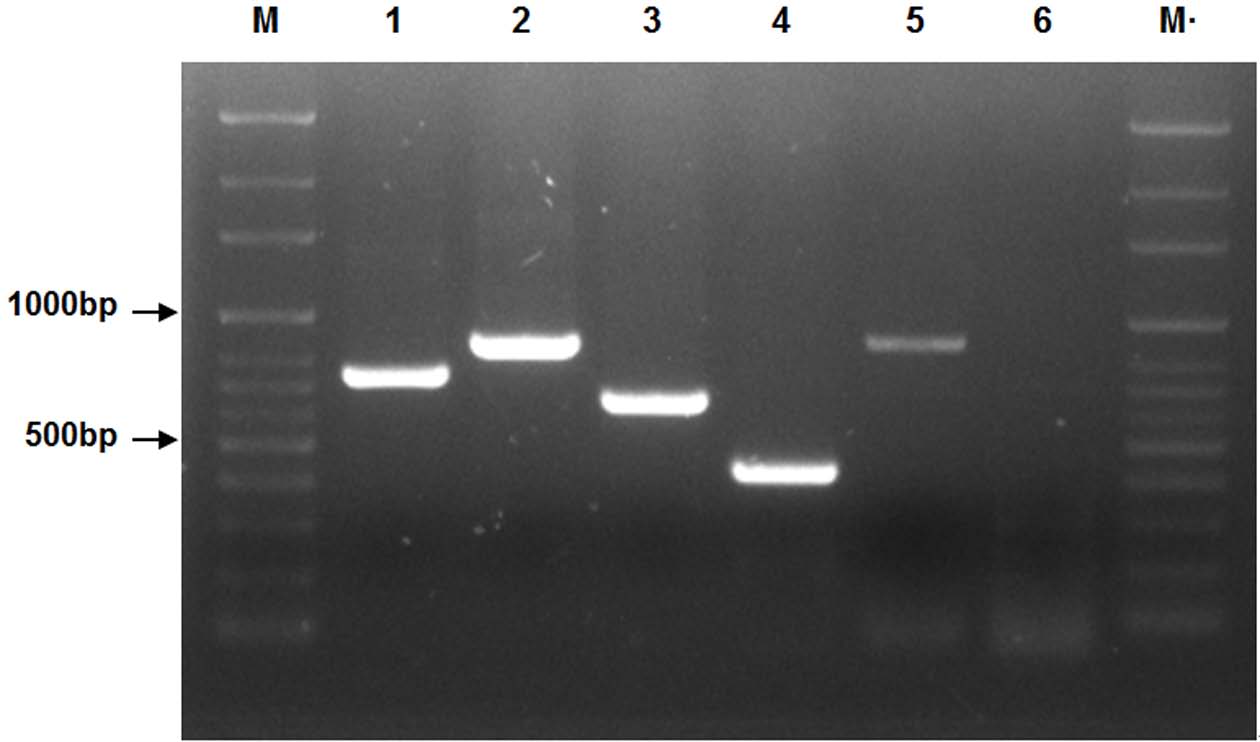
Detection of biosynthetic genes of Bacillus sp. GH1-13 by PCR amplification. M. marker (100 bp ladder), 1; bacilysin (bacD, 749 bp), 2; bacillomycin (BmyA, 875 bp), 3; iturin (ituA, 647 bp), 4; surfactin (srfA, 441 bp), 5; fengycin (fenD, 964 bp), 6; zwittermicin (zwiA, 779 bp). PCR products were detected on 1.5% agarose gel.
선발 균주의 동정
GH1-13 균주의 동정을 위해 16S rRNA와 gyr B 유전자를 바탕으로 MEGA5 프로그램을 이용하여 분자유전학적인 계통도를 작성하였다(Fig. 5). 16S rRNA 유전자를 바탕으로 작성한 계통도에서 GH1-13 균주는 Bacillus methylotrophicus KACC 13105(T) 균주와 높은 상동성(100%)을 보였다(Fig. 5(a)). 계통도의 신뢰성을 확보하기 위한 Bootstrap 값도 69%로 비교적 높은 값을 나타내어 Bacillus methylotrophicus인 것으로 추정되었다. gyrB 유전자를 바탕으로 작성한 계통도에서 GH1-13 균주는 Bacillus velezensis BCRC 17467(T) 균주와 Bacillus methylotrophicus KACC 13105(T) 균주에 같은 높은 상동성(98.6%)을 보였다(Fig. 5(b)). 최근 계통유전체학을 바탕으로 두 종(B. methylotrophicus와 B. velezensis)과 더불어 B. amyloliquefaciens subsp. plantarum과 B. oryzicola를 포함하여 모두 B. velezensis로 분류되어져야 한다고 보고되었다(Dunlap et al., 2016). 16S rRNA 유전자와 gyrB 유전자의 분석 결과와 Dunlap et al. (2016)의 보고에 따라 GH1-13 균주의 분류학적인 위치는 B. velezensis로 판단된다. 결론적으로 본 연구에서는 B. velezensis GH1-13균주가 작물의 생육촉진과 병 방제를 동시에 해결할 수 있는 강력한 복합기능성 미생물제로의 가능성이 있음을 보여주었다. 그러나 B. velezensis GH1-13균주 처리가 실제 현장에서 항균활성에 미치는 영향에 대한 보다 정밀한 평가가 요구되며, 현장 적용성 평가를 위한 후속 연구가 수행되어져야 할 것으로 판단된다.
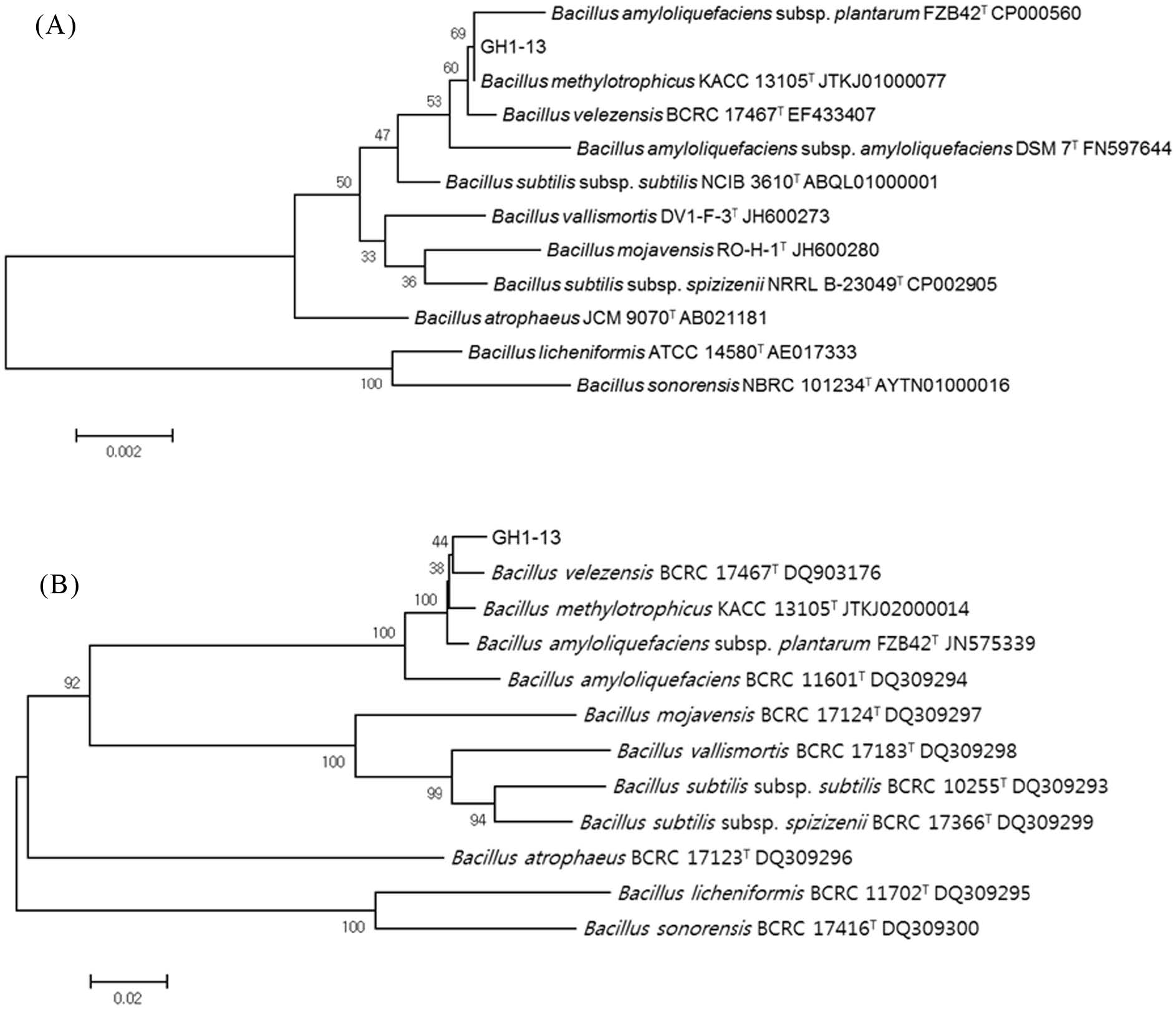
Neighbor-joining phylogenetic tree based on partial 16S rRNA (A) and gyrB (B) gene sequences (1,416 bp and 1,065 bp, respectively) of Bacillus strains. Bootstrap values are indicated at branch points. Strain or culture collection and accession numbers are indicated next to species name. T means type strain.
Acknowledgments
본 연구는 농촌진흥청 공동연구사업(과제번호: 010825)의 지원에 의해 수행되었습니다.
References
- Ahn, J. H., B. C. Kim, B. Y. Kim, S . J. Kim, J. Song, S. W. Kwon, and H. Y. Weon, (2014), Paenibacillus cucumis sp, nov. Isolated from Greenhouse Soil. J Microbiol, 52(6), p460-464.
-
Athukorala, S. N. P., W. G. D. Fernando, and K. Y. Rashid, (2009), Identification of antifungal antibiotics of Bacillus species isolated from different microhabitats using polymerase chain reaction and maldi-tof mass spectrometry, Can. J. Microbiol, 55(9), p1021-1032.
[https://doi.org/10.1139/W09-067]

-
Avis, T. J., V. Gravel, H. Antoun, and R. J. Tweddell, (2008), Multifaceted beneficial effects of rhizosphere microorganisms on plant health and productivity, Soil Biol. Biochem, 40(7), p1733-1740.
[https://doi.org/10.1016/j.soilbio.2008.02.013]

-
Cawoy, H., W. Bettiol, P. Fickers, and M. Ongena, (2011), Bacillus-based biological control of plant diseases, p273-302, In D.M. Stoytcheva (ed.), Pesticides in the modern world - pesticides use and management, InTech.
[https://doi.org/10.5772/17184]

-
Chowdhury, P. S., A. Hartmann, X. Gao, and R. Borriss, (2015), Biocontrol mechanism by root-associated Bacillus amyloliquefaciens FZB42 - a review, Front. Microbiol, 6(780), p1-11.
[https://doi.org/10.3389/fmicb.2015.00780]

- Dunlap, C. A., S. J. Kim, S. W. Kwon, and A. P. Rooney, (2016), Bacillus velezensis is not a later heterotypic synonym of Bacillus amyloliquefaciens; Bacillus methylotrophicus, Bacillus amyloliquefaciens subsp, Plantarum and ‘Bacillus oryzicola’ are later heterotypic synonyms of Bacillus velezensis based on phylogenomics. Int. J. Syst. Evol. Microbiol, 66(3), p1212-1217.
-
Fukushima, M., K. Kakinuma, and R. Kawaguchi, (2002), Phylogenetic analysis of Salmonella, Shigella, and Escherichiacoli strains on the basis of the gyrB gene sequence, J. Clin. Microbiol, 40(8), p2779-2785.
[https://doi.org/10.1128/JCM.40.8.2779-2785.2002]

-
Idris, E. E., D. J. Iglesias, M. Talon, and R. Borriss, (2007), Tryptophan-dependent production of indole-3-acetic acid (iaa) affects level of plant growth promotion by Bacillus amyloliquefaciens FZB42, Mol. Plant-Microbe Interact, 20(6), p619-626.
[https://doi.org/10.1094/MPMI-20-6-0619]

- Insam, H., and M. S. A. Seewald, (2010), Volatile organic compounds (vocs) in soils, Biol. Fertil. Soils, 46(3), p199-213.
-
Kim, B. Y., J. H. Ahn, H. Y. Weon, J. Song, S. I. Kim, and W. G. Kim, (2012), Isolation and characterization of Bacillus species possessing antifungal activity against ginseng root rot pathogens, Korean J. Pestic. Sci, 16(4), p357-363.
[https://doi.org/10.7585/kjps.2012.16.4.357]

-
Lee, S. Y., H. Y. Weon, J. J. Kim, J. H. Han, and W. G. Kim, (2013), Control effect of the mixture of Bacillus amyloliquefaciens M27 and plant extract against cucumber powdery mildew, Korean J. Pestic. Sci, 17(4), p435-439.
[https://doi.org/10.7585/kjps.2013.17.4.435]

-
Naqqash, T., S. Hameed, A. Imran, M. K. Hanif, A. Majeed, and J. D. van Elsas, (2016), Differential response of potato toward inoculation with taxonomically diverse plant growth promoting rhizobacteria, Front. Plant Sci, 7(1), p144.
[https://doi.org/10.3389/fpls.2016.00144]

- Nicholson, L. W., (2002), Roles of Bacillus endospores in the environment, Cell. Mol. Life Sci, 59(3), p410-416.
-
Ongena, M., and P. Jacques, (2008), Bacillus lipopeptides: Versatile weapons for plant disease biocontrol, Trends Microbiol, 16(3), p115-125.
[https://doi.org/10.1016/j.tim.2007.12.009]

-
Park, K., D. Paul, and W. H. Yeh, (2006), Bacillus vallismortis extn-1-mediated growth promotion and disease suppression in rice, Plant Pathol. J, 22(3), p278-282.
[https://doi.org/10.5423/PPJ.2006.22.3.278]

-
Raaijmakers, J. M., I. De Bruijn, O. Nybroe, and M. Ongena, (2010), Natural functions of lipopeptides from Bacillus and Pseudomonas: More than surfactants and antibiotics, Fems Microbiol. Rev, 34(6), p1037-1062.
[https://doi.org/10.1111/j.1574-6976.2010.00221.x]

-
Ramarathnam, R., S. Bo, Y. Chen, W. G. D. Fernando, G. Xuewen, and T. de Kievit, (2007), Molecular and biochemical detection of fengycin- and bacillomycin d-producing Bacillus spp., antagonistic to fungal pathogens of canola and wheat, Can. J. Microbiol, 53(7), p901-911.
[https://doi.org/10.1139/W07-049]

- Richardson, A. E., J. M. Barea, A. M. McNeill, and C. Prigent-Combaret, (2009), Acquisition of phosphorus and nitrogen in the rhizosphere and plant growth promotion by microorganisms, Plant Soil, 321(1), p305-339.
-
Santoyo, G., M. d. C. Orozco-Mosqueda, and M. Govindappa, (2012), Mechanisms of biocontrol and plant growthpromoting activity in soil bacterial species of Bacillus and Pseudomonas: A review, Biocontrol Sci. Technol, 22(8), p855-872.
[https://doi.org/10.1080/09583157.2012.694413]

-
Song, J., S. C. Lee, J. W. Kang, H. J. Baek, and J. W. Suh, (2004), Phylogenetic analysis of streptomyces spp, Isolated from potato scab lesions in korea on the basis of 16s rrna gene and 16s-23s rdna internally transcribed spacer sequences. Int. J. Syst. Evol. Microbiol, 54(1), p203-209.
[https://doi.org/10.1099/ijs.0.02624-0]

-
Tamura, K., D. Peterson, N. Peterson, G. Stecher, M. Nei, and S. Kumar, (2011), Mega 5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods, Mol. Biol. Evol, 28(10), p2731-2739.
[https://doi.org/10.1093/molbev/msr121]

-
Velivelli, S. L. S., P. De, Vos, P. Kromann, S. Declerck, and B. D. Prestwich, (2014), Biological control agents: From field to market, problems, and challenges, Trends Biotechnol, 32(10), p493-496.
[https://doi.org/10.1016/j.tibtech.2014.07.002]

-
Wang, L. T., F. L. Lee, C. J. Tai, and H. Kasai, (2007), Comparison of gyrB gene sequences, 16s rRNA gene sequences and DNA-DNA hybridization in the Bacillus subtilis group, Int. J. Syst. Evol. Microbiol, 57(8), p1846-1850.
[https://doi.org/10.1099/ijs.0.64685-0]

- Yamamoto, S., and S. Harayama, (1995), PCR amplification and direct sequencing of gyrB genes with universal primers and their application to the detection and taxonomic analysis of Pseudomonas putida strains, Appl. Environ. Microbiol, 61(3), p1104-1109.