살균제 디페노코나졸 분해 세균 분리 및 특성 분석
초록
트리아졸계 살균제는 세계적으로 사용량이 가장 많은 살균제 중 하나로 토양 반감기가 상대적으로 길다. 본 연구에서는 전국 과수원 토양으로부터 트리아졸계 살균제 테부코나졸, 플루퀸코나졸, 디페노코나졸을 분해할 수 있는 세균의 순수 분리를 시도하였다. 농화배양 과정에서 세 종의 살균제 중 디페노코나졸만이 100% 분해되었으며 이 배양액에서 디페노코나졸을 분해하는 세균 균주들을 순수분리하였다. rep-PCR 밴드 패턴 비교를 통해 이 균주들은 모두 동일 균주임을 확인하였으며 이 중 C8-2 균주만을 이후 연구에 사용하였다. 이 균주는 16S rRNA 유전자 염기서열에 기반하여 Sphingomonas 속 C8-2 균주로 동정되었으며 최소배지에서 100 mg/L의 디페노코나졸을 24시간 내에 분자량 296의 물질로 전환하였다. 이 분해산물은 토양 세균 및 곰팡이에 대한 저해 효과가 디페노코나졸에 비해 현저히 감소하여 향후 토양 및 농작물의 디페노코나졸 무독화에 C8-2 균주를 사용할 수 있을 것으로 기대한다.
Abstract
Triazole fungicides occupy an important portion in the global fungicide market and are relatively persistent in soil compared to the other fungicides, suggesting possible adverse effects of the fungicides on human health and environment. In this study, we tried to isolate microorganisms from orchard soils, which can decompose the triazole fungicides, tebuconazole, fluquinconazole, and difenoconazole. Only difenoconazole was completely degraded in the enrichment culture, from which several difenoconazole-degrading bacteria were isolated. They showed the same rep-PCR pattern thus only one strain, C8-2, was further studied. The strain was identified as Sphingomonas sp. C8-2 based on its 16S rRNA gene sequence and decomposed 100 mg/L of difenoconazole in a minimum medium to an unknown metabolite with a molecular weight of 296 within 24 hours. The inhibition effect of the metabolite against representative soil microorganisms significantly decreased compared to that of difenoconazole thus the bacterial strain is expected to be used for the detoxification of difenoconazole in soil and crop.
Keywords:
Bacteria, degradation, difenoconazole, triazole fungicide키워드:
세균, 생분해, 디페노코나졸, 트리아졸계 살균제서 론
환경에서 농약의 분해는 주로 미생물, 특히 세균에 의해 일어난다는 것이 일반적인 통설이다(Fenner et al., 2013). 농약은 적용 대상에 따라 살충제, 제초제, 살균제로 나눌 수 있으며 이 중 살균제를 분해하는 미생물에 대한 연구는 살충제나 제초제에 비해 매우 적은데 이는 살균제의 급성독성이 상대적으로 낮기 때문으로 생각된다. 살균제 중 트리아졸(triazole)계 살균제는 3개의 질소원자를 갖는 트리아졸기를 함유하는 침투이행성의 살균제로 예방과 치료 효과를 모두 가지며 곰팡이 세포막 형성에 필수적인 에르고스테롤의 생합성을 저해하여 살균효과를 나타낸다(Bromilow et al., 1999a; Sehnem et al., 2010). 각종 원예 및 밭작물의 흰가루병, 잿빛곰팡이병, 낙엽병 등의 방제에 사용되며 세계적으로 가장 소비량이 많은 살균제 계통 중 하나로 2020년까지 꾸준히 사용될 것으로 예측되고 있다(Research and markets, 2014).
최근 연구에서 트리아졸계 살균제는 수중 생물에 대한 독성(Mu et al., 2016) 뿐만 아니라 내분비계 장애물질로서 인간을 비롯한 포유류의 성적 발달에 영향을 미칠 수 있음이 보고되었다(Taxvig et al., 2007; Zhou et al., 2016). 국내에서 생산 및 수입되고 있는 트리아졸계 살균제로는 테부코나졸(Tebuconaozle), 디페노코나졸(Difenoconazole), 헥사코나졸(Hexaconazole), 플루퀸코나졸(Fluquinconazole) 등 17종이 있으며(KCPA, 2016) 트리아졸계 살균제는 일반적으로 토양에서 잔류기간이 상대적으로 긴 것으로 보고되어(Bromilow et al., 1999a,b; Kim et al., 2003; Badawi et al., 2016) 토양 및 농작물에 오랜 기간 남아 환경에 영향을 미칠 가능성이 크다. 따라서 이를 무독화할 수 있는 경제적이고 안전한 방법이 요구된다.
본 연구에서는 전국에서 채취한 과수원 토양으로부터 트리아졸계 살균제를 분해할 수 있는 미생물을 순수 분리하고 그 분해 특성을 구명하며 그 분해산물이 토양미생물에 미치는 영향을 분석하고자 하였다.
재료 및 방법
수입량 및 생산량이 많고 상대적으로 토양반감기가 긴 테부코나졸(597일), 플루퀸코나졸(350일), 디페노코나졸(318일) (www.pesticideinfo.org)을 선발하여 실험에 사용하였는데, 테부코나졸은 바이엘 크롭사이언스로부터, 플루퀸코나졸과 디페노코나졸은 아그로텍으로부터 각각 원제를 제공받아 사용하였다.
경기, 강원, 충청, 경상, 전라 지역의 22개 과수원에서 채취한 토양을 2 mm 체로 거른 후 100 g을 취하였다. 각각의 살균제 원제를 100 mg/mL의 농도로 아세톤에 용해시킨 후, 100 mg/kg soil의 농도가 되도록 토양에 처리하였다. 질소와 인을 황산암모늄과 인산염의 형태로 각각 5 mg N/kg soil과 0.5 mg P/kg soil의 농도로 되도록 토양에 첨가한 후 주기적으로 혼합하면서 1개월 동안 실온에서 관리하였다. 토양 1 g을 취하여 살균제 100 mg/L를 포함하는 최소배지 5 mL에 첨가 후 28oC에서 150 rpm으로 진탕 배양하면서 1개월 간격으로 살균제 농도를 측정하였다. 최소배지의 조성은 다음과 같다(1 L 당 첨가량): Na2HPO4·12H2O 3.58 g, KH2PO4 1.361 g, (NH4)2SO4 0.3 g, MgSO4·7H2O 0.05 g, CaCl2·H2O 0.0058 g, yeast extract 0.01 g, trace minerals 10 mL, vitamin solution 10 mL. Trace mineral과 vitamin solution은 DSMZ medium 141에 따라 조제하였다(www.dsmz.de). 살균제 농도는 Shimadzu SCL-10Avp HPLC system (Shimadzu, Japan)을 이용하여 조사하였는데 컬럼은 YMC-Triart C18, 이동상은 아세토니트릴:물=7:3, 유량은 0.7 mL/min이었으며 photodiode array detector를 이용하여 200 nm에서 분석하였다. 시료는 아세토니트릴로 1/20로 희석하고 0.2 μm 필터로 여과 후 분석에 사용하였다. 살균제의 감소가 확인되면 0.5 mL를 취하여 동일 배지 5 mL에 접종하고 동일 조건에서 배양하면서 수시로 살균제의 감소를 확인하였다. 살균제의 감소가 확인되면 이 배양액을 R2A agar (Difco, USA) 배지에 도말하고 28oC에서 배양하였다. 형성된 콜로니를 살균제 100 mg/L를 포함하는 최소배지에 접종 후 배양하여 살균제 분해 여부를 판정하였다.
결과 및 고찰
세 종의 살균제 중 디페노코나졸만 100% 분해되었으며 다른 살균제는 분해가 관찰되지 않았다. 이는 알려진 토양반감기와 상관이 있는 것으로 판단된다. 디페노코나졸 분해가 확인된 5개의 서로 다른 배양액으로부터(충북 충주, 전남 보성, 경기 이천, 경남 창원, 강원 춘천에서 채취한 토양시료 이용) 디페노코나졸을 분해할 수 있는 5균주를 순수분리 하였으며 (GTG)5 프라이머를 이용한 rep-PCR (Jarocki et al., 2016) 분석을 통해 이들이 모두 동일 균주임을 확인하였다(Fig. 1). 이 중 C8-2 균주만을 선택하여 이후 연구에 사용하였으며 16S rRNA 유전자 염기서열 분석을 통해 Sphingomonas 속으로 동정하였다(Fig. 2).
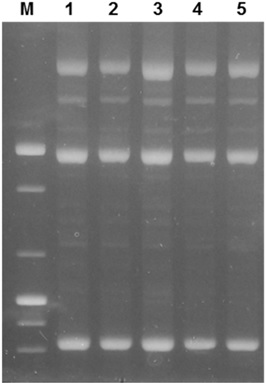
rep-PCR band patterns of difenoconazole-degrading bacteria isolated in this study. M, 100 bp size marker; 1, strain KW2222; 2, strain C11; 3, stain I10; 4, stain B7; 5, stain C8-2.
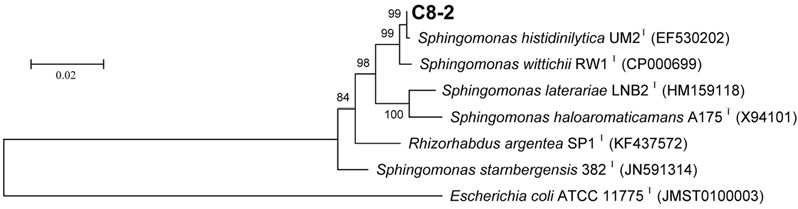
Neighbor-joining tree based on the 16S rRNA genes of the difenoconazole-degrading strain C8-2 and related type strains of the genus Sphingomonas. E. coli was used as an outgroup.
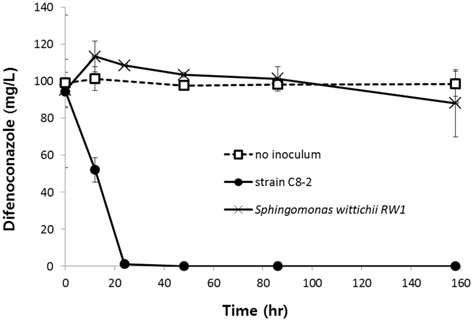
C8-2 균주의 디페노코나졸 분해 특성을 구명하기 위하여 nutrient agar (Oxoid, USA) 배지에 28oC로 5일간 배양 후 디페노코나졸 100 mg/L가 포함된 최소배지에 흡광도(OD600)가 0.1이 되도록 접종하였다. 접종한 배지를 28oC, 150 rpm에서 진탕 배양하면서 시간에 따른 디페노코나졸 농도를 조사하였다. 비교 균주로는 C8-2 균주에 대한 16S rRNA 유전자 염기서열 유사도가 99.5%이면서 다양한 유기합성화합물을 분해하는 것으로 알려진 Sphingomonas wittichii RW1(=KCTC 42168) (Miller et al., 2010)을 비교 균주로 사용하였다.
S. wittichii의 경우 배양 160시간 동안 디페노코나졸이 유의하게 감소하지 않은 반면 C8-2 균주는 24시간 이내에 검출한계 미만으로 감소하였다(Fig. 3).
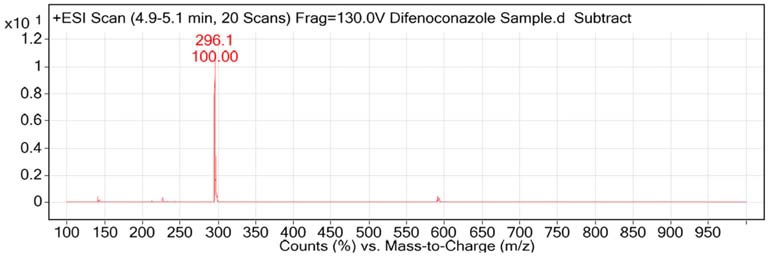
C8-2 균주 배양액의 크로마토그램에서 디페노코나졸 외에 머무름 시간 3분대에 새로운 피크가 생성된 것을 확인할 수 있었다(Fig. 4). 이 피크의 면적은 디페노코나졸 농도와 역의 상관 관계를 보였으며(Fig. 5) 이를 통해 디페노코나졸의 분해 산물인 것으로 추정되었다. 이 피크는 배양 160시간까지 잔존하여 C8-2 균주가 이 물질을 이용하지 못하는 것으로 판단된다. 이 3분대의 피크는 알려진 디페노코나졸 분해산물인 1,2,4-트리아졸이나 CGA-205375(1-[2-chloro-4-(4-chloro-phenoxy)-phenyl]-2-(1,2,4-triazol)-1-yl-ethanol) 의 피크와는 일치하지 않았다(Fig. 4). 이 물질의 분자량을 텐덤질량분석기를 이용하여 분석하였다. 분석은 전북대학교 공동실험실습관에서 수행하였으며 Agilent 1200 series HPLC와 6410 triple-quadrupole mass spectrometer를 이용하였다. 이동상은 0.1% 포름산과 0.1% 포름산을 포함한 아세토니트릴 용액을 사용하여 ESI positive 모드에서 분석하였다. 컬럼은 Pheomenex Kinetex C18 (2.1 × 50 mm, 2.6 μm I.D.)을 사용하였으며 컬럼온도는 30도, 흐름속도는 0.5 mL/min, 주입부피는 5 μL였다. 분석 결과 3분대 피크를 형성한 물질은 분자량 296임을 확인하였다(Fig. 6).
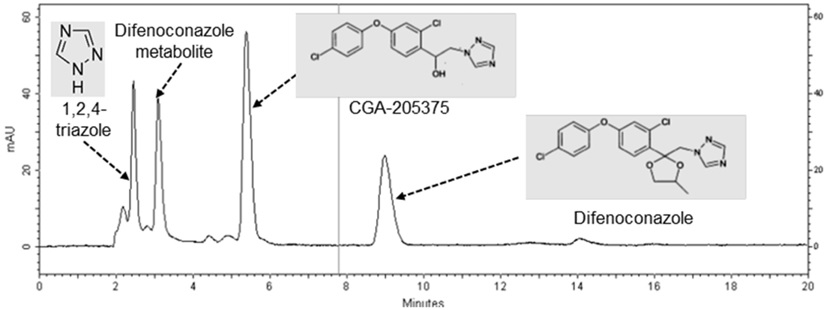
HPLC chromatogram of 1/10 dilution of the 20-day culture medium of strain C8-2 initially containing 100 mg/L of difenoconazole, to which 2.5 mg/L (the final concentration) of each of difenoconazole, CGA-205375, and 1,2,4-triazole was added.
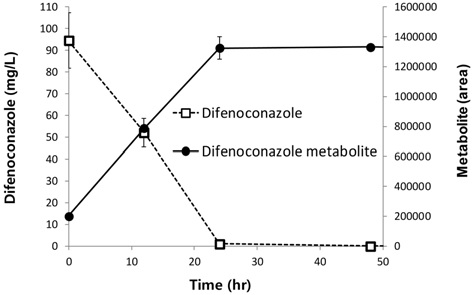
Variations of difenoconazole and a putative difenoconazole metabolite in the culture medium of strain C8-2 initially containing 100 mg/L of difenoconazole
토양세균인 Pseudomonas putida KACC 10266, Streptomyces graminisoli KACC 16472, Bacillus subtilis KACC 10854와 토양곰팡이인 Penicillium roqueforti KACC 47196를 농촌진흥청 농업미생물자원센터(KACC)에서 분양받아 디페노코나졸과 C8-2 균주에 의한 디페노코나졸 분해산물이 각각의 토양미생물의 생장에 미치는 영향을 조사하였다.
세균의 경우 디페노코나졸 0, 1, 5, 10 mg/L가 첨가된 R2A 액체 배지에 각 세균의 콜로니를 접종하고 28oC, 150 rpm에서 진탕배양하면서 시간에 따른 흡광도(OD600)를 측정했고, P. roqueforti KACC 47196의 경우 디페토코나졸이 0, 1, 5, 10 mg/L가 첨가된 Potato dextrose agar 배지(Oxoid, USA)에 접종한 후 28oC에서 배양하면서 균사의 성장을 육안으로 관찰하였다.
C8-2 균주에 의한 디페노코나졸 분해산물이 토양미생물에 미치는 영향 분석을 위해 먼저 C8-2 균주를 디페노코나졸 100 mg/L가 포함된 최소배지에 접종하고 28oC에서 20일간 진탕배양 후 0.45 μm 공극의 필터로 여과한 액을 1배(1×), 0.5배(0.5×), 0.1배(0.1×), 0.05배(0.05×), 0.01배(0.01×x), 0배(0×) 희석한 액에 세균의 경우 R2A를, 곰팡이의 경우 Potato dextrose agar를 정량대로 첨가하였다. 이 배지에 세균 또는 곰팡이 균사를 접종하고 세균의 경우 28oC, 150 rpm으로 진탕 배양하면서 시간에 따른 흡광도(OD600)를 측정했고 곰팡이의 경우 균사의 성장을 육안으로 관찰하였다.
결 론
실험결과 토양세균인 B. subtilis와 S. graminisoli의 성장은 디페노코나졸 농도가 1 mg/L에서 10 mg/L로 증가함에 따라 점차 저해되었고(Fig. 7A) 토양 곰팡이인 P. roqueforti의 성장은 디페노코나졸 농도 1 mg/L 이상에서 완전히 억제되었다(Fig. 8A). 반면 C8-2 균주의 분해산물은 토양세균인 B. subtilis와 S. graminisoli, 토양곰팡이인 P. roqueforti의 성장에 대한 저해효과가 현저히 감소함(Fig. 7B, 8B)을 확인할 수 있었다.
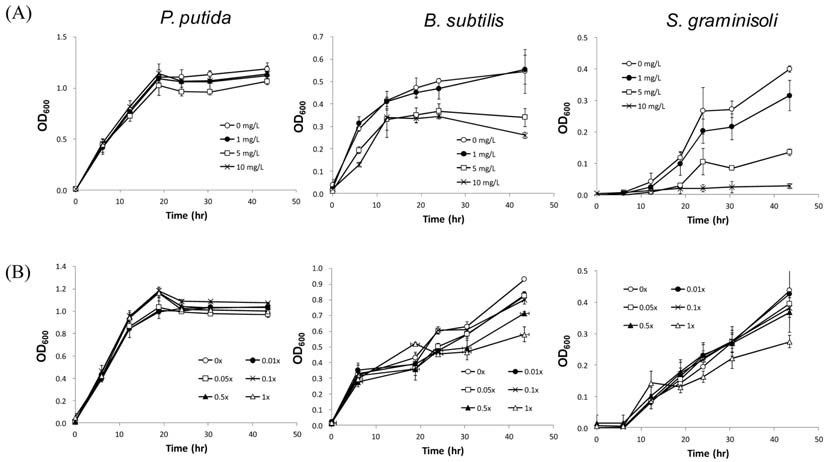
Effects of difenoconazole (A) and the putative difenoconazole metabolite (B) on the soil bacteria Pseudomonas putida, Bacillus subtilis, and Streptomyces graminisoli. Values are means ± standard deviations, n=3.
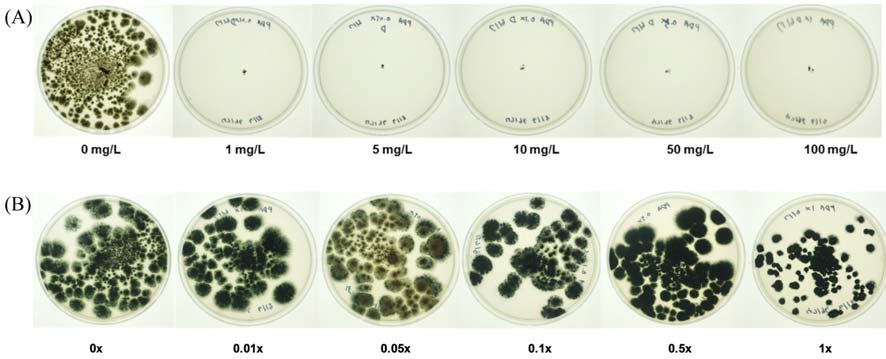
Effects of difenoconazole (A) and the putative difenoconazole metabolite (B) on the soil fungus Penicillium roqueforti. Only one of the triplicates for each treatment is presented.
이상의 결과로부터 C8-2 균주는 디페노코나졸을 빠르게 분자량 296의 물질로 전환하였으며 이 물질은 원 물질에 비해 토양 미생물에 대한 영향이 현저히 감소하였다. 따라서 향후 이 균주를 디페노코나졸 오염 토양 및 농산물의 정화에 이용할 수 있을 것으로 기대한다.
Acknowledgments
본 논문은 농촌진흥청 국립농업과학원 농업과학기술 연구개발사업(과제번호: PJ01094902)의 지원으로 수행된 결과이며 연구비 지원에 감사드립니다.
References
-
Badawi, N., A. E. Rosenbom, A. M. Jensen, and S. R. Sorensen, (2016), Degradation and sorption of the fungicide tebuconazole in soils from golf greens, Environ. Pollut, 219, p368-378.
[https://doi.org/10.1016/j.envpol.2016.10.045]

-
Bromilow, R. H., A. A. Evans, and P. H. Nicholls, (1999a), Factors affecting degradation rates of five triazole fungicides in two soil types: 1, Laboratory incubations. Psetic. Sci, 55, p1129-1134.
[https://doi.org/10.1002/(SICI)1096-9063(199912)55:12<1129::AID-PS72>3.0.CO;2-U]

-
Bromilow, R. H., A. A. Evans, and P. H. Nicholls, (1999b), Factors affecting degradation rates of five triazole fungicides in two soil types: 2, Field studies. Psetic. Sci, 55, p1135-1142.
[https://doi.org/10.1002/(SICI)1096-9063(199912)55:12%3C1135::AID-PS73%3E3.0.CO;2-1]

-
Fenner, K., S. Canonica, L. P. Wackett, and M. Elsner, (2013), Evaluating pesticide degradation in the environment: blind spots and emerging opportunities, Science, 341, p752-758.
[https://doi.org/10.1126/science.1236281]

-
Jarocki, P., M. Podleśny, E. Komoń-Janczara, J. Kucharska, A. Glibowska, and Z. Targoński, (2016), Comparison of various molecular methods for rapid differentiation of intestinal bifidobacteria at the species, subspecies and strain level, BMC Microbiol, 16, p159.
[https://doi.org/10.1186/s12866-016-0779-3]

- KCPA, (2016), Agrochemical year book, Korea Crop Protection Association, Seoul, Korea.
-
Kim, I. S., J. H. Shim, and Y. T. Suh, (2003), Laboratory studies on formation of bound residues and degradation of propiconazole in soils, Pest Manag. Sci, 59, p324-330.
[https://doi.org/10.1002/ps.642]

-
Miller, T. R., A. L. Delcher, S. L. Salzberg, E. Saunders, J. C. Detter, and R. U. Halden, (2010), Genome sequence of the dioxin-mineralizing bacterium Sphingomonas wittichii RW1, J. Bacteriol, 192, p6101-6102.
[https://doi.org/10.1128/JB.01030-10]

-
Mu, X., T. Chai, K. Wang, L. Zhu, Y. Huang, G. Shen, Y. Li, X. Li, and C. Wang, (2016), The developmental effect of difenoconazole on zebrafish embryos: A mechanism research, Environ. Pollut, 212, p18-26.
[https://doi.org/10.1016/j.envpol.2016.01.035]

- Research and markets, (2014), Global fungicides market, (type, http://www.researchandmarkets.com/research/7dtjwc/global_fungicides).
-
Sehnem, N. T., P. Souza-Cruz, C. Peralba Mdo, and M. A. Ayub, (2010), Biodegradation of tebuconazole by bacteria isolated from contaminated soils, J. Environ. Sci. Heal. B, 45, p67-72.
[https://doi.org/10.1080/03601230903404499]

-
Taxvig, C., U. Hass, M. Axelstad, M. Dalgaard, J. Boberg, H. R. Andeasen, and A. M. Vinggaard, (2007), Endocrine-disrupting activities in vivo of the fungicides tebuconazole and epoxiconazole, Toxicol. Sci, 100, p464-473.
[https://doi.org/10.1093/toxsci/kfm227]

-
Zhou, J., J. Zhang, F. Li, and J. Liu, (2016), Triazole fungicide tebuconazole disrupts human placental trophoblast cell functions, J. Hazard. Mater, 308, p294-302.
[https://doi.org/10.1016/j.jhazmat.2016.01.055]