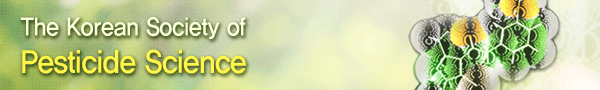
인삼 지상부 주요 병원균의 Pyraclostrobin, Fluazinam과 Prochloraz에 대한 감수성 변화
초록
인삼 지상부에 발생한 잿빛곰팡이병, 점무늬병, 탄저병의 병징에서 Botrytis cinerea, Alternaria panax, Colletotrichum panacicola를 2005년과 2006년, 그리고 2016년과 2017년에 각각 분리하여 pyraclostrobin, fluazinam, prochloraz에 대한 감수성 변화를 조사하였다. 살균제의 효과는 한천희석법을 사용하여, 병원균의 균사생장 억제효과를 조사하였다. B. cinerea, A. panax, C. panacicola 모두 2016년과 2017년에 분리한 병원균이 pyraclostrobin에 대한 감수성이 감소하면서 저항성 균주의 발생을 확인할 수 있었다. B. cinerea는 2005년과 2006년 균주에서 이미 저항성 균주가 분리되었지만, 2016년과 2017년에는 저항성 균주의 분리 비율이 93.5%까지 상승하였다. A. panax의 경우 EC50값이 10.0 μg/ml를 넘어가는 균주가 2균주 분리되었다. 특히 C. panacicola의 경우에는 저항성 균주가 전혀 분리되지 않았던 2005년과 2006년과는 다르게, 2016년과 2017년에는 93.0%가 분리되었다. 하지만 fluazinam과 prochloraz-manganese에 대해서는 3종의 병원균 모두에서 저항성 균주가 분리되지 않았다. Pyraclostrobin 저항성 균주를 확인하기 위해서 병원균의 cyt b 유전자를 분석한 결과, 저항성 균주에서는 모두 G143A의 점돌연변이가 발견되었다. 치환된 염기서열을 이용하여 allele-specific PCR을 수행한 결과, 저항성 균주인 B. cinerea, A. panax, C. panacicola에서만 260 bp, 250 bp, 352 bp의 PCR 산물이 증폭되었다. 본 연구를 통해서 확립된 allele-specific PCR은 포장에서 분리한 인삼 병원균의 pyraclostrobin에 대한 저항성 여부를 신속하고 정확하게 검정할 수 있을 것으로 생각한다.
Abstract
In 2005 and 2006, and in 2016 and 2017, Botrytis cinerea, Alternaria panax, and Colletotrichum panacicola were isolated from lesions of gray mold, leaf spot, and anthracnose on the aerial parts of ginseng. Pathogenic susceptibility to pyraclostrobin, fluazinam, and prochloraz was investigated by using all isolates of these pathogens found in ginseng. The efficacy of the fungicides was tested by using the agar dilution method to inhibit the mycelial growth of the pathogens. Showing a decrease in susceptibility of all the pathogens isolated in 2016 and 2017 to pyraclostrobin, it was possible to be confirmed the occurrence of resistant isolates. Although resistant isolates were already obtained from B. cinerea isolates in 2005 and 2006, the isolation rate of resistant isolates increased to 93.5% in 2016 and 2017. For A. panax, two isolates, APES12 and APES9-5, with EC50 value exceeding 10.0 μg/ml were collected in 2016. For C. panacicola specially, 93.0% of resistant isolates were collected in 2016 and 2017. In contrast, no resistant isolates were collected in either 2005 or 2006. Furthermore, none of the three pathogens studied had any confirmed resistant isolates to fluazinam and prochloraz-manganese. To confirm pyraclostrobin-resistant isolates, analysis of the cyt b gene revealed the presence of the point mutation G143A in all resistant isolates. Through the allele-specific PCR, PCR products of 260 bp, 250 bp, and 352 bp were amplified only from the resistant isolates B. cinerea, A. panax, and C. panacicola. It is our conclusion that the allele-specific PCR protocol established through this study can rapidly and accurately test pyraclostrobin-resistance of ginseng pathogens isolated from the field.
Keywords:
Ginseng disease, Botrytis cinerea, Alternaria panax, Colletotrichum panacicola, Resistance to pyraclostrobin, Allele-specific PCR키워드:
인삼의 병, pyraclostrobin 저항성서 론
인삼은 저온 다습한 재배 조건과 한 포장에서 몇 년 동안 재배를 하기 때문에 병해충에 의한 피해가 심하다. 인삼에 발생하는 식물병은 20여종이 보고되어 있는데, 이 중 잿빛곰팡이병, 점무늬병, 탄저병은 지상부에서 발생하여 인삼의 생육과 생산량에 피해를 준다(Kim et al., 2008). 인삼잿빛곰팡이병원균인 Botrytis cinerea는 기주 범위가 넓은 식물 병원균으로 인삼의 잎, 열매, 줄기의 지재부 등을 침입하고, 병반 상에 회색의 분생포자와 검은색의 균핵을 형성한다. B. cinerea는 4월에 올라오는 인삼의 뇌두를 침입하기 시작하고, 6월 중순 이후 특히 장마철에는 잎과 열매를 침입한다. 인삼 점무늬병은 Alternaria panax에 의해 발생하는 병으로, 5월 중순부터 6월 상순에 잎과 잎자루에 발생한다. 초기 부정형의 갈색 병반이 형성되기 시작하여 엽맥을 따라 큰 부정형 괴사 병반을 형성하며 심하면 잎이 낙엽지기도 하는데, 병원균은 바람에 의해 쉽게 전파되는 공기전염성 병이다(Lee et al., 1990; Putnama and du Toit, 2003; Uchida et al., 1984). 인삼 탄저병은 1922년 처음 보고되었으며 Colletotrichum panacicola에 의해 잎, 줄기, 열매에 모두 발생한다(Chung and Bae, 1979; Han et al., 2004). 주로 잎에 발생이 심한데, 발생 초기에 잎에 연한 갈색 반점이 형성되고, 확대되면서 부정형의 큰 병반을 형성한다. 병원균은 빗방울에 의해서 전반되며, 장마철 이후 고온 다습한 기후에서 발생하여 큰 피해를 준다.
인삼 지상부의 주요한 병인 3종의 병을 방제하기 위해서는, 살균제를 처리하여 방제하는 방법이 확실한 방제 효과를 기대할 수 있다(Kim et al., 2007). 잿빛곰팡이병, 점무늬병, 탄저병 등의 방제를 위하여 공통으로 등록되어 있는 살균제는 주로 다3군, 다5군, 사1군 등에 포함되어 있다. 라1군 살균제는 탄저병을 제외한 잿빛곰팡이병과 점무늬병의 방제에 사용된다. 동일한 작물에서 발생하는 다른 병을 방제하기 위하여 처리하는 살균제가 중복될 경우, 인지하지 못하는 과정에서 한 종류의 병원균에 대해서 동일한 작용기작을 갖는 살균제가 여러 차례 중복 처리되는 경우가 생길 수 있다. 이미 인삼에서 분리한 B. cinerea를 가지고 일부 살균제에 대한 저항성을 검정한 결과를 보더라도 저항성균의 발생이 심각한 상황임을 알 수 있다. Benzimidazole계에 속하는 carbendazim에 대한 저항성 기준을 EC50값 기준으로 10.0 μg/ml로 결정하고, 저항성 검정을 실시한 결과, 2005년에 분리한 89개 균주 중 87.6%가, 2006년에 분리한 147개 균주 중에서는 96.6%가 저항성이었다(Kim, 2006). 다3 군에 속하는 strobilurin계 살균제는 넓은 적용 범위를 갖는 살균제로서, 매우 다양한 식물병원균의 방제에 빈번하게 사용되다보니 저항성 발생이 많이 보고되고 있으며, 국내에서도 B. cinerea와 Colletotrichum spp. 등에서 저항성이 보고되어 있는 상태이다(Kim et al., 2014; Kim et al., 2019). 하지만 5월 중순 이후부터 연속적으로 발생하는 인삼 지상부 병원균인 B. cinerea, A. panax, C. panacicola에 대해서 공통적으로 등록되어 사용하고 있는 살균제에 대한 종합적인 저항성 검정은 이루어지지 않은 상태이다. 따라서 본 연구에서는 인삼 지상부에서 잿빛곰팡이병, 점무늬병, 탄저병을 일으키는 3종의 병원균에 대해서 pyraclostrobin과 fluazinam, prochloraz-manganese 등 총 3종의 살균제를 선발하여, 각 병원균의 살균제 저항성 검정을 실시하였다. 이를 위해서 2005년과 2006년에 분리한 각 병원균의 균주와 2016년과 2017년에 분리한 균주를 사용하여, 약 10년 동안의 기간 사이에 각 병원균의 살균제에 대한 감수성 변화를 조사하였다. 또한 pyraclostrobin에 대해서 저항성인 병원균의 cyt b 유전자를 분석하여 점 돌연변이의 발생이 저항성 발현에 영향을 미치는지의 여부를 조사하였다. 또한 점 돌연변이 위치를 기반으로 저항성균의 검출을 신속하고 용이하게 검출할 수 있는 allele-specific PCR 방법을 확립하였다.
재료 및 방법
병원균 채집 및 분리
인삼의 잿빛곰팡이병균, 점무늬병균, 탄저병균은 2005년과 2006년에 Kim(2006)과 Lee(2008)이 병든 인삼 잎에서 단포자 분리하여 충북대학교 식물진균병학실험실에서 보관하던 균주와 2016년과 2017년에 분리한 균주를 사용하였다. B. cinerea는 괴산, 충주, 음성, 증평 등 충북 4개 지역의 인삼 포장에서 잿빛곰팡이병의 병징을 보이는 줄기 및 잎을 채집하여 병원균을 단포자 분리하였다. 채집한 병반은 화염 멸균한 칼로 건전 부위와 병징 부위를 1:1의 비율이 되도록 5x5 mm의 크기로 잘라 1% 차아염소산나트륨과 70% 에탄올에서 표면 소독한 후, 멸균증류수로 세척하였다. 표면소독한 조직을 여과지에 올려서 건조시킨 후, 물 한천 배지에 올려놓고 20oC에서 배양하였다. 암 조건에서 1일간 배양한 뒤, 배지에 형성된 균총의 선단에서 균사 조각을 떼어 300 μg/ml의 streptomycin이 첨가된 potato dextrose agar (PDA; DifcoTM, Becton, Dickinson and Company, Sparks, MD21152 USA) 배지에 옮겨 배양한 후, 형성된 분생포자를 단포자 분리하여 순수배양체를 확보하였다. A. panax는 충남 금산과 충북 음성, 증평의 인삼 포장에서 병든 인삼의 줄기 및 잎을 채집하여 25oC의 플라스틱 상자에서 습실 처리하였다. 병반 표면에 형성된 분생포자를 해부현미경으로 관찰하며, 멸균한 파스퇴르 유리 피펫(지름: 1.5-1.8 mm, 길이: 90 mm)을 사용하여 단포자 분리하였다. 떼어낸 분생포자 한 개를 25oC의 PDA 배지에 접종하여 2일간 배양한 후, 배지에 형성된 균총의 선단부를 떼어 순수배양체를 확보하였다. C. panacicola는 괴산, 충주, 음성, 증평 등 충북 4개 지역과 강원도 홍천의 인삼 포장에서 탄저병의 병징을 보이는 잎을 채집하여 단포자 분리하였다. 병징 상에 형성된 탄저병균의 포자를 화염 멸균한 백금이를 이용하여 떼어 300 μg/ml의 streptomycin이 첨가된 멸균증류수에 현탁시켜 포자 현탁액을 만들었다. 포자 밀도를 1×104 개/ml로 조절한 현탁액 50 μl를 300 μg/ml의 streptomycin이 첨가된 PDA 배지에 고르게 도말한 후, 25oC에서 2일간 배양하였다. 배지에 형성된 균총을 떼어내어 순수배양체로 사용하였다. 실험에 사용한 세 가지 병원균의 균주 수는 B. cinerea가 44균주, A. panax가 40균주, C. panacicola가 76균주이었다.
병원균 동정
인삼 잿빛곰팡이병균, 점무늬병균, 탄저병균의 ITS 영역의 염기서열을 분석하여 병원균을 동정하였다. 병원균의 균사를 PDA 배지에 접종하여, 잿빛곰팡이병균은 20oC 암조건에서 3일, 점무늬병균은 25oC 암조건에서 10일, 탄저병균은 25oC 암조건에서 7일 동안 배양하였다. 배지 표면에 형성된 각 병원균의 균사를 멸균한 슬라이드글라스로 긁어 수확하여 2 ml의 eppendorf microtube에 glass beads (지름; 0.1 mm)와 함께 넣고, -70oC에서 12시간 이상 동결 건조한 후, bead beater (bullet blender, NEXTADVANCE, USA)를 이용하여 마쇄하였다. Exgene Plant SV kit (ExgeneTM Plant SV, GENEALL BIOTECHNOLOGY, KOREA)를 이용하여 마쇄한 균사체에서 gDNA를 추출하였다. ITS5 (5’-GGA AGT AAA AGT CGT AAC AAG G-3’)와 ITS4 (5’-TCC TCC GCT TAT TGA TAT GC-3’) primer를 이용하여 병원균의 ITS 영역을 증폭한 후 염기서열을 분석하였다. 유전자 증폭을 위하여 희석된 gDNA 2 μl, EzPCRTM 5x PCR Master Mix 4 μl, 각각의 primer 1 μl, 멸균증류수 12 μl를 혼합해 총 20 μl의 PCR 반응액을 만들었다. ITS 영역을 증폭하기 위하여 95oC에서 4분 pre-denaturation을 수행한 뒤, 95oC에서 denaturation 30초, 52oC에서 annealing 30초, 72oC에서 extension 45초의 반응을 총 35회 반복하였고, 72oC에서 7분 final extension 단계를 거친 뒤 반응을 종료하였다. PCR 산물 확인을 위하여 0.5X TBE buffer와 agarose E를 이용해 1% agarose gel을 만들고 safe viewTM classic을 권장 사용량에 맞게 첨가한 후, 100 V의 전압에서 40분 동안 전기영동하였다. UV-transilluminator (FluoroBox, NEO science, KOREA)를 사용하여 증폭된 PCR 산물을 확인하였다. ExpinTM PCR SV kit (ExpinTM PCR SV kit, GENEALL BIOTECHNOLOGY, KOREA)를 사용하여 PCR 증폭 산물을 정제한 뒤, Macrogen (Daejen, Korea)에 의뢰하여 증폭된 유전자의 염기서열을 분석하였다. 증폭한 병원균의 염기서열은 NCBI (National Center for Biotechnology Information) 홈페이지의 Basic Local Alignment Search Tool (BLAST, https://blast.ncbi.nlm.nih.gov/Blast.cgi)에서 유사도를 확인한 후 동정하였다.
인삼 병원균에 대한 살균제의 효과 검정
인삼 지상부의 3가지 병원균에 대한 pyraclostrobin (a.i. 20% WG, 다3), fluazinam (a.i. 50% WP, 다5), prochloraz-manganese (a.i. 50% WP, 사1)의 균사생장 억제효과를 직경 6 mm의 Petri 접시에 PDA 배지를 부어 한천희석법으로 조사하였다. 병원균에 대한 pyraclostrobin의 균사생장 억제효과를 조사하기 위해서, PDA 배지에 대체 호흡을 억제하는 salicylhydroxamic acid (SHAM; Sigma-Aldrich Chemical Sciences Co., St. Louis, Missouri, USA)을 dimethyl sulfoxide (DMSO; Junsei Chemical Co., Ltd., Tokyo, Japan)에 용해시켜 최종 농도가 100 μg/ml가 되도록 첨가하여 실험하였다. 또한 세균 오염을 방지하기 위해 모든 PDA 배지에 streptomycin의 최종농도가 300 μg/ml이 되도록 첨가하였다. 각 병원균에 대해서 실험에 사용한 3종 살균제의 배지에서 농도는 Table 1과 같이 조절하여 사용하였다. 한천희석법을 위해서 B. cinerea는 20oC에서, A. panax와 C. panacicola는 25oC에서 일정 기간을 배양한 후, 균총의 선단부에서 직경 3 mm의 균사 조각을 떼어내어 접종원으로 사용하였다. 접종원으로 떼어낸 균사 조각은 균사면이 배지 표면에 닿도록 뒤집어서 접종하였다. 모든 병원균은 살균제를 첨가하지 않은 무처리 배지에서 균총의 직경이 30 mm 이상이 되었을 때 각 처리구에서 균총의 직경을 조사하였다. 살균제의 균사생장 억제율(%)은 살균제 무처리구와 처리구의 균총의 직경을 비교하여 아래와 같이 계산하였다.
병원균의 RNA 추출과 cDNA 합성
동결 건조한 병원균의 균사체를 bead beater를 이용하여 마쇄하고, Hyrid-R kit (GeneAll Hyrid-R, GENEALL BIOTECHNOLOGY, KOREA)를 이용하여 RNA를 추출하였다. 추출한 RNA는 nano drop (OPTIAEN NanoQ, KLAB, KOREA)를 사용해 순도와 농도를 확인하였고, 모든 RNA는 50 ng/ml로 희석하여 실험에 이용하였다. 추출한 RNA는 5X RT Master Mix (Reverse Transcription 5X Master Mix, ELPISBIOTECH, KOREA)을 이용하여 cDNA를 합성하였다. 합성을 위해 5X RT Master Mix 4 μl, Nuclease-free water 11 μl, RNA 5μl를 첨가하여 반응액을 만든 뒤, 37oC에서 60분, 94oC에서 5분 PCR을 수행한 후 종료하였다.
Cyt b 유전자의 증폭
Cyt b 유전자를 증폭하기 위하여 B. cinerea는 gDNA를 사용하였으며, A. panax와 C. panacicola는 추출한 RNA를 사용하여 합성한 cDNA를 주형으로 사용하였다. Table 2에서 보는 것과 같이, B. cinerea는 Qo 13ext/Qo 14ext primer를 사용하였으며, A. panax는 CBF1/Apcytb-R primer를 사용하였다(Ma et al., 2003; Samuel et al., 2011). C. panacicola는 본 연구 과정에서 자체 제작한 Cpcyt-F/Cpcyt-R primer를 사용하였다. 유전자 증폭을 위한 PCR 조건은 95oC에서 2분 pre-denaturation을 수행한 뒤, 95oC에서 1분 denaturation, 1분 annealing, 72oC에서 1분 extension을 35회 반복하였고, 72oC에서 5분 final extension 단계를 거친 뒤 반응을 종료하였다. 각 병원균에 따라서 annealing 온도를 B. cinerea는 56oC, A. panax는 56.1oC, C. panacicola는 54.5oC로 조절하였다. 확보된 PCR 산물들을 확인하기 위해 0.5X TBE buffer로 1% agarose gel을 만들어 safe viewTM classic을 권장 사용량에 맞춰 첨가하였으며, PCR 산물에 loading dye를 산물 5 μl 당 1 μl씩 첨가하여 75 V에서 60분간 전기영동 한 후 UV-transilluminator를 사용하여 증폭산물을 확인하였다. 증폭된 시료들은 ExpinTM PCR SV kit(GeneAll, Seoul, korea)를 사용해 정제한 후 Macrogen(Daejeon, Korea)에 염기서열 분석을 의뢰하였다. 분석된 cytochrome b 유전자 염기서열은 BioEdit ver. 7.0.9.0을 이용해 정리한 뒤, PHYDIT ver. 3.1에서 정렬하였다. NCBI GenBank의 염기서열을 통해 비교하여 유연관계를 분석하였다.
Allele-specific PCR을 이용한 병원균의 저항성 검정
Pyraclostrobin에 대한 저항성 균주와 감수성 균주를 보다 빠르게 검출하기 위해 B. cinerea는 Jiang(2009)이 사용한 primer 한 쌍과 A. panax와 C. panacicola는 pyraclostrobin 저항성 균주의 cytochrome b 유전자에서 아미노산 치환 부위를 사용해 reverse primer 3′말단 끝이 저항성 균주들의 치환된 428번째 염기가 위치하도록 allele-specific primer를 제작하여 allele-specific PCR을 수행하였다(Table 3). PCR 반응액은 DNA 100 ng, 10x Thumb Taq Reaction Buffer 2.5 μl, dNTP (각 10 mM) 0.5 μl, 10 pmole의 Qo13ext와 Qo14ext primer 각 1 μl, 5X Band HelperTM 2.5 μl, BioFACTTM Thumb Taq (2.5 units/μl, BioFACTTM, Daejeon, Korea) 0.25 μl, 멸균증류수 첨가하여 총 25 μl가 되게 만들었다. 반응조건은 3분간 95oC에서 pre-denaturation을 수행한 후, 40초 동안 94oC에서 denaturation, 40초 동안 각 primer에 따라 온도를 달라하여 annealing, 1분 30초 동안 72oC에서 extension을 35회 반복하였고, 72oC에서 5분 동안 반응시킨 후 종료하였다(Table 6.). 얻어진 PCR 산물들을 확인하기 위해 0.5X TBE buffer로 1% agarose gel을 만들어 safe viewTM classic을 권장 사용량에 맞춰 첨가하였으며, PCR 산물에 loading dye를 산물 5 μl 당 1 μl씩 첨가하여 75 V에서 60분간 전기영동 한 후 UV-transilluminator를 사용하여 증폭산물을 확인하였다.
결과 및 고찰
인삼 지상부 3가지 병의 병원균 분리 및 동정
병든 인삼에서 분리한 잿빛곰팡이병균과 점무늬병균, 탄저병균은 ITS5와 ITS4의 primer를 사용하여 증폭한 ITS 영역 염기서열의 유사성을 NCBI에서 조사한 결과, B. cinerea, A. panax, C. panicicola로 동정되었다. Alternaria 속과 Colletotrichum 속 병원균을 정확하게 동정하기 위해서는 다중 유전자 염기서열을 이용한 계통 분석을 실시하여야 하지만, 본 연구에서는 살균제의 반응을 조사하는 것이 목적이었음으로 다중 유전자의 염기서열 분석을 통한 병원균의 동정은 실시하지 않았다.
분리 시기가 다른 병원균 집단의 살균제에 대한 감수성 변화
Table 4에서 보는 것과 같이, 각 병원균의 평균 EC50값을 계산할 때, EC50값이 50.0 μg/ml 이상인 균주의 EC50값은 50.0 μg/ml로 계산하였다. 실험에 사용한 pyraclostrobin, fluazinam, prochloraz의 수용해도가 1.9 mg/l, 0.135 mg/l, 34.4 mg/l이기 때문에, 너무 높은 EC50값에 대한 신뢰도가 크지 않다. 따라서 평균 EC50값을 계산할 때, 50.0 μg/ml 이상의 값을 모두 50.0 μg/ml로 계산하였다.
Pyraclostrobin에 대한 3종 병원균의 EC50값을 비교하여 보면, 2005-2006년 균주보다 2016-2017년 균주 집단에서 증가하였으나, 통계적으로 유의성 있게 증가한 결과는 탄저병균밖에 없었다. B. cinerea의 평균 EC50값을 2005-2006년 집단과 2016-2017년 집단 간에 비교하여 보면 29.298 μg/ml (0.039 - 50.0 μg/ml 이상)에서 41.344 μg/ml (0.117 - 50.0 μg/ml 이상)로 상승하였다. 그런데, 평균 EC50값이 상승하였음에도 불구하고 통계적인 유의성이 없었던 것은, 2005-2006년 집단에서 이미 저항성인 균주가 다수 분리되었기 때문에 평균 EC50값에서 2016-2017년 집단과 통계적으로 유의한 차이가 없었다(Fig. 1A). A. panax 역시 평균 EC50값이 1.889 μg/ml (0.013 – 6.430 μg/ml)에서 3.714 μg/ml (0.117 – 39.712 μg/ml)로 상승하였지만, 통계적인 유의성은 없었다(Fig. 1B). 반면에 C. panacicola에 대한 2005-2006년 집단의 평균 EC50값은 0.017 μg/ml (0.001 이하 - 0.118 μg/ml)에서 2016-2017년에는 11.837 μg/ml (0.001 이하 - 50.0 μg/ml 이상)로 상승하면서 통계적으로도 높은 유의성을 나타내었다. 이 결과는 Fig. 1C에서 보는 것처럼 2005-2006년 집단에는 저항성인 균주가 전혀 없었던 반면에, 약 10년이 지난 후인 2016-2017년 집단에서는 93.0%의 균주가 저항성인 균주로 나타났기 때문이다.
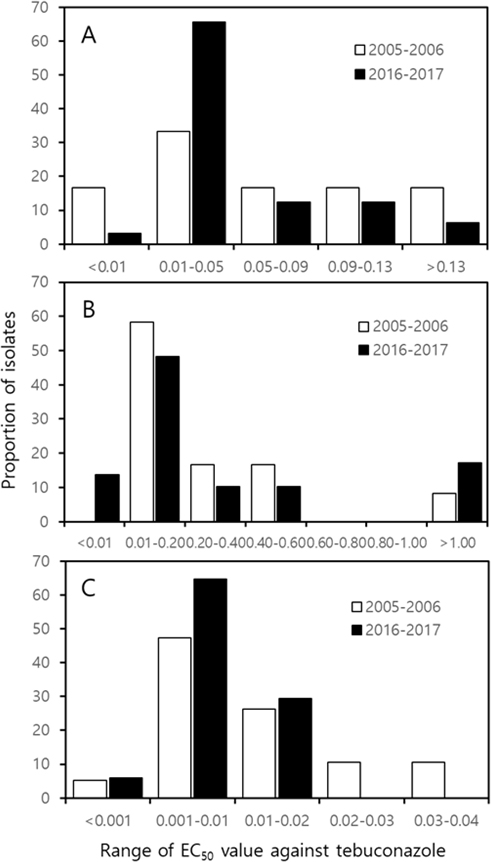
Distribution of EC50 value (μg/ml) of Botrytis cinerea (A, D and G), Alternaria panax (B, E and H) and Colletotrichum panacicola (C, F and I) to pyraclostrobin (A, B and C), fluazinam (D, E and F) and prochloraz (G, H and I), respectively. The isolates of pathogens used in the experiment were collected in 2005 and 2006, and in 2016 and 2017, respectively. The EC50 value represents the concentration that inhibits mycelial growth by 50%. The EC50 value of each isolate for each fungicide was calculated by obtaining the mycelial growth inhibition rate using the agar dilution method.
Fluazinam은 3종의 병원균 모두에 대해서 높은 균사생장 억제효과를 보이고 있었으며, 2005-2006년과 2016-2017년 균주 집단의 평균 EC50값에 큰 차이가 없었고, 통계적으로도 유의성이 없었다. B. cinerea의 경우 2005-2006년에 분리한 B. cinerea GBGK16만이 다른 균주에 비해서 높은 EC50값을 보였나, 그 값이 0.009 μg/ml로 매우 낮기 때문에, 저항성으로 판정하기 어려웠다.
Prochloraz의 효과도 분리 시기가 다른 각 병원균의 균주 집단에 대해서 통계적인 유의성은 없었다. 식물 병원성 진균의 ergosterol 생합성을 저해하는 prochloraz는 양적 저항성 보이는 살균제로 알려져 있다(Deising et al., 2008). 질적 저항성을 보이는 실균제의 감수성과 저항성 균주 간에는 살균제 반응에서 현저한 차이가 있지만, 양적 저항성을 보이는 살균제에 대한 병원균의 감수성 반응은 서서히 감소하기 때문에, 저항성 병원균의 발생을 판단하기가 쉽지 않다. Fig. 2에서 보는 것과 같이 균주의 최빈값 구간은 분리한 기간과 관계없이 B. cinerea는 0.01-0.05 μg/ml, A. panax는 0.01-0.20 μg/ml, 그리고 C. panacicola는 0.001-0.01 μg/ml로 동일하였다. C. panacicola의 경우 2016-2017년 집단에서 EC50값이 0.02 μg/ml 이상인 균주의 비율이 2005-2006년 집단보다 감소하였지만, B. cinerea와 A. panax의 분포는 매우 유사하였다. 이처럼 EC50값의 분포가 유사한 것을 보면, 병원균의 prochloraz에 대한 반응이 크게 변화하지 않았음을 알 수 있다. 하지만 A. panax에서 EC50값이 1.0 μg/ml/를 상회하는 균주가 2005-2006년에는 한 균주 밖에 없었는데, 2016-2017년에는 5균주로 증가하고 있기 때문에 인삼점무늬병균의 prochloraz에 대한 저항성 검정이 지속적으로 진행되어야 할 필요가 있다.
cyt b 유전자의 분석
Fig. 1에서 보는 것과 같이 3종 살균제 중에서 저항성 균주를 확인할 수 있었던 살균제는 pyraclostrobin이었다. 그런데 균사생장 억제효과를 검정하는 한천희석법의 결과를 가지고서만 저항성 발생을 정확하게 결정하기란 쉽지 않다. Fig. 1에서 보는 것과 같이 실험한 각 병원균 균주의 EC50값에 차이가 있다고 하여도, 저항성의 기준이 되는 EC50값의 농도를 설정하기란 매우 어렵다. 따라서 pyraclostrobin과 같이 질적 저항성을 보이는 살균제의 저항성 검정에서는 살균제 작용점 유전자에서의 돌연변이 여부를 조사해야 한다(Holger et al., 2008; Yin et al., 2023). 이미 Botrytis, Alternaria, Colletotrichum 속에서는 QoI 살균제에 대한 저항성 발현이 보고되어 있다(Avenot and Michailides, 2020; Kim et al., 2014; Kim et al., 2019). 사과에서 분리한 220개의 Botrytis cinerea 균주 중에서 19.5%인 43균주가 pyraclostrobin에 대해서 저항성인 것으로 판명되었으며, 모든 저항성 균주는 cyt b 유전자에서 G143A의 돌연변이를 가지고 있었다(Yin et al., 2012). 미국 캘리포니아에서는 Alternaria spp.의 QoI 살균제에 대한 저항성을 검정하기 위해서, azoxystrobin을 처리한 이력이 있는 피스타치오 과원과 처리 이력이 없는 과원에서 분리한 Alternaria spp.의 균주를 대상으로 포자 발아 억제 실험을 실시하였다. 감수성 균주의 EC50값은 0.008 – 0.045 μg/ml이었던 반면에, 저항성 균주의 EC50값은 100 μg/ml 이상이었다. Azoxystrobin에 대한 EC50값이 100 μg/ml 이상인 Alternaria spp. 균주의 cyt b 유전자 분석 결과, B. cinerea와 동일하게 G143A의 돌연변이가 발견되었다(Ma et al., 2003). 다양한 작물에서 병을 일으키는 Colletotrichum속의 병원균에서도 QoI 살균제에 대한 저항성이 보고되어 있다. 고추, 블루베리, 사과, 딸기, 포도, 복숭아, 잔디 등 다양한 작물에서 분리한 QoI 살균제 저항성인 Colletotrichum spp.의 cyt b 유전자에서 G143A와 F129L의 돌연변이가 발견되었다(Cortaga et al., 2023).
본 논문에서도 실험에 사용한 3종 병원균의 pyraclostrobin에 대한 저항성이 cyt b 유전자의 143번째 아미노산의 치환에 의해서인 지를 조사하기 위해서 각 병원균의 cyt b 유전자를 분석한 결과, 143번째의 아미노산 코돈이 GGT인 glycine에서, GCT인 alanine으로 치환되어 있음이 밝혀졌다(Fig. 3).

Comparison of partial nucleotide sequence of the cyt b gene amplified with each PCR primer pair in Botrytis cinerea, Alternaria panax and Colletotrichum panacicola. The primers used for amplification of the cyt b gene are as shown in Table 1. Among each pathogen, isolates susceptible and resistant to pyraclostrobin were selected, the cyt b gene was amplified, and the nucleotide sequences were compared. The isolates susceptible to pyraclostrobin were B. cinerea JP7, A. panax APES7 and APJP2, and C. panacicola PGYA2, and the isolates resistant to pyraclostrobin were B. cinerea ES10, A. panax APES9-5, and C. panacicola PGNS6.
Fig. 1에서 보는 것처럼 한천희석법에서 얻은 EC50값을 가지고서 QoI 살균제에 대한 저항성 기준을 정하기는 어려웠다. 특히 A. panax와 C. pancicola의 경우에는 그 기준의 설정이 매우 모호하여, 실험에 사용한 모든 균주의 cyt b 유전자를 분석하여 점돌연변이의 여부를 검정하였다. 그 결과 B. cinerea의 경우에는 EC50값이 1.0 μg/ml 이하인 균주에서는 cyt b 유전자의 143번째 아미노산 코돈에서의 점돌연변이가 없었지만, 2.0 μg/ml 이상의 균주에서는 G143A의 점돌연변이를 발견할 수 있었다. 2005년과 2006년에 분리한 13개의 균주 중에서는 6개의 균주가 감수성, 그리고 7개의 균주가 저항성이었으며, 2016년과 2017년에 분리한 31개 균주 중에서는 2개가 감수성 균주였으며, 29개의 균주가 저항성 균주로 나타났다(Fig. 1B). 이미 10여년 전부터 저항성 균주의 분리 비율이 53.8%를 상회하였으며, 10여년이 지난 후에는 93.5%를 상회하였다. A. panax의 경우는 2005년과 2006년에 분리한 12개의 균주 중에서 EC50값이 6.430 μg/ml로 가장 높았던 GJHW1 균주에서는 점돌연변이가 없었으며, 2016년과 2017년에 분리한 28개 균주 중에서는 EC50값이 14.141과 39.712 μg/ml인 APES12와 APES9-5에서만 점돌연변이가 발견되었고, 8.204와 8.994 μg/ml인 APJP2와 PGGS5에서는 점돌연변이가 발견되지 않았다(Fig. 1C). A. panax에서 보는 것처럼 질적 저항성을 보이는 pyraclostrobin에 대한 저항성 판정 기준을 한천희석법을 통해서 얻은 병원균의 EC50값만을 가지고서 결정할 수는 없으며, 병원균의 cyt b 유전자 분석을 통한 점돌연변이 유무를 동시에 검정하여 저항성 발현에 대한 판정을 내려야 한다. 이런 과정을 거쳐서 cyt b 유전자에서 점돌연변이를 가지는 A. panax 균주의 EC50값은 10.0 μg/ml 이상이었으므로, Fig. 1B에서는 40개의 A. panax 균주 중에서 APES12와 APES9-5만이 저항성으로 판정하였다. QoI 살균제에 대해서 저항성인 Alternaria spp.에서는 G143A의 점돌연변이뿐만 아니라 F129L의 점돌연변이도 보고되어 있다(Duba et al., 2018; Odilbekov et al., 2016). 하지만 인삼에서 분리한 2개의 저항성 균주는 G143A의 돌연변이만이 검출되었고, F129L 점돌연변이는 없었다. A. panax의 나머지 균주에서도 F129L 돌연변이는 확인할 수 없었다. C. panacicola의 경우에는 2005년과 2006년에 분리한 33개 균주의 EC50값은 모두 1.0 μg/ml 이하이었으나, 2015년과 2016년 분리 균주 중에서는 PGYA2 균주를 비롯한 3개 균주만이 1.0 μg/ml 이하이었고, 나머지 40개 균주는 1.0 μg/ml 이상이었다. Cyt b 유전자 분석을 통한 점돌연변이 확인 결과에서도, EC50값이 1.0 μg/ml 이상이었던 균주에서는 모두 G143A 점돌연변이를 확인할 수 있었다. 이상의 결과를 통해서 pyraclostrobin에 대한 B. cinerea, A. panax, C. panacicola의 저항성 기준은, EC50값을 사용할 경우 각각 2.0, 10.0, 1.0 μg/ml로 결정하였으며, 저항성 발현 여부 확정을 위해서는 반드시 cyt b 유전자 상에서의 점돌연변이 발생 유무를 확인해야 한다.
병원균에 대한 allele-specific PCR.
Allele-specific PCR은 여러 가지 식물병원균의 살균제 저항성 검정에 사용할 수 있는 간편하고 신속한 검정 방법이다(Isa and Kim, 2022; Ma and Michailides, 2005). Benzimidazole계 살균제에 대해서 고도 저항성을 보이는 Monilinia fructicola는 β-tubulin 유전자의 198번째 코돈이 GAA에서 GCA로 치환되어 있었으며, 이 치환 부위를 이용하여 HRR/HRF의 allele-specific PCR의 primer를 제작하였다. 이 primer는 고도 저항성을 보이는 M. fructicola에서만 469 bp의 산물이 증폭되기 때문에 쉽게 저항성 검정을 할 수 있었다(Ma et al., 2003b). 본 실험에서도 실험에 사용한 3종의 병원균 각각에서 감수성과 저항성 균주를 임의로 15개씩 선발하여, allele-specific PCR을 수행하였다. 실험에 사용한 3종 병원균의 cyt b 유전자에서 점 돌연변이가 발생한 특정 부위만을 증폭시킬 수 있는 Bc-AR-F/Bc-AR-R, CBF1/Apcyt-al-R, Cpcyt-F/Cpcyt-al-R primer를 제작하여 allele-specific PCR을 수행한 결과, 저항성인 B. cinerea, A. panax, C. panacicola의 균주에서만 260 bp, 250 bp, 352 bp의 PCR 산물이 증폭되었다(Fig. 4). Allele-specific PCR은 포장에 살균제 저항성 균이 존재하는 지의 여부를 신속하게 검정할 수 있는 매우 유용한 검정 기술이기 때문에, 포장에서 살균제 저항성 관리에는 필요한 검정 방법이다(Ma et al., 2003b). 그런데 allele-specific PCR 방법은 살균제 저항성 균의 존재 여부만을 검정할 수 있고, 양적으로 어느 정도의 저항성 균이 존재하는 지의 여부를 알 수는 없다. 작물을 재배하는 포장에서의 살균제 저항성 관리와 처리할 살균제를 선정하기 위해서는 저항성 균의 비율을 알 수 있는 PCR 방법의 확립도 필요하다.
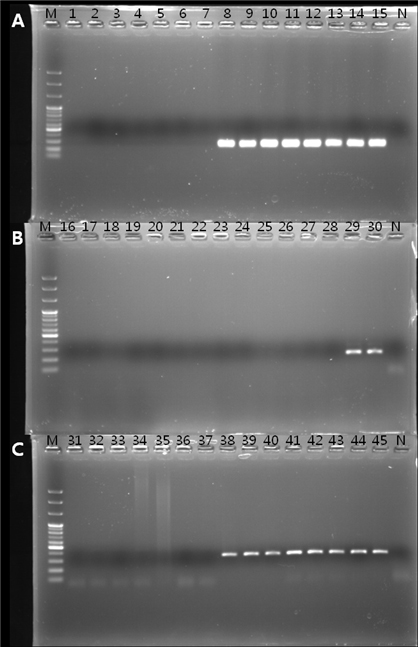
Allele-specific PCR for detecting isoletes of Botrytis cinerea (A), Alternaria panax (B) and Colletotrichum panacicola (C) resistant to pyraclostrobin. A total of 15 B. cinerea isolates were used in this experiment, numbered 1 to 15. Of these, isolates 1 to 7 were sensitive to the fungicide, and the remaining 8 isolates were resistant. The numbers for each isolate were as follows. 1; GBGN24, 2; GBJM4, 3; GBGC18, 4; 77803, 5; 70048, 6; JP7, 7; GS9, 8; ES4, 9; ES10, 10; JP9, 11; JP6, 12; GBGN47, 13; GBKP5, 14; GBES3, and 15; GBJP8. Numbers 16 to 30 were A. panax isolates. Among the 15 isolates, only APES12 and APES9-5 were resistant to pyraclostrobin. The numbers for each isolate were as follows. 16; GJJM2, 17; GJJM13, 18; GJHS1, 19; GJHW1, 20; GJYW1, 21; GJJM5, 22; GJGC1, 23; PGGS4, 24; APJP1, 25; APJP2, 26; APES1, 27; APJP2, 28; PSGS3, 29; APES12, and 30; APES9-5. C. panacaicola isolates were from 31 to 45. There were seven susceptible isolates from 31 to 37, and the rest were all resistant isolates. The susceptible and resistant isolates were as follows. 31; GPYY8, 32; GPPY3, 33; GPPS2, 34; GPOC2, 35; GPNN4, 36; GPAM1-6, 37; PGYA2, 38; PGES4, 39; PGHP1, 40; PGCUJ4, 41; PGES3, 42; PGGS7, 43; PGNS6, 44; PGYA1, 45; PGHC2. M and N represent 100 bp ladder and negative control, respectively.
본 실험의 결과에서 보는 것과 같이 10여 년의 차이를 두고 분리한 병원균의 살균제에 대한 저항성 검정을 실시한 결과, fluazinam(다5 군)과 prochloraz(사1 군)와 같이 저항성 발생에 대한 보고가 없거나, 양적 저항성을 보이는 살균제에 대해서 B. cinerea, A. panax, C. panacicola 균주의 감수성 변화는 없었다. 하지만 질적 저항성을 보이는 pyraclostrobin에 대해서는 2005년과 2006년에 이미 저항성이 발현되어 있던 B. cinerea의 저항성 분리 비율이 상승하였고, 전혀 나타나지 않았던 C. panacicola처럼 살균제의 사용 기간이 길어지면서 저항성 균주 분리 비율이 93.0%를 상회하게 되는 경우도 있었다. 반면에 A. panax는 저항성 균주가 분리되는 비율이 7.1%에 그치고 있었다. 이상의 결과에서 보면 pyraclostrobin에 대한 식물 병원균의 저항성화는 병원균의 종류에 따라서 달라질 수 있음을 알 수 있었다. 또한 저항성 검정을 하는 살균제의 저항성 종류가 질적 저항성인가 양적 저항성인가에 따라서도 저항성 발생 정도에 큰 차이가 있음을 알 수 있었다. 질적 저항성의 대표적인 살균제인 pyraclostrobin에 대해서는 인삼의 주요 지상부 병원균인 B. cinerea, A. panax, C. panacicola 모두에서 저항성인 균주가 발생하였던 반면에, 양적 저항성을 보이는 prochloraz에 대해서는 특별히 저항성 균주의 발생을 확인할 수 없었다.
Acknowledgments
이 논문은 과학기술정보통신부의 재원으로 한국연구재단 국가생명연구자원 선진화사업의 지원을 받아 수행된 연구입니다 (No. 2021M3H9A1081268).
이해상충관계
저자는 이해상충관계가 없음을 선언합니다.
References
-
Avenot HF, Michailides TJ, 2020. Occurrence and extent of boscalid resistance in populations of Alternaria alternata from California pistachio orchards. Plant Dis. 104(???):306-314.
[https://doi.org/10.1094/PDIS-04-19-0699-SR]

- Chung HS, Bae HW, 1979. Ginseng anthracnose in Korea - Factors affecting primary inoculum, growth of the pathogen, disease development and control. Korean J. Pl. Prot. 18(1):35-41.
-
Cortaga CQ, Cordez BWP, Dacones LS, Balendres MAO, Cueva FMD, 2023. Mutations associated with fungicide resistance in Colletotrichum species: A Review. Phytoparasitica 51:569-562.
[https://doi.org/10.1007/s12600-023-01063-0]

-
Deising HB, Reimann S, Pascholati SF, 2008. Mechanisms and significance of fungicide resistance. Braz. J. Microbiol. 39:286-295.
[https://doi.org/10.1590/S1517-83822008000200017]

-
Duba A, Goriewa K, Wachowska U, Wiwart M, 2018. Alternaria alternata (Fr.) Keissl with mutation G143A in the Cyt b gene is the source of a difficult-to-control allergen. Environ. Sci. Pollut. Res. 25:469–478.
[https://doi.org/10.1007/s11356-017-0426-z]

-
Han KD, Alam S, Lee TS, Lee MW, 2004. Ginseng anthracnose caused by Colletotrichum dematium. Plant Pathol. J. 20(3):196-199.
[https://doi.org/10.5423/PPJ.2004.20.3.196]

-
Holger BD, Sven R, Sérgio FP, 2008. Mechanisms and significance of fungicide resistance. Brazilian J. Microbiol. 39:286-295.
[https://doi.org/10.1590/S1517-83822008000200017]

-
Hu S, Zhang S, Xiao W, Liu Y, Yu H, et al., 2023. Diversity and characterization of resistance to pyraclostrobin in Colletotrichum spp. from strawberry. Agronomy
[https://doi.org/10.3390/agronomy13112824]

-
Isa DA, Kim HT, 2022. Cytochrome b gene-based assay for monitoring the resistance of Colletotrichum spp. to pyraclostrobin. Plant Pathol. J. 38(6):616-628.
[https://doi.org/10.5423/PPJ.OA.06.2022.0081]

-
Kim AH, Kim SB, Han KD, Kim HT, 2014. Monitoring for the resistance of strobilurin fungicide against Botrytis cinerea causing gray mold disease. Korean J. Pestic Sci. 18(3):161-167. (in Korean)
[https://doi.org/10.7585/kjps.2014.18.3.161]

- Kim HJ, Cheong SS, Kim DW, Park JS, Ryu J, et al., 2008. Investigation into disease and pest incidence of Panax ginseng in Jeonbuk province. Korean J. Medicinal Crop Sci. 16(1):33-38. (in Korean)
- Kim J, 2006. Detection and molecular analysis of Botrytis cinerea causing ginseng grey mold against carbendazim and diethofencarb. MS Diss., Chungbuk National Univ., Cheongju Korea.
-
Kim J, Lee SW, Min JY, Bae YS, Shin MU, et al., 2007. Development of control system with fungicides against diseases of ginseng plant. Res. Plant Dis. 13(3):164-169.
[https://doi.org/10.5423/RPD.2007.13.3.164]

-
Kim S, Min J, Kim HT, 2019. Occurrence and mechanism of fungicide resistance in Colletotrichum acutatum causing pepper anthracnose against pyraclostrobin. Korean J. Pestic Sci. 23(3):202-211.
[https://doi.org/10.7585/kjps.2019.23.3.202]

- Lee JH, Yu YH, Kim YH, Ohh SH, Park WM, 1990. Morphological characteristics and pathogenicity of Alternaria isolates causing leaf and stem blights and black root rot of Korean ginseng. Korean J. Plant Pathol. 6(1):13-20. (in Korean)
- Lee SW, 2008. Survey and chemical control scheme for aerial disease of ginseng and fungicide resistance of Botrytis cinerea. PhD Diss., Chungbuk National Univ., Cheongju Korea.
-
Ma Z, Felts D, Michailides TJ, 2003a. Resistance to azoxystrobin in Alternaria isolates from pistachio in California. Pestic. Biochem. Physiol. 77:66-74.
[https://doi.org/10.1016/j.pestbp.2003.08.002]

-
Ma Z., Yoshimura M., Michailides T.J., 2003b. Identification and characterization of benzimidazole resistance in Monilinia fructicola from stone fruit orchards in California. Appl. Environ. Microbiol. 69:7145–7152.
[https://doi.org/10.1128/AEM.69.12.7145-7152.2003]

-
Ma J, Michailides TJ, 2005. Advances in understanding molecular mechanisms of fungicide resistance and molecular detection of resistant genotypes in phytopathogenic fungi. Crop Protect. 24:853–863.
[https://doi.org/10.1016/j.cropro.2005.01.011]

-
Odilbekov F, Edin E, Garkava-Gustavsson L, Hovmalm HP, Liljeroth E, 2016. Genetic diversity and occurrence of the F129L substitutions among isolates of Alternaria solani in south-eastern Sweden. Hereditas 153.
[https://doi.org/10.1186/s41065-016-0014-0]

-
Putnama ML, du Toit LJ, 2003. First report of alternaria blight caused by Alternaria panax on ginseng (Panax quinquefolius) in Oregon and Washington, USA. Plant Pathology 52:406.
[https://doi.org/10.1046/j.1365-3059.2003.00828.x]

-
Samuel S, Papayiannis LC, Leroch M, Veloukas T, Hahn M, et al., 2011. Evaluation of the incidence of the G143A mutation and cytb intron presence in the cytochrome bc-1 gene conferring QoI resistance in Botrytis cinerea populations from several hosts. Pest. Manag. Sci. 67:1029–1036.
[https://doi.org/10.1002/ps.2226]

-
Uchida JY, Aragaki M, Yoshimura MA, 1984. Alternaria leaf spots of Brassaia actinophylla, Dizygotheca elegantissima and Tupidanthus calptratus. Plant Dis. 68:447–449.
[https://doi.org/10.1094/PD-68-447]

-
Yin YN, Kim YK, Xiao CL, 2012. Molecular characterization of pyraclostrobin resistance and structural diversity of the cytochrome b gene in Botrytis cinerea from apple. Phytopathology 102(???):315-322.
[https://doi.org/10.1094/PHYTO-08-11-0234]

-
Yin Y, Miao J, Shao W, Liu X, Zhao Y, Ma Z, 2023. Fungicide resistance: Progress in understanding mechanism, monitoring, and management. Phytopathology 113:707-718.
[https://doi.org/10.1094/PHYTO-10-22-0370-KD]

Kyung Sun Myung, ENBIO Co., LTD., Associate research engineer, Research investigation, data analysis, and first draft preparation https://orcid.org/0009-0009-9800-4096
Abdulkareem Abdullahi, Department of Plant Medicine, College of Agriculture, Life and Environment Science, Chungbuk National University, PhD student, https://orcid.org/0000-0002-3841-2356 Development of allele-specific PCR and data analysis
Heung Tae Kim, Department of Plant Medicine, College of Agriculture, Life and Environment Science, Chungbuk National University, Professor, https://orcid.org/0000-0001-7132-0587 Establishment of experimental plan and methodology proposal, writing original paper and editing