유기염소계 살충제 엔도설판이 토양세균 군집에 미치는 영향 평가
초록
유기염소계 살충제 엔도설판은 2012년 국제적으로 사용이 금지되었으나 인도와 중국을 비롯한 국가들에서 여전히 사용되고 있으며 높은 잔류성과 이동성으로 인해 다양한 환경에서 검출되고 있다. 본 연구에서는 엔도설판이 토양세균의 군집구조에 미치는 영향을 16S rRNA 유전자 파이로시퀀싱 기법을 이용하여 분석하였다. 엔도설판을 100 mg/kg의 수준으로 밭토양에 처리했을 때 세균의 Operational taxonomic unit (OTU) 수와 다양성 지수가 감소했다가 서서히 증가하였으며 Proteobacteria와 Verrucomicrobia 문의 점유율은 증가하고 Chloroflexi와 Spirochaetes 문의 점유율은 감소한 것으로 나타나 엔도설판 처리가 토양세균 군집구조를 변화시키는 것을 확인하였다. 엔도설판 처리 시 Sphingomonas와 Burkholderia 속에 속하는 OTU의 점유율이 배양 초기에 높게 나타났으며 Pseudonocardia와 Opitutus 속에 속하는 OTU의 점유율은 배양 후기에 높게 나타났다. 이전 연구에서 Sphingomonas, Burkholderia, Pseudonocardia 속에 속하는 균주들이 엔도설판을 분해하였으므로 이 속에 속하는 OTU들이 토양에서 엔도설판의 분해에 단계적으로 관여했을 것으로 추정된다.
Abstract
Although a global ban on the use of endosulfan, an organochloline insecticide, has taken effect in mid-2012, it has been still used in several countries, including India and China, and detected in diverse environments in the world due to its relative persistence and semi-volatility. In this study, the effect of endosulfan on soil bacterial community was investigated using 16S rRNA gene pyrosequencing method. When endosulfan was applied to an upland soil at a rate of 100 mg/kg soil (ES soil), the number of operational taxonomic units (OTU) and diversity indices for bacteria initially decreased and gradually recovered to the level of the non-treated soil (NT soil) during an eight-week incubation period. At bacterial phylum level, relative abundances of Proteobacteria and Verrucomicrobia were higher while those of Chloroflexi and Spirochaetes were lower in the ES soil than in the NT soil, suggesting that an endosulfan application affects the bacterial community structure in soil. In the ES soil, the relative abundances of the OTUs affiliated to the genera Sphingomonas and Burkholderia increased in the initial period of incubation while those affiliated to the genera Pseudonocardia and Opitutus increased in the late period of incubation. Because the first three genera contain bacterial strains reported to degrade endosulfan, they are expected to be involved in the degradation of endosulfan, probably one after another.
Keywords:
Bacterial community, endosulfan, pyrosequencing, soil키워드:
세균 군집, 엔도설판, 파이로시퀀싱, 토양서 론
유기염소계 살충제인 엔도설판(Endosulfan)은 높은 독성, 생물농축성, 이동성, 난분해성 때문에 2012년 ‘잔류성 유기오염물질(POPs, persistent organic pollutants)에 관한 스톡홀름 협약’에서 국제적으로 생산 및 사용이 금지되었다(http://chm.pops.int). 그러나 인도, 중국 및 여러 개발도상국가에서 여전히 사용되고 있으며 토양, 퇴적층, 민물, 해수, 대기 등 다양한 환경에서 최근까지 검출되고 있다(Weber et al., 2010; Morris et al., 2016).
우리나라에서도 2011년부터 엔도설판의 농약 등록이 취소되었으며 2015년부터는 국내 잔류성유기오염물질에 포함되어 그 관리 체계가 강화되었다. 농약 등록이 취소되기 전까지 엔도설판은 시설재배지, 밭토양, 과수원 토양에서 가장 높은 빈도(~70%)로 검출된 농약이었으며(Park et al., 2013), 최근 연구에 따르면 엔도설판의 대사산물이면서 그 이상의 독성 및 잔류성을 갖는 엔도설판 설페이트(Endosulfan sulfate)가 시설재배지 토양에서 최대 118.6 μg/kg까지 검출되었다(Lim et al., 2016a; 2016b). 또한 농산물에서도 잔류허용기준(0.1 mg/kg 이하)이 초과된 경우가 보고되었으며 최대 4.8 mg/kg까지 검출되었다(Jang, 2015).
엔도설판이 허용기준을 초과하여 검출되는 경우는 많지 않지만 환경과 인간에 대한 만성적 영향을 감안한다면(Brinati et al., 2016; Pato ka et al., 2016; Téllez-Bañuelos et al., 2016) 환경 중에서 엔도설판의 거동을 계속 감시해야 한다.
다양한 생물학적, 비생물학적 요인이 농약 분해에 관여하지만 일반적으로 미생물 특히 세균이 가장 큰 역할을 하는 것으로 생각되고 있으므로(Fenner et al., 2013) 농약 분해에 관여하는 세균 및 그 분해경로를 구명하는 것이 중요하다. 현재까지 농화배양법을 이용하여 엔도설판을 대사하는 다양한 세균 및 곰팡이가 순수 분리되었으나(Kataoka and Takagi, 2013) 이들이 실제 환경에서 기여하는 역할은 검증되지 않았다. 또한 이전 연구에서 배양 및 비배양 방법을 사용하여 엔도설판이 토양 미생물 군집에 미치는 영향을 조사하였으나(DeLorenzo et al., 1999; Gupta et al., 2013; Zhang et al., 2015) 최신 분석기술인 차세대염기서열분석법을 통한 심도있는 조사는 수행되지 않았다.
본 연구에서는 토양에 엔도설판이 첨가되었을 때 토양세균의 군집 변화를 차세대염기서열분석법인 파이로시퀀싱 방법을 이용하여 관찰하였다. 이를 통해 실제 토양환경에서 엔도설판의 분해에 관여하는 미생물과 엔도설판이 토양세균 군집 구조에 미치는 영향을 구명하고자 하였다. 이 연구 결과는 전통적인 농화배양 방법으로 얻은 미생물과 실제 환경 조건에서 나타나는 미생물 사이의 괴리를 줄이고 보다 현실적인 정화 방법을 개발하는데 기여할 수 있을 것으로 생각된다.
재료 및 방법
Microcosm 조성 및 토양 시료 채취
토양 시료는 전북 완주군 이서면 소재 국립농업과학원의 밭 시험포장에서 채취하였다. 채취한 토양은 2 mm의 실험용 체로 걸러 암석 조각을 제거하였으며 성분 분석을 위한 토양은 실험실에서 3일간 풍건한 후 토성과 화학적 성분을 농업기술실용화재단에 의뢰하여 분석하였다. pH는 시료 5 g에 증류수 25 ml를 가하고 1시간 혼합 후 측정하였고 유기물 함량은 Wakely와 Black의 방법(Allison, 1965)으로 측정하였으며 총질소와 유효인산은 각각 Kjeldahl법과 Lancaster법(NIAST, 1988)으로 측정하였다. 치환가능한 칼슘, 마그네슘, 칼륨 이온의 함량은 ICP-AES (GBC Intergra-XMP, Australia)를 이용하여 분석하였다. 토양의 토성은 사질양토로서 모래 76.6%, 침적토 14.4%, 점토 9.0%의 비율로 구성되었으며 화학적 성분은 Table 1과 같다. 엔도설판 처리는 채취한 토양 1 kg을 24×18×8 cm의 사각플라스틱 통에 담고 지오릭스 유제(경농, 엔도설판 35%, 유화제/안정제/용제 65%) 285.7 μL를 증류수 100 mL에 희석하여 토양 1 kg에 골고루 분무하였으며(엔도설판 유효농도 100 mg/kg soil), 대조구에는 동량의 멸균수를 분무하여 실온에 보관하면서 주 1회 멸균된 시약 스푼으로 고르게 혼합하였다. 미생물 분석용 토양시료는 1주일 간격으로 8주 동안 5지점에서 10 g 씩 총 50 g을 채취하여 -70oC의 초저온 냉동고에 보관하면서 DNA 추출에 사용하였다.
토양 DNA 추출
시기별로 채취하여 보관 중인 토양 시료 1 g으로부터 Fast DNA SPIN kit (MP Biochemicals, USA)을 이용하여 제조사의 방법대로 토양 DNA를 추출하였다. 추출한 DNA는 미량분광광도계(ACTgene, USA)를 이용하여 정량하였다.
16S rRNA 유전자 PCR 및 파이로시퀀싱 분석
세균의 16S rRNA 유전자는 각 시료로부터 추출된 DNA를 주형으로 하여 PCR 반응으로 증폭하였다. PCR 반응에 사용한 primer로 정방향 프라이머는 V1-9F (5’-X-AC-GAGTTTGATCMTGGCTCAG-3’), 역방향 프라이머는 V3-541R (5’-X-AC-WTTACCGCGGCTGCTGG-3’)을 사용하였다(Chun et al., 2010). 프라이머 중 X는 7~11개의 뉴클레오타이드로 구성된 바코드로서 시료를 구별하기 위해 사용되었다. PCR 반응액은 총 50 μl로 하였으며 1×PCR buffer(Roche, Germany), 4종의 dNTP 각각 0.2 mM, 정방향 및 역방향 프라이머 0.4 μM, bovine serum albumin 1 mg/ml, Taq DNA polymerase 1.25 units (Roche, Germany), DNA 50 ng으로 조제하였다. PCR은 touch-down 방식으로 3단계로 수행하였다. 초기 94oC에서 5분간 denaturation 반응 후, denaturation (94oC 30초)-annealing (60oC 45초)-extension(72oC 90초)을 10회 반복하였다. 이 때 1회 반복 시마다 annealing 온도를 0.5oC씩 낮추었다. 다음 단계에서 denaturation(94oC 30초)-annealing (55oC 45초)-extension (72oC 90초)을 20회 반복하였다. 증폭된 PCR 산물은 QIAquick Gel Extraction Kit (QIAGEN, Germany)으로 정제한 후 서울대학교 농생명과학공동기기원에 의뢰하여 454 GS FLX Titanium Sequencing System (Roche, Germany)을 이용하여 파이로시퀀싱을 수행하였다.
파이로시퀀싱 결과 분석
파이로시퀀싱 결과는 Mothur (version 1.27.0) (Schloss et al., 2009) 프로그램을 이용하여 분석하였다. 파이로시퀀싱으로 얻은 데이터는 예비 여과과정을 통해 길이가 짧거나 프라이머의 불일치가 있는 시퀀스를 제거한 후 PyroNoise 알고리즘(Quince et al., 2011)을 이용하여 파이로시퀀싱 에러를 수정하고 UCHIME 프로그램(Edgar et al., 2011)을 이용하여 키메라 시퀀스를 제거하였다. 다음으로 RDP database(version 9) (Cole et al., 2009)를 이용하여 진핵생물, 고세균, 클로로플라스트, 미토콘드리아, 계(kingdom) 수준에서 분류가 안된 염기서열들을 제거한 후, 각 시료의 염기서열을 4,512개로 표준화하였다. 표준화된 염기서열을 SINA Aligner(version 1.1) (Pruesse et al., 2012)를 이용하여 정렬한 후 average-neighbor 알고리즘을 이용하여 97% 유사도를 기준으로 operational taxonomic unit (OTU)으로 분류하였다. Good의 coverage와 다양성 지수 계산을 위해서는 Mothur 프로그램을 이용하였다.
결과 및 고찰
세균군집 다양성
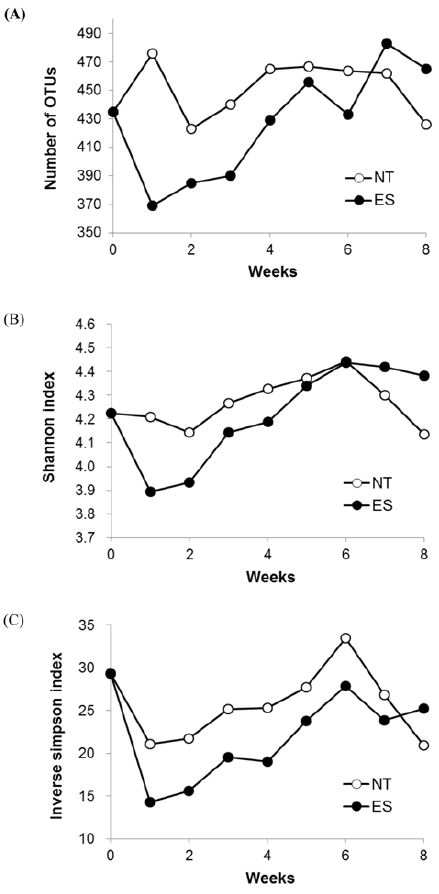
무처리 토양과 엔도설판 처리 토양에서 얻은 16S rRNA 유전자 염기서열 중 낮은 품질의 염기서열을 제거하고 4,512개~6,329개의 염기서열이 남았다. 시료 간 종 풍부도와 다양성 비교를 위해 모든 시료의 염기서열 수를 최소 염기서열 수인 4,512개로 표준화하였다. 이때 Good’s coverage는 0.936~0.953으로 선택한 염기서열 수가 시료의 종 다양성을 나타내기에 적절하였다. Fig. 1에 무처리 토양과 엔도설판 처리 토양에서 배양 기간에 따른 OTU 수 및 다양성 지수의 변화를 나타냈다. 무처리 토양의 경우 8주 동안 OTU 수는 큰 변화가 없었으며(Fig. 1A) 다양성 지수의 경우 6주까지 서서히 증가하다가 감소하는 경향(Fig. 1B, C)을 나타내었다. 반면 엔도설판 처리 토양의 경우 OTU 수 및 다양성 지수가 1주차에 감소했다가 서서히 증가하여 6~7주차에는 무처리 토양 수준에 도달하였다. 이는 엔도설판 처리가 토양 세균의 종 풍부도 및 다양성을 감소시켰음을 의미한다. 농약 살포가 토양 세균의 다양성을 감소시킨다는 연구 결과는 이전에도 보고되었다(Wang et al., 2008; Itoh et al., 2014). 이는 농약이 민감한 세균종의 밀도를 감소시키고 농약을 이용할 수 있거나 내성이 있는 특정 세균종의 밀도를 증가시키기 때문으로 추정된다. 배양 기간에 따른 OTU 수 및 다양성 지수의 증가는 엔도설판 분해로 인해 토양 세균에 대한 약제의 영향이 감소되었기 때문으로 추정된다.
세균 그룹 별 점유율의 변화
실험실 배양 전후 토양시료의 세균군집은 Bacteroidetes, Proteobacteria, Actinobacteria, Chloroflexi 문이 우점하는 것으로 나타났다(Fig. 2). 이 중 Bacteroidetes와 Proteobacteria 문의 점유율은 실험실 배양 직후 급격히 증가했다가 서서히 감소했으며(Fig. 3A, B) Actinobacteria와 Chlorolfexi 문의 점유율은 급격히 감소했다가 서서히 증가하는 양상을 나타냈다(Fig. 3C, D). Bacteroidetes와 Proteobacteria 문에 속하는 세균들은 그람음성균이며 Actinobacteria 문에 속하는 세균들과 Chloroflexi 문의 대부분을 차지했던 Thermosporothrix 속은 그람양성균이다(Yabe et al., 2016). 이전 연구에서 근권 및 비근권 토양에서 우기에는 그람음성균이, 건기에는 그람양성균이 우점하는 현상이 관찰되었는데 이는 그람양성균이 건조한 조건에 대한 내성이 크기 때문으로 추정되었다(Kavamura et al., 2013). 본 연구에서 실험실 배양 시 첨가된 10%의 수분이 그람음성균의 성장을 촉진했으며 배양 기간에 따라 수분이 소실되면서 본래의 군집 구조로 천이가 일어난 것으로 추정된다.
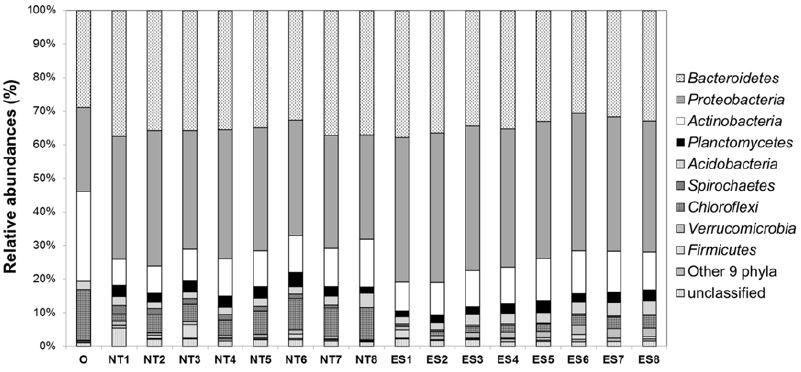
Variation in phylum distribution of bacterial community in non-treated (NT) and endosulfan-treated (ES) soils according to incubation time. The numbers indicate the incubation time (week). The symbol ‘O’ represents the initial soil before incubation.
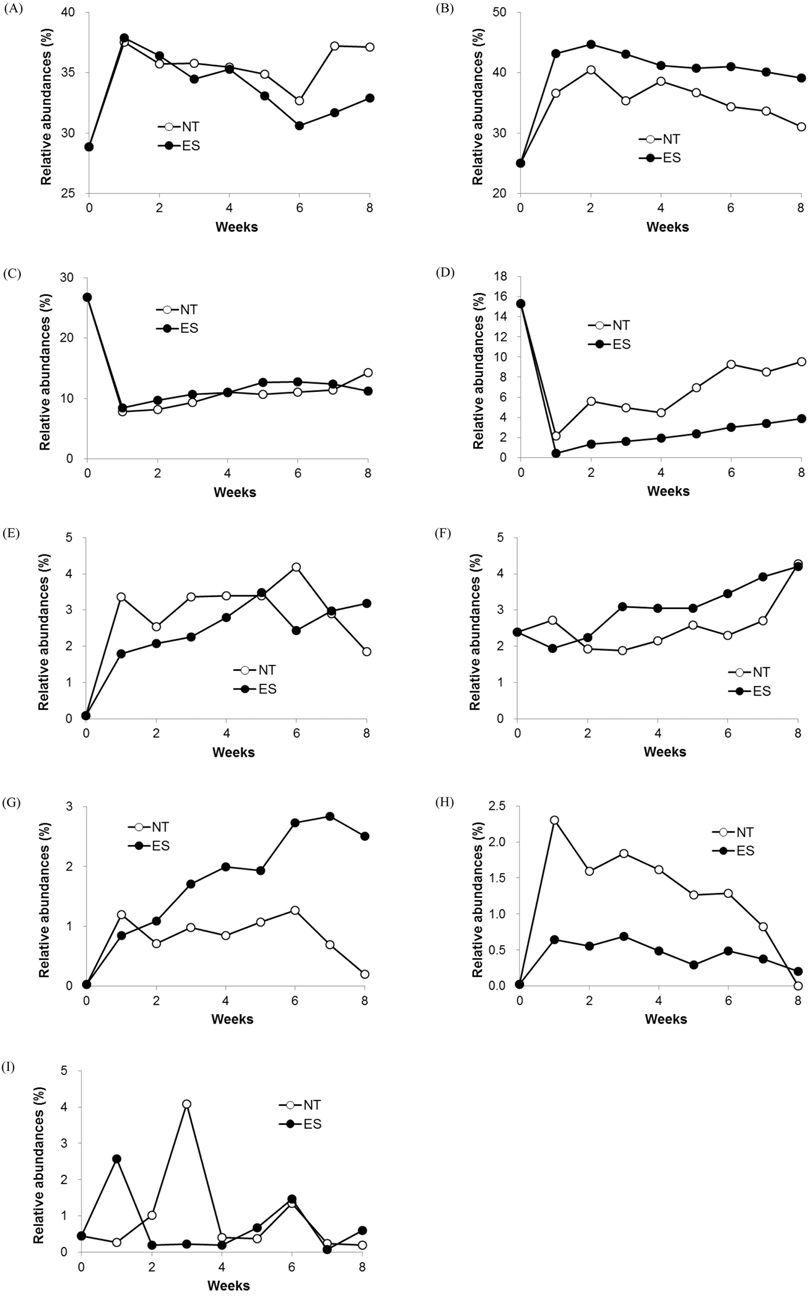
Variations in relative abundances of bacterial phyla in the non-treated (NT) and endosulfan-treated (ES) soils according to the incubation time. (A) Bacteroidetes; (B) Proteobacteria; (C) Actinobacteria; (D) Chloroflexi; (E) Planctomycetes; (F) Acidobacteria; (G) Verrucomicrobia; (H) Spirochaetes; (I) Firmicutes.
Proteobacteria와 Verrucomicrobia 문의 점유율은 엔도설판 처리 시 무처리에 비해 증가하였으며(Fig. 3B, G), Chloroflexi와 Spirochaetes 문의 점유율은 엔도설판 처리 시 무처리에 비해 감소하였다(Fig. 3D, H). 이러한 점유율의 변화는 엔도설판 처리에 대해 전자와 후자의 반응이 다름을 나타낸다. 전자의 경우 엔도설판에 대해 내성이 있거나 이 살충제를 영양분으로 이용했을 가능성이 있으며 후자의 경우 엔도설판 처리가 성장에 불리한 점으로 작용했을 것으로 추정된다.
α-Proteobacteria에 속하는 세균 중 Sphingomonas 속으로 분류된 Otu0050은(Fig. 5) 배양 초기에 무처리 토양에 비해 엔도설판 처리토양에서 그 점유율이 높았다(Fig. 4A). Sphingomonas 속에 속하는 세균들은 인간이 만든 다양한 유기화합물들을 분해할 수 있는 것으로 알려져 있다(Basta et al., 2004; Stolz, 2009). Suh (2004)는 Sphingomonas wittichii RW1은 엔도설판의 두 이성질체를 엔도설판 다이올과 엔도설판 에테르로 전환한다고 보고하였다. 따라서 RW1 균주와 유전적으로 유사한 Otu0050이(96% 일치, Fig. 5) 배양 초기에 엔도설판 분해에 관여했을 것으로 추정된다.
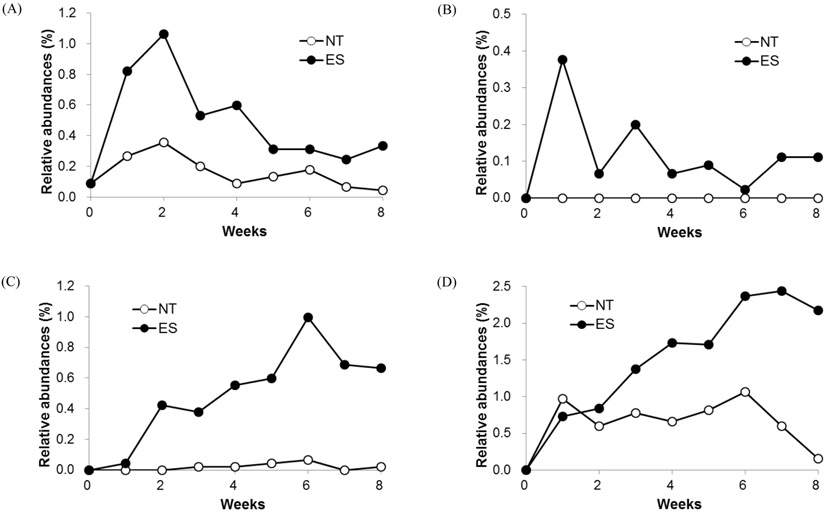
Variations in relative abundances of selected operational taxonomic units (OTUs) in the non-treated (NT) and endosulfantreated (ES) soils according to the incubation time. (A) Otu0050; (B) Otu0139; (C) Otu0057; (D) Otu0019.

Neighbor-joining tree of putative endosulfan-degrading bacteria (in bold), the nearest type strains, and the close relatives reported to degrade endosulfan in the previous studeis based on their 16S rRNA gene sequences.
β-Proteobacteria에 속하는 세균 중 Burkholderia 속으로 분류된 Otu0139는(Fig. 5) 무처리 토양에서는 검출되지 않았으나 엔도설판 처리토양에서는 배양 1주 후 그 점유율이 최대 0.38%까지 증가하였다(Fig. 4B). 이전 연구에서 Burkholderia 속 균주에 의한 엔도설판 분해가 보고되었음(Hussain et al., 2007; Kumar et al., 2008)을 고려할 때 실험 토양에서 검출된 Otu0139가 엔도설판 분해에 관여했을 가능성이 높다.
Actinobacteria 문 중 Pseudonocardia 속으로 분류된 Otu0057(Fig. 5)의 점유율은 무처리 토양에서는 배양 기간 동안 낮게 유지되었으나 엔도설판 처리 토양에서는 배양 후기로 갈수록 증가하는 것으로 나타났다(Fig. 4C). Pseudonocardia 속 균주들은 유기염소화합물을 포함하여 다양한 유기오염물질을 분해하는 것으로 알려져 있으며(Huang and Goodfellow, 2012), 엔도설판 및 엔도설판 설페이트의 분해가 보고된 Pseudonocardia sp. KSF27 (Sakakibara et al., 2011)의 16S rRNA 유전자와 Otu0057이 99% 일치함을 고려할 때 이 OTU가 배양 후기에 엔도설판 분해에 관여했을 것으로 추정된다.
Verrucomicrobia 문에 속하는 세균 중 Opitutus 속으로 분류된 Otu0019는(Fig. 5) 초기에는 거의 검출되지 않았다가 1주 배양 후 증가하였으며 엔도설판 처리 토양에서 배양 후기로 갈수록 증가하는 경향을 나타내었다(Fig. 4D). Opitutus 속에 포함된 종은 현재까지 O. terrae가 유일하며 이 종은 탄수화물을 이용하는 절대혐기성균으로(Chin et al., 2001) 이 균에 의한 유기화학물질의 분해는 알려진 바가 없다. 만약 이 OTU가 엔도설판 분해에 관여한다면 이 문에 속하는 세균에 의한 유기합성 화학물질 분해의 최초 보고가 될 것이다.
본 연구에서는 16S rRNA 유전자 파이로시퀀싱 기법을 이용하여 살충제 엔도설판이 토양세균 군집구조에 미치는 영향을 분석하였다. 엔도설판을 유효농도 100 mg/kg soil로 토양에 살포하였을 때 세균의 종 풍부도와 다양성은 감소했으며 Proteobacteria 문과 Verrucomicrobia 문의 점유율은 무처리 토양에 비해 높아지고 Chloroflexi 문과 Spirochaetes 문의 점유율은 낮아졌다. 이는 약제 처리가 토양세균 군집구조를 변화시켰음을 나타내는 것으로 이러한 변화가 토양 기능에 어떤 영향을 미쳤는지 향후 연구를 통해 밝혀져야 할 것이다.
Sphingomonas 속 및 Burkholderia 속 세균들의 점유율이 엔도설판 처리 토양에서 무처리에 비해 배양 초기에 높게 나타나 이 속의 세균들이 엔도설판의 초기 변환에 관여했을 것으로 추정되며 Pseudonocardia와 Opitutus 속 세균은 배양 후기에 높게 나타나 이 속의 세균들이 이전 단계에서 생산된 엔도설판 대사산물을 이용했을 것으로 추정된다. 이 가설을 뒷받침하기 위해 향후 토양에 잔존하는 엔도설판 및 그 대사산물에 대한 분석이 필요할 것으로 판단된다.
또한 본 연구에서 추정된 엔도설판 분해세균을 순수분리 함으로써 이를 엔도설판 오염 토양 및 농산물의 정화에 이용할 수 있을 것으로 기대한다.
Acknowledgments
본 논문은 농촌진흥청 국립농업과학원 농업과학기술 연구개발사업(과제번호: PJ01094901)의 지원으로 수행된 결과이며 연구비 지원에 감사 드립니다.
References
- Allison, L. E., (1965), Organic Carbon, p1367-1376, In C.A. B (ed.), Methods of Soil Analysis. Part II, Am. Soc. Agron. Inc. Publ., Madison, Wisconsin, USA.
-
Basta, T., A. Keck, J. Klein, and A. Stolz, (2004), Detection and characterization of conjugative degradative plasmids in xenobiotic-degrading Sphingomonas strains, J. Bacteriol, 186, p3862-3872.
[https://doi.org/10.1128/jb.186.12.3862-3872.2004]

-
Brinati, A., J. M. Oliveira, V. S. Oliveira, M. S. Barros, B. M. Carvalho, L. S. Oliveira, M. E. L. Queiroz, S. L. P. Matta, and M. B. Freitas, (2016), Low, chronic exposure to endosulfan induces bioaccumulation and decreased carcass total fatty acids in neotropical fruit bats, B. Environ. Contam. Tox, 97, p626-631.
[https://doi.org/10.1007/s00128-016-1910-8]

-
Chin, K. J., W. Liesack, and P. H. Janssen, (2001), Opitutus terrae gen. nov., sp. nov., to accommodate novel strains of the division Verrucomicrobia isolated from rice paddy soil, Int. J. Syst. Evol. Microbiol, 51, p1965-1968.
[https://doi.org/10.1099/00207713-51-6-1965]

-
Chun, J., K. Kim, J. H. Lee, and Y. Choi, (2010), The analysis of oral microbial communities of wild-type and toll-like receptor 2-deficient mice using a 454 GS FLX Titanium pyrosequencer, BMC Microbiol, 10, p101.
[https://doi.org/10.1186/1471-2180-10-101]

-
Cole, J. R., Q. Wang, E. Cardenas, J. Fish, B. Chai, R. J. Farris, A. S. Kulam-Syed-Mohideen, D. M. McGarrell, T. Marsh, G. M. Garrity, et al , (2009), The Ribosomal Database Project: improved alignments and new tools for rRNA analysis, Nucleic Acids Res, 37, pD141-D145.
[https://doi.org/10.1093/nar/gkn879]

-
DeLorenzo, M. E., G. I. Scott, and P. E. Ross, (1999), Effects of the agricultural pesticides atrazine, deethylatrazine, endosulfan, and chlorpyrifos on an estuarine microbial food web, Environ. Toxicol. Chem, 18, p2824-2835.
[https://doi.org/10.1897/1551-5028(1999)018<2824:eotapa>2.3.co;2]

-
Edgar, R. C., B. J. Haas, J. C. Clemente, C. Quince, and R. Knight, (2011), UCHIME improves sensitivity and speed of chimera detection, Bioinformatics, 27, p2194-2200.
[https://doi.org/10.1093/bioinformatics/btr381]

-
Fenner, K., S. Canonica, L. P. Wackett, and M. Elsner, (2013), Evaluating pesticide degradation in the environment: blind spots and emerging opportunities, Science, 341, p752-758.
[https://doi.org/10.1126/science.1236281]

-
Gupta, S., R. Gupta, and S. Sharma, (2013), Impact of chemicaland bio-pesticides on bacterial diversity in rhizosphere of Vigna radiata, Ecotoxicology, 22, p1479-1489.
[https://doi.org/10.1007/s10646-013-1134-1]

- Huang, Y., and M. Goodfellow, (2012), Genus I. Pseudonocardia, In M. Goodfellow (ed.), Bergey's manual of systematic bacteriology, Phylum XXVI. Actinobacteria phyl. nov., Springer, New York.
- Hussain, S., M. Arshad, M. Saleem, and A. Khalid, (2007), Biodegradation of alpha- and beta-endosulfan by soil bacteria, Biodegradation, 18, p731-740.
-
Itoh, H., R. Navarro, K. Takeshita, K. Tago, M. Hayatsu, T. Hori, and Y. Kikuchi, (2014), Bacterial population succession and adaptation affected by insecticide application and soil spraying history, Front. Microbiol, 5, p457.
[https://doi.org/10.3389/fmicb.2014.00457]

- Jang, S. K., (2015), Spring greens currently on the market in Gwangju city are generally safe from pesticides, (http://www.gwangnam.co.kr/read.php3?aid=1427881270207794006).
-
Kataoka, R., and K. Takagi, (2013), Biodegradability and biodegradation pathways of endosulfan and endosulfan sulfate, Appl. Microbiol. Biotechnol, 97, p3285-3292.
[https://doi.org/10.1007/s00253-013-4774-4]

- Kavamura, N. V., R. G. Taketani, M. D. Lançoni, F. D. Andreote, R. Mendes, and I. Soares de Melo, (2013), Water regime influences bulk soil and rhizosphere of Cereus jamacaru bacterial communities in the Brazilian Caatinga biome, PLOS ONE, 8, pe73606.
-
Kumar, M., C. V. Lakshmi, and S. Khanna, (2008), Biodegradation and bioremediation of endosulfan contaminated soil, Bioresour Technol, 99, p3116-3122.
[https://doi.org/10.1016/j.biortech.2007.05.057]

- Lim, S. J., Y. T. Oh, Y. S. Jo, J. H. Ro, G. H. Choi, J. Y. Yang, and B. J. Park, (2016a), Persistent organic pollutants (POPs) residues in greenhouse soil and strawberry organochlorine pesticides, Korean J. Environ. Agr, 35, p6-14.
- Lim, S. J., Y. T. Oh, J. H. Ro, J. Y. Yang, G. H. Choi, S. H. Ryu, B. C. Moon, and B. J. Park, (2016b), Investigation of resiudal organochlorine pesticides in green Perilla (Perilla frutescens var. japonica Hara) greenhouse soil and its leaves, Korean J. Pestic. Sci, 20, p221-227.
-
Morris, A. D., D. C. G. Muir, K. R. Solomon, R. J. Letcher, M. A. McKinney, A. T. Fisk, B. C. McMeans, G. T. Tomy, C. Teixeira, X. Wang, et al , (2016), Current-use pesticides in seawater and their bioaccumulation in polar bear-ringed seal food chains of the Canadian Arctic, Environ. Toxicol. Chem, 35, p1695-1707.
[https://doi.org/10.1002/etc.3427]

- NIAST, (1988), Methods of soil chemical analysis. National Institute of Agricultural Science and Technology (NIAST), Rural Development Administratio (RDA), Suwon, Korea.
-
Park, B. J., B. M. Lee, C. S. Kim, K. H. Park, J. H. Kim, H. Kwon, S. W. Park, G. H. Choi, and S. J. Lim, (2013), Longterm monitoring of pesticide residues in arable soils in Korea, Korean J. Pestic. Sci, 17, p283-292.
[https://doi.org/10.7585/kjps.2013.17.4.283]

- Patočka, J., Q. Wu, T. C. C. França, T. C. Ramalho, R. Pita, and K. Kuča, (2016), Clinical aspects of the poisoning by the pesticide endosulfan, Química Nova, 39, p987-994.
-
Pruesse, E., J. Peplies, and F. O. Glöckner, (2012), SINA: accurate high throughput multiple sequence alignment of ribosomal RNA genes, Bioinformatics, 28, p1823-1829.
[https://doi.org/10.1093/bioinformatics/bts252]

-
Quince, C., A. Lanzen, R. Davenport, and P. Turnbaugh, (2011), Removing noise from pyrosequenced amplicons, BMC Bioinformatics, 12, p38.
[https://doi.org/10.1186/1471-2105-12-38]

-
Sakakibara, F., K. Takagi, R. Kataoka, H. Kiyota, Y. Sato, and S. Okada, (2011), Isolation and identification of dieldrindegrading Pseudonocardia sp. strain KSF27 using a soilcharcoal perfusion method with aldrin trans-diol as a structural analog of dieldrin, Biochem Biophys Res Commun, 411, p76-81.
[https://doi.org/10.1016/j.bbrc.2011.06.096]

-
Schloss, P. D., S. L. Westcott, T. Ryabin, J. R. Hall, M. Hartmann, E. B. Hollister, R. A. Lesniewski, B. B. Oakley, D. H. Parks, C. J. Robinson, et al , (2009), Introducing mothur: open-source, platform-independent, communitysupported software for describing and comparing microbial communities, Appl. Environ. Microbiol, 75, p7537-7541.
[https://doi.org/10.1128/aem.01541-09]

-
Stolz, A., (2009), Molecular characteristics of xenobiotic-degrading sphingomonads, Appl Microbiol Biotechnol, 81, p793-811.
[https://doi.org/10.1007/s00253-008-1752-3]

- Suh, Y. D., (2004), Biodegadation of the endosulfan by Sphingomonas wittichii RW1, J. Korea Soc. Environ. Admin, 10, p287-294.
- Téllez-Bañuelos, M. C., J. Haramati, K. Franco-Topete, J. Peregrina-Sandoval, R. Franco-Topete, and G. P. Zaitseva, (2016), Chronic exposure to endosulfan induces inflammation in murine colon via β-catenin expression and IL-6 production, J. Immunotoxicol, 13, p842-849.
-
Wang, M.-C., Y.-H. Liu, Q. Wang, M. Gong, X.-M. Hua, Y.-J. Pang, S. Hu, and Y.-H. Yang, (2008), Impacts of methamidophos on the biochemical, catabolic, and genetic characteristics of soil microbial communities, Soil Biol. Biochem, 40, p778-788.
[https://doi.org/10.1016/j.soilbio.2007.10.012]

-
Weber, J., C. J. Halsall, D. Muir, C. Teixeira, J. Small, K. Solomon, M. Hermanson, H. Hung, and T. Bidleman, (2010), Endosulfan, a global pesticide: A review of its fate in the environment and occurrence in the Arctic, Sci. Total Environ, 408, p2966-2984.
[https://doi.org/10.1016/j.scitotenv.2009.10.077]

-
Yabe, S., Y. Sakai, and A. Yokota, (2016), Thermosporothrix narukonensis sp. nov., belonging to the class Ktedonobacteria, isolated from fallen leaves on geothermal soil, and emended description of the genus Thermosporothrix, Int. J. Syst. Evol. Microbiol, 66, p2152-2157.
[https://doi.org/10.1099/ijsem.0.001004]

-
Zhang, J., J. Qin, C. Zhao, C. Liu, H. Xie, and S. Liang, (2015), Response of bacteria and fungi in soil microcosm under the presence of pesticide endosulfan, Water Air Soil Poll, 226, p109.
[https://doi.org/10.1007/s11270-015-2309-6]